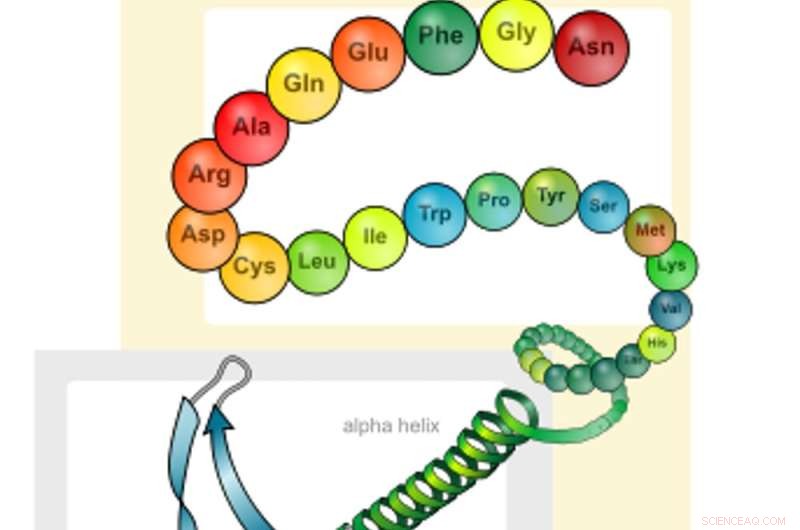
Aminosyrakedjor vikas till proteiner. Kredit:LadyofHats/Wikimedia Commons
Alla levande organismer använder proteiner, som omfattar ett stort antal komplexa molekyler. De utför ett brett utbud av funktioner, från att låta växter använda solenergi för syreproduktion till att hjälpa ditt immunsystem att bekämpa patogener till att låta dina muskler utföra fysiskt arbete. Många läkemedel är också baserade på proteiner.
För många områden inom biomedicinsk forskning och läkemedelsutveckling finns det dock inga naturliga proteiner som kan fungera som lämpliga utgångspunkter för att bygga nya proteiner. Forskare som designade nya läkemedel för att förhindra covid-19-infektion, eller utvecklade proteiner som kan slå på eller stänga av gener eller förvandla celler till datorer, var tvungna att skapa nya proteiner från grunden.
Denna process av de novo proteindesign kan vara svår att få rätt. Proteiningenjörer som jag har försökt hitta sätt att mer effektivt och exakt designa nya proteiner med de egenskaper vi behöver.
Lyckligtvis kan en form av artificiell intelligens som kallas djupinlärning vara ett elegant sätt att skapa proteiner som inte fanns tidigare – hallucination.
Designa proteiner från grunden
Proteiner består av hundratals till tusentals mindre byggstenar som kallas aminosyror. Dessa aminosyror är anslutna till varandra i långa kedjor som viks ihop för att bilda ett protein. Ordningen i vilken dessa aminosyror är kopplade till varandra bestämmer varje proteins unika struktur och funktion.
Den största utmaningen som proteiningenjörer står inför när de designar nya proteiner är att ta fram en proteinstruktur som kommer att utföra en önskad funktion. För att komma runt detta problem skapar forskare vanligtvis designmallar baserade på naturligt förekommande proteiner med liknande funktion. Dessa mallar har instruktioner om hur man skapar de unika vecken av varje särskilt protein. Men eftersom en mall måste skapas för varje enskild veck är denna strategi tidskrävande, arbetskrävande och begränsad av vilka proteiner som finns tillgängliga i naturen.
Under de senaste åren har olika forskargrupper, inklusive labbet jag arbetar i, utvecklat ett antal dedikerade djupa neurala nätverk – datorprogram som använder flera bearbetningslager för att "lära sig" av indata för att göra förutsägelser om en önskad utdata.
När den önskade produktionen är ett nytt protein, sätts miljontals parametrar som beskriver olika aspekter av ett protein in i nätverket. Vad som förutspås är en slumpmässigt vald sekvens av aminosyror som är mappad till den mest sannolika 3D-struktur som sekvensen skulle ta.
Nätverksförutsägelser för en slumpmässig aminosyrasekvens är suddiga, vilket innebär att den slutliga strukturen av proteinet inte är särskilt tydlig, medan både naturligt förekommande proteiner och proteiner byggda från grunden producerar mycket mer väldefinierade proteinstrukturer.
Hallucinerande nya proteiner
Dessa observationer antyder ett sätt att nya proteiner kan genereras från grunden – genom att justera slumpmässiga indata till nätverket tills förutsägelser ger en väldefinierad struktur.
Proteingenereringsmetoden som mina kollegor och jag utvecklade liknar konceptuellt datorseendemetoder som Googles DeepDream, som hittar och förstärker mönster i bilder.
Dessa metoder fungerar genom att ta nätverk som är tränade att känna igen mänskliga ansikten eller andra mönster i bilder, som formen på ett djur eller ett föremål, och vända dem så att de lär sig att känna igen dessa mönster där de inte finns. I DeepDream till exempel får nätverket godtyckliga ingångsbilder som justeras tills nätverket kan känna igen ett ansikte eller någon annan form i bilden. Även om den slutliga bilden inte ser mycket ut som ett ansikte för en person som tittar på den, skulle den göra det för det neurala nätverket.
Produkterna av denna teknik kallas ofta för hallucinationer, och det här är vad vi också kallar våra designade proteiner.
Vår metod börjar med att skicka en slumpmässig aminosyrasekvens genom ett djupt neuralt nätverk. De resulterande förutsägelserna är initialt suddiga, med oklara strukturer, som förväntat för slumpmässiga sekvenser. Därefter introducerar vi en mutation som ändrar en aminosyra i kedjan till en annan och skickar denna nya sekvens genom nätverket igen. Om denna förändring ger proteinet en mer definierad struktur, så behåller vi aminosyran och vi introducerar en annan mutation i sekvensen.
För varje upprepning av denna process kommer proteinerna närmare och närmare den verkliga form de skulle ta om de producerades i naturen. Det krävs tusentals repetitioner för att skapa ett helt nytt protein.
Genom att använda denna process genererade vi 2 000 nya proteinsekvenser som förutspåddes att vikas till väldefinierade strukturer. Av dessa valde vi ut över 100 som var de mest distinkta i form för att fysiskt återskapa i labbet. Slutligen valde vi ut tre av toppkandidaterna för detaljerad analys och bekräftade att de var nära matchningar med de former som våra hallucinerade modeller förutspådde.
Varför hallucinera nya proteiner?
Vår hallucinationsmetod förenklar avsevärt proteindesignpipelinen. Genom att eliminera behovet av mallar kan forskare direkt fokusera på att skapa ett protein baserat på önskade funktioner och låta nätverket ta hand om strukturen för dem.
Vårt arbete öppnar flera vägar för forskare att utforska. Vårt labb undersöker för närvarande hur man bäst kan använda denna hallucinationsmetod för att generera ännu mer specificitet i funktionen hos designade proteiner. Vårt tillvägagångssätt kan också enkelt utvidgas till att designa nya proteiner med andra nyligen utvecklade djupa neurala nätverk.
De potentiella tillämpningarna av de novo-proteiner är enorma. Med djupa neurala nätverk kommer forskare att kunna skapa ännu fler proteiner som kan bryta ner plast för att minska miljöföroreningar, identifiera och svara på ohälsosamma celler och förbättra vacciner mot befintliga och nya patogener – bara för att nämna några.