Genomredigering gör intåg i biomedicinsk forskning och medicin. Genom att använda verktyg för modellering av biomolekyler accelererar ett japanskt forskarlag takten och sänker kostnaderna för zinkfingernukleasteknologi (ZFN), ett primärt genredigeringsverktyg.
I en studie publicerad i Advanced Science , forskare från Hiroshima University och Japanese National Institute of Advanced Industrial Science and Technology visar hur maskininlärningsdrivna modulära monteringssystem kan förbättra genredigering.
"Genomredigering är ett lovande verktyg för behandling av genetiska störningar inom ett antal olika områden", säger Shota Katayama, docent vid Genome Editing Innovation Center vid Hiroshima University. "Genom att förbättra effektiviteten hos genredigeringsteknologier kan vi uppnå större precision i modifieringar av den genetiska informationen i levande celler."
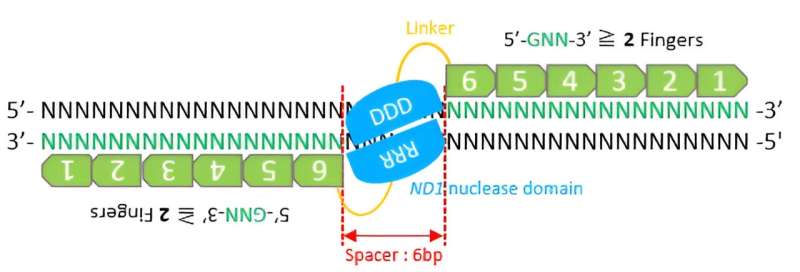
Vid sidan av CRISPR/Cas9 och TALEN är zinkfingernukleas ett viktigt verktyg inom genomredigering. Konstruerade för att bryta vissa bindningar inom polynukleotidkedjan av en DNA-molekyl, är dessa chimära proteiner uppbyggda av två domäner sammansmälta:DNA-bindande och DNA-klyvningsdomäner. Den zinkfingerproteinbindande domänen (ZF) känner igen den riktade DNA-sekvensen inom hela genomet, medan klyvningsdomänen involverar ett speciellt DNA-skärande enzym som kallas ND1-endonukleaser.
ZFN har några fördelar jämfört med CRISPR/Cas9 och TALEN:för det första, till skillnad från för CRISPR-Cas9, har patenten för ZFN redan löpt ut, vilket utesluter höga patentersättningar för industriella tillämpningar. För det andra är ZFN:er mindre, vilket gör att ZFN-kodande DNA enkelt kan paketeras i en viral vektor med begränsat lastutrymme för in vivo och kliniska tillämpningar.
För att klippa DNA måste två ZFN bindas. Därför måste de designas i par för att vara funktionella på alla nya platser. Det har dock visat sig vara utmanande att konstruera funktionella ZFN och förbättra deras genomredigeringseffektivitet.
"Vi har gjort enorma framsteg när det gäller metoder för att härleda zinkfingeruppsättningar för nya genomiska mål, men det finns fortfarande utrymme att förbättra våra metoder för design och urval," sa Katayama.
Selektionsbaserade metoder kan användas för att konstruera sammansatta ZF-proteiner, men dessa metoder är arbetsintensiva och tidskrävande. Ett alternativt tillvägagångssätt för att konstruera sammansatta ZF-proteiner är sammansättningen av ZF-moduler med användning av standardtekniker för molekylärbiologi. Denna metod ger forskare en mycket enklare metod att konstruera sammansatta ZF-proteiner.
Modulärt sammansatta ZFN-enheter har dock ett litet antal funktionella ZFN-par med en felfrekvens på 94 % för de testade ZFN-paren.
I sin studie syftade forskarna från Hiroshima University och Japanese National Institute of Advanced Industrial Science and Technology på att skapa ett mer effektivt, lättkonstruerat zinkfingernukleas för genredigering med hjälp av allmänt tillgängliga resurser i ett modulärt sammansättningssystem.
En viktig faktor vid utformningen av ZFN är antalet zinkfingrar som krävs för effektiv och specifik klyvning. Teamet antog att den modulära sammansättningen av ZF-modulerna skulle vara användbar för att konstruera ZFN med fem eller sex fingrar.
I sin publikation presenterade forskargruppen en metod för att öka effektiviteten i konstruktionen av funktionella ZFN och förbättringen av deras genomredigeringseffektivitet med hjälp av tre biomolekylmodelleringsverktyg:AlphaFold, Coot och Rosetta.
Av de 10 ZFN som testades fick forskarna två funktionella par. Dessutom ökade konstruktionen av ZFNs med hjälp av AlphaFold, Coot och Rosetta effektiviteten av genomredigering med 5 %, vilket visar effektiviteten hos konstruktion av ZFN baserade på strukturell modellering.
Mer information: Shota Katayama et al, Engineering of Zinc Finger Nucleases Through Structural Modeling Improves Genome Editing Efficiency in Cells, Advanced Science (2024). DOI:10.1002/advs.202310255
Journalinformation: Avancerad vetenskap
Tillhandahålls av Hiroshima University