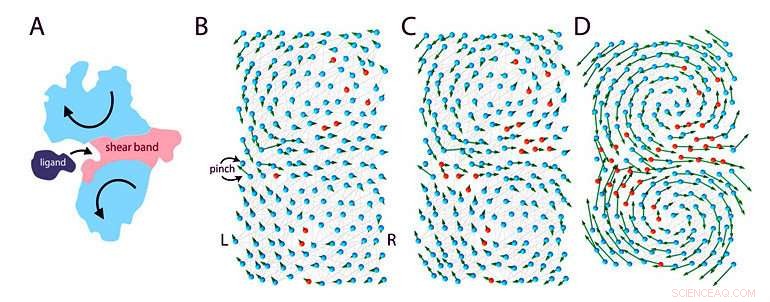
Figur 1:Elastisk modell av en proteinbindning till en ligand. (A) När ett protein binder till en ligand, den genomgår storskalig rörelse (pilar) som är signaturer för böjande funktionella proteiner. Detta är endast möjligt tack vare förekomsten av vissa "diskett" -regioner (rosa "skjuvband") över proteinet som separerar de stela (blå) regionerna i proteinet i två domäner. (B)-(D) Teamet modellerade ett 200 aminosyraprotein under olika utvecklingsstadier:passerar från ett icke-funktionellt (B) till ett funktionellt (D) tillstånd. Proteinet är utformat som ett elastiskt fjädernätverk med två sorters aminosyror, modellerade som pärlor:rosa aminosyror är flexibla och blå aminosyror är styva. Forskarna efterliknar evolutionen genom att ändra en slumpmässig aminosyra vid tidpunkten (mutation) från rosa till blått. Initialt, proteinet är mestadels styvt och icke-funktionellt. Under evolutionen flexibla aminosyror tillsätts, några användbara, vissa inte. Över tid, en "diskett" region bildas i mitten av molekylen vilket gör proteinet mer flexibelt att böja och binda till liganden. Modellen uppskattade att en effektiv lösning uppnås efter tusen mutationer. Kredit:Institute for Basic Science
Ett internationellt team har utvecklat en modell som simulerar proteinutveckling. Börjar från stel, opunktionella proteiner, datormodellen visar hur utvecklande proteinkomponenter kan samverka för att ge upphov till dynamiska och effektiva molekylära maskiner. Flexibilitet gör att proteiner kan ändra sin 3D -konformation för att binda andra molekyler:denna egenskap är avgörande för deras funktion. Prof. Tsvi Tlusty och Dr Sandipan Dutta vid Center for Soft and Living Matter, inom Institute for Basic Science (IBS, Sydkorea), i samarbete med professor Albert Libchaber från Rockefeller University och professor Jean-Pierre Eckmann från University of Geneva har efterliknat genutveckling för att få proteiner som kan böja och binda till andra molekyler. Att förstå detta förhållande är en av de mest eftertraktade aspekterna av proteinbiologi; det kan hjälpa till att förklara läkemedelsverkan av läkemedel som binder till deras mål.
Evolutionen har format den levande världen vi ser runt oss i miljarder år. Miljontals proteiner arbetar harmoniskt för att hålla igång dessa livsprocesser. De är ansvariga för en väl fungerande organism:de känner igen andra molekyler (ligander), binda till dem och konvertera dem. Andra har transportfunktion, ge struktur, och stöd till cellerna. Gener lagrar informationen om produktion och design av dessa molekylära maskiner. Dock, trots årtionden av forskning, att utarbeta "kartan" som drar vägen från gener till proteinfunktion är inte trivialt.
Enligt en ny hypotes, proteinfunktionen är beroende av "flexibla leder". Den här studien, publicerad i Förfaranden från National Academy of Sciences ( PNAS ), undersöker kopplingen mellan funktion och flexibilitet genom att modellera proteiner som elastiska nätverk. I denna modell, proteiner är gjorda av flexibla (polära) och styva (hydrofoba) aminosyror som är förbundna med molekylära "fjädrar". Om vissa delar av proteinet är tillräckligt flexibla, de bildar en "diskett" kanal, och hela molekylmaskinen kan böja sig som ett gångjärn. Denna rörelse låter dem binda effektivt till andra molekyler. Bindningen mellan en ligand och ett styvt eller flexibelt protein kan ses som en boll som landar på en sten eller en mjuk kudde. Bollen kommer troligen att studsa bort efter att ha träffat berget, men det är mer troligt att kudden accepterar det. Därför, det flexibla proteinet är ett bättre bindemedel.
I denna modell, gener lagrar detaljerna i proteindesignen på ett binärt sätt:flexibla aminosyror lagras som nollor och styva aminosyror som enor. Som ett resultat, hela proteinstrukturen kan förenklas som en kod, som 11110001 ... 111, liknande det digitala minnet på en dator. Dock, inte alla koder ger upphov till funktionella proteiner, till exempel en kod med enbart:111111… 1111, skulle ge upphov till ett helt stelt protein, oförmögen att röra sig, och icke -funktionell. Bland alla möjliga koder, bara vissa producerar ett funktionellt protein med en "diskett" region i mitten som kan välkomna liganden.
Modellen efterliknar evolutionen genom att ändra en slumpmässig aminosyra åt gången. Under evolutionen nollorna och enorna i genen bläddras slumpmässigt genom en process som kallas mutation. De flesta mutationer ger ingen skillnad, eller leda till icke-funktionella proteiner, men vissa sällsynta mutationer kan ge upphov till ett mer effektivt protein. Väsentligen, både funktionella och icke-funktionella proteiner produceras under evolutionen, men enligt Darwins teori om "överlevnad för de starkaste", bara de funktionella proteinerna bevaras och de icke-funktionella proteinerna dör så småningom ut.
Hur ser en "funktionell" kod ut? Svaret är inte enkelt. Faktiskt, antalet koder för ett funktionellt protein, även ett enkelt protein, är enorm, större än universums storlek. Dock, använder tekniker för dataanalys, det är möjligt att söka efter dolda mönster i alla funktionella koder för att leta efter några förenande egenskaper. Till exempel, "diskett" -kanalen i proteinet har intressanta och säregna egenskaper, och en mutation i ena änden av kanalen har långdistanseffekter som starkt kan påverka underhållet av mutationer av andra avlägsna aminosyror.
"I framtiden, vi planerar att utforska hur vi kan tillämpa denna studie på riktiga proteiner, som kinaser, "sade gruppledaren Tsvi Tlusty, en korrespondent i studien. "Dessutom, studien öppnar vägar för att undersöka utvecklingen av andra proteinfunktioner, som molekylärt igenkänning. Med enorma databaser, som har utvecklats genom år av forskning, kan förmodligen avslöja några underliggande fenomen om utvecklingen av proteiner. "