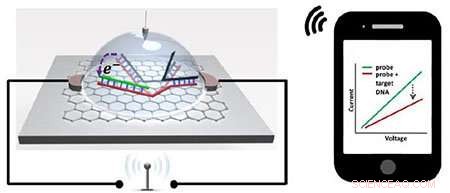
Illustration av grafenbaserat SNP-detekteringschip som trådlöst sänder signal till en smartphone. Upphovsman:Lal et al.
Ett team som leds av University of California San Diego har utvecklat ett chip som kan upptäcka en typ av genetisk mutation som kallas en enda nukleotidpolymorfism (SNP) och skicka resultaten i realtid till en smartphone, dator, eller annan elektronisk enhet. Chippet är minst 1, 000 gånger känsligare för att upptäcka en SNP än nuvarande teknik.
Förskottet, publicerad 9 juli i Avancerade material , kan leda till billigare, snabbare och bärbar biosensorer för tidig upptäckt av genetiska markörer för sjukdomar som cancer.
En SNP är förändringen i en enda nukleotidbas (A, C, G eller T) i DNA -sekvensen. Det är den vanligaste typen av genetisk mutation. Medan de flesta SNP inte har någon märkbar effekt på hälsan, vissa är förknippade med ökad risk att utveckla patologiska tillstånd som cancer, diabetes, hjärtsjukdom, neurodegenerativa störningar, autoimmuna och inflammatoriska sjukdomar.
Traditionella SNP -detektionsmetoder har flera begränsningar:de har relativt dålig känslighet och specificitet; de kräver förstärkning för att få flera kopior för detektion; de kräver användning av skrymmande instrument; och de kan inte fungera trådlöst.
Den nya DNA-biosensorn utvecklad av UC San Diego-ledda teamet är ett trådlöst chip som är mindre än en nagel och kan upptäcka en SNP som finns i picomolära koncentrationer i lösning.
"Miniatyriserad chipbaserad elektrisk detektion av DNA kan möjliggöra detektering av specifika DNA-sekvenser och polymorfismer på fältet och på begäran för tidig diagnos eller prognos av pågående hälsokriser, inklusive virus- och bakterieinfektionsbaserade epidemier, "sa Ratnesh Lal, professor i bioingenjör, maskinteknik och materialvetenskap vid UC San Diego Jacobs School of Engineering.
Chippet fångar i huvudsak en DNA -sträng som innehåller en specifik SNP -mutation och producerar sedan en elektrisk signal som skickas trådlöst till en mobil enhet. Den består av en grafenfälteffekttransistor med en specialkonstruerad bit dubbelsträngad DNA fäst vid ytan. Denna bit av DNA är böjd nära mitten och formad som en pincett. Ena sidan av dessa så kallade "DNA-pincetter" -koder för en specifik SNP. När en DNA -sträng med den SNP närmar sig, det binder till den sidan av DNA-pincetten, öppna dem och skapa en förändring i elektrisk ström som detekteras av grafenfälteffekttransistorn.
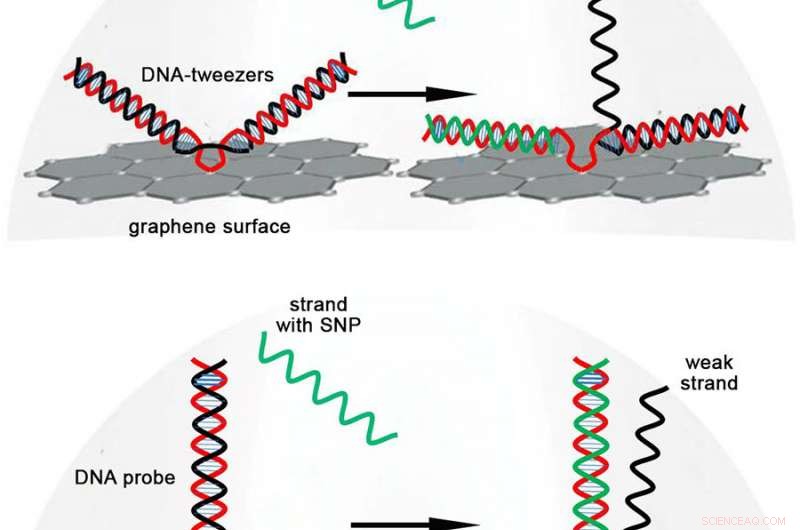
Tecknad film som jämför DNA-strängförskjutning med hjälp av DNA-pincetten (överst) kontra DNA-sonden inbyggd i föregående chip (botten). Upphovsman:Deependra Kumar Ban
Projektet leds av Lal och involverar team vid Institute of Engineering in Medicine vid UC San Diego, Kinesiska vetenskapsakademien i Kina, University of Pennsylvania, Max Planck Institute for Biophysical Chemistry i Tyskland, och Inre Mongoliet Agricultural University i Kina.
Förskjutning av DNA -sträng
Det som driver denna teknik är en molekylär process som kallas förskjutning av DNA -strängar - när en DNA -helix utbyter en av dess strängar mot en ny komplementär sträng. I detta fall, DNA-pincetten byter en sina strängar mot en med en viss SNP.
Detta är möjligt på grund av det speciella sättet på vilket DNA-pincetten konstrueras. En av strängarna är en "normal" sträng som är ansluten till grafentransistorn och innehåller den komplementära sekvensen för en specifik SNP. Den andra är en "svag" sträng där några av nukleotiderna ersätts med en annan molekyl för att försvaga dess bindningar till den normala strängen. En sträng som innehåller SNP kan binda starkare till den normala strängen och förskjuta den svaga strängen. Detta lämnar DNA-pincetten med en netto elektrisk laddning som enkelt kan detekteras av grafen-transistorn.
Nytt och förbättrat SNP -detekteringschip
Detta arbete bygger på det första etikett- och förstärkningsfria elektroniska SNP-detekteringschipet som Lals team tidigare utvecklat i samarbete med Gennadi Glinksy, en forskare vid UC San Diego Institute of Engineering in Medicine, och andra UC San Diego -forskare. Det nya chipet har lagt till trådlös kapacitet och är minst 1, 000 gånger känsligare än sin föregångare.
Det som gör det nya chipet så känsligt är utformningen av DNA-pincetten. När den SNP-innehållande strängen binder, det öppnar upp DNA-pincetten, ändra sin geometri så att de blir nästan parallella med grafenytan. Detta bringar DNA:s nettoladdning nära grafenytan, ger en större signal. I kontrast, DNA -sonden inbyggd i det tidigare chipet har en struktur som inte kan föras närmare grafenytan, så det genererar en svagare signal vid bindning av en SNP-innehållande sträng.
Nästa steg inkluderar att designa arraychips för att upptäcka upp till hundratusentals SNP i ett enda test. Framtida studier kommer att innebära att testa chipet på blod och andra kroppsvätskeprover tagna från djur eller människor.