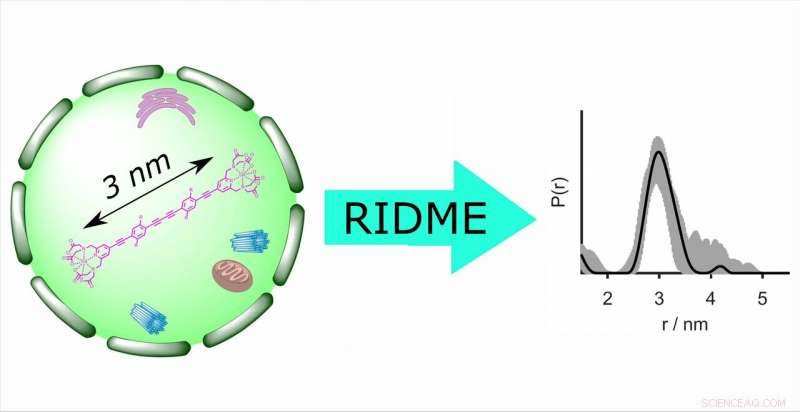
Avståndsbestämning i celler med EPR avslöjar viktig strukturell information om biomakromolekyler under naturliga förhållanden. För första gången, den pulsade EPR-tekniken RIDME (avslappningsinducerad dipolär moduleringsförbättring) användes för avståndsmätningar inuti celler. Det ger en fem gånger förbättrad känslighet jämfört med den tidigare använda dubbla elektron-elektronresonansmetoden. Kredit:Forskargruppen Professor Malte Drescher, Universitetet i Konstanz
I en gemensam tidning, forskare från universitetet i Konstanz, Bielefeld University och ETH Zürich visar för första gången att elektronparamagnetisk resonans (EPR)-tekniken RIDME (avslappningsinducerad dipolär modulering) kan användas för att bestämma avstånden mellan gadolinium(III)-baserade spinnetiketter i celler. Avståndsbestämning i cellen genom elektronparamagnetisk resonans (EPR) avslöjar viktig strukturell information om biomakromolekyler, inklusive deras konformation såväl som viknings- och utvikningsprocesser.
Konventionella metoder för cellbestämning av avstånd såsom dubbel elektron-elektronresonans (DEER eller PELDOR) är i princip mycket mindre känsliga än RIDME, ger upp till fem gånger mindre moduleringsdjup, har vissa begränsningar med avseende på exciteringsbandbredd och är tekniskt mer krävande. Som en enkelfrekvensteknik som använder sig av avslappningsinducerade spin flips för att bestämma avståndet mellan två spinnetiketter, dvs mellan två oparade elektroner, RIDME övervinner alla dessa nackdelar.
Denna teknik gör det möjligt för forskarna att arbeta med molekyler under naturliga förhållanden, som professor Malte Drescher och huvudförfattare Dr Mykhailo Azarkh, båda från universitetet i Konstanz, betona:"Vi började med att analysera konformationen av ett protein inuti cellen. Med mindre känsliga tekniker, vi tvingas infoga och tagga mycket protein för att kunna observera det, vilket inte alls är vad som händer i naturen. Helst vi vill arbeta med koncentrationer som är fysiologiskt relevanta. Eftersom RIDME är mycket känsligare än DEER, det tillåter oss att göra just det. Vi är nu i en position att ta itu med frågor som vi annars inte skulle kunna ta itu med."
Prestandan hos RIDME i cellen utvärderades vid Q-band med hjälp av styva molekylära linjaler märkta med Gd(III)-PyMTA och mikrofogade i Xenopus laevis (afrikansk klogroda) oocyter. Med andra ord, forskarna använde ett modellsystem där det exakta avståndet mellan spinnetiketterna redan var känt, så att de kan verifiera RIDME-mätningarna. Den resulterande artikeln med titeln "Gd(III)–Gd(III) Relaxation-Induced Dipolar Modulation Enhancement for In-Cell Electron Paramagnetic Resonance Distance Determination" publicerades online i Journal of Physical Chemistry Letters den 13.03.2019.
In-cell RIDME-avståndsbestämning utvecklades och testades som en del av det pågående ERC-finansierade projektet "SPICE – Spectroscopy in cells", för vilken Malte Drescher, Heisenberg professor för spektroskopi av komplexa system vid universitetet i Konstanz, och hans forskargrupp erkändes med ett ERC Consolidator Grant värt cirka två miljoner euro 2017. Deras mål är att utveckla nya metoder för spektroskopi som tillåter dem att utforska större och mer komplexa biologiska strukturer på cellens molekylära nivå.
Nästa steg i denna forskningslinje kommer att vara att identifiera andra lämpliga spin-etiketter och att utveckla RIDME för användning i molekyler där avståndet mellan spin-etiketterna är okänt. Särskilt fokus kommer att ligga på molekyler associerade med neurodegenerativa sjukdomar som Alzheimers och Parkinsons.