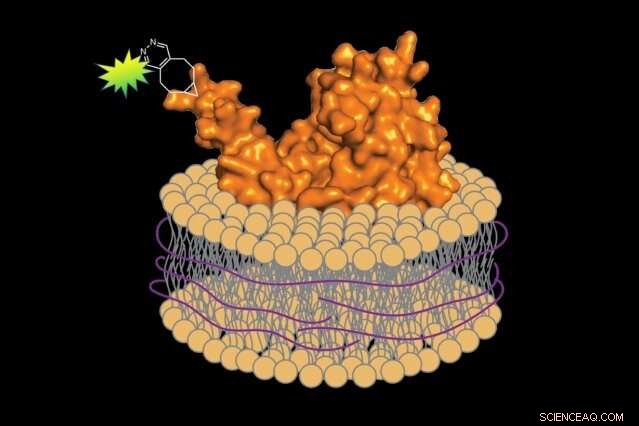
Med en diskmedelsfri metod, biologer kan förbereda fluorescensmärkta proteiner tillsammans med ett litet segment av det associerade cellmembranet, bevara proteinets naturliga miljö. Kredit:Jean-Marie Swiecicki
Alla celler har ett lipidmembran som omger deras inre komponenter - bildar en skyddande barriär för att kontrollera vad som kommer in och vad som stannar utanför. Proteinerna inbäddade i dessa membran är viktiga för livet; de hjälper till att underlätta transport av näringsämnen, energiomvandling och lagring, och mobilkommunikation. De är också viktiga vid mänskliga sjukdomar, och representerar cirka 60 procent av godkända läkemedelsmål. För att studera dessa membranproteiner utanför cellens komplexitet, forskare måste använda tvättmedel för att ta bort membranet och extrahera dem. Dock, att bestämma det bästa tvättmedlet för varje protein kan innebära omfattande försök och misstag. Och, Att ta bort ett protein från dess naturliga miljö riskerar att destabilisera den veckade strukturen och störa funktionen.
I en studie publicerad den 9 december in Cellkemisk biologi , forskare från MIT utarbetade ett snabbt och generaliserbart sätt att extrahera, rena, och märka membranproteiner för avbildning utan tvättmedel alls – ta med en del av det omgivande membranet för att skydda proteinet och simulera dess naturliga miljö. Deras tillvägagångssätt kombinerar väletablerade kemiska och biokemiska tekniker på ett nytt sätt, effektivt isolera proteinet så att det kan fluorescensmärkas och undersökas under ett mikroskop.
"Jag skämtar alltid om att det inte är särskilt verklighetstroget att studera proteiner i tvål, " säger seniorförfattaren Barbara Imperiali, professor i biologi och kemi. "Vi har skapat ett arbetsflöde som gör att membranproteiner kan avbildas samtidigt som de bibehåller deras inhemska identiteter och interaktioner. Förhoppningsvis kommer nu färre människor att dra sig för att studera membranproteiner, med tanke på deras betydelse i många fysiologiska processer."
Som medlem i Imperiali-labbet, tidigare postdoc och huvudförfattare Jean-Marie Swiecicki undersökte membranproteiner från den livsmedelsburna patogenen Campylobacter jejuni. I den här studien, Swiecicki fokuserade på PglC och PglA, två membranproteiner som spelar en roll för att bakterierna ska kunna infektera mänskliga celler. Hans experiment krävde märkning av PglC och PglA med fluorescerande taggar för att spåra dem. Dock, han var inte nöjd med befintliga metoder för att göra det.
I vissa fall, de fluorescerande taggar som måste inkorporeras i proteinet för att visualisera det är för stora för att placeras på definierade positioner. I andra fall, dessa taggar lyser inte tillräckligt starkt, eller stör proteinets struktur och funktion.
För att undvika sådana problem, Swiecicki bestämde sig för att använda en metod som kallas "onaturlig aminosyramutagenes." Aminosyror är de enheter som utgör proteinet, och onaturlig aminosyramutagenes involverar tillsats av en ny aminosyra innehållande en konstruerad kemisk grupp inom proteinsekvensen. Denna kemiska grupp kan sedan märkas med en starkt glödande tagg.
Swiecicki infogade den genetiska koden för C. jejuni-membranproteinerna i en annan bakterie, Escherichia coli. Inuti E. coli, han kunde inkorporera den onaturliga aminosyran, som skulle kunna modifieras kemiskt för att lägga till den fluorescerande märkningen.
När det var dags att ta bort proteinerna från membranet, han ersatte tvättmedlet med ett annat ämne:en polymer av styren-maleinsyra (SMA). Till skillnad från tvättmedel, SMA lindar in det extraherade proteinet och ett litet segment av det associerade membranet i ett skyddande skal, bevara sin ursprungliga miljö. Imperiali förklarar, "Det är som en halsduk som skyddar din nacke från kylan."
Swiecicki kunde sedan övervaka de glödande proteinerna under ett mikroskop för att verifiera att hans teknik var tillräckligt selektiv för att isolera enskilda membranproteiner. Hela processen, han säger, tar bara några dagar, och är i allmänhet mycket snabbare och mer tillförlitlig än tvättmedelsbaserade extraktionsmetoder, som kan ta månader och kräver expertis från högutbildade biokemister för att optimera.
"Jag skulle inte säga att det är en magisk kula som kommer att fungera för varje enskilt protein, " säger han. "Men det är ett mycket effektivt verktyg som kan göra det lättare att studera många olika typer av membranproteiner." Så småningom, han säger, det kan till och med hjälpa till att underlätta undersökningar av läkemedel med hög genomströmning.
"Som någon som arbetar med membranproteinkomplex, Jag kan intyga det stora behovet av bättre metoder för att studera dem, " säger Suzanne Walker, en professor i mikrobiologi vid Harvard Medical School som inte var inblandad i studien. Hon hoppas kunna utvidga tillvägagångssättet som beskrivs i artikeln till proteinkomplexen hon undersöker i sitt eget labb. "Jag uppskattade den omfattande detaljen i texten om hur man framgångsrikt tillämpar strategin, " tillägger hon.
Nästa steg kommer att testa tekniken på däggdjursproteiner, och isolering av flera proteiner samtidigt i SMA-skalet för att observera deras interaktioner. Och, självklart, varje ny teknik förtjänar ett namn. "Vi arbetar fortfarande på en catchy akronym, " säger Imperiali. "Några idéer?"
Den här historien återpubliceras med tillstånd av MIT News (web.mit.edu/newsoffice/), en populär webbplats som täcker nyheter om MIT-forskning, innovation och undervisning.