
En illustration visar ett stort histokompatibilitetsprotein (grått) som omfattar en peptid hämtad från ett SARS-CoV-virus (rosa). Komplexet hjälper till att utlösa aktiveringen av T-celler som är en del av immunsystemet. Rice University forskare upptäckte en icke-ankarbindande rest i peptiden som både kunde bidra till bindning och till T-cellsaktiveringen som behövs för att besegra viruset. Kredit:Kavraki Lab/Rice University
"Position 4" verkade inte vara viktig förrän forskarna tittade länge på en viss peptid.
Den del av peptiden som hämtats från ett SARS-CoV-virus visade sig ha en oväntad men betydande inverkan på hur den stabilt binder till en receptor som är central för immunsystemets förmåga att attackera sjuka celler.
I en studie publicerad av Proceedings of the National Academy of Sciences , forskare vid Rice Universitys Brown School of Engineering och University of Texas MD Anderson Cancer Center avslöjade modeller vid atomär upplösning som inte bara beskriver bindningen utan också, för första gången, de obindande mekanismerna som ligger till grund för en nyckelkomponent i immunsystemet.
De säger att bättre förståelse för hela mekanismen kan leda till framsteg inom immunterapi som ökar kroppens förmåga att bekämpa sjukdomar.
Risdataforskaren Lydia Kavraki, alumnen Jayvee Abella och postdoktorn Dinler Antunes, ledde studien.
"Att hitta bra mål för att utlösa ett skyddande immunsvar är mycket utmanande, särskilt inom cancerforskning, " Sa Antunes. "Faktumet att just denna peptid förutspåddes att inte binda till HLAs (humana leukocytantigener) genom sekvensbaserade metoder framhäver en blind fläck i vår nuvarande förutsägelsekapacitet.
"Genom att införliva strukturanalys, vi kan upptäcka bidraget från dessa sekundära interaktioner till peptidbindning och stabilitet, förhoppningsvis gör det möjligt för oss att hitta bättre mål för utveckling av antivirala vacciner och T-cellsbaserad cancerimmunterapi, " han sa.
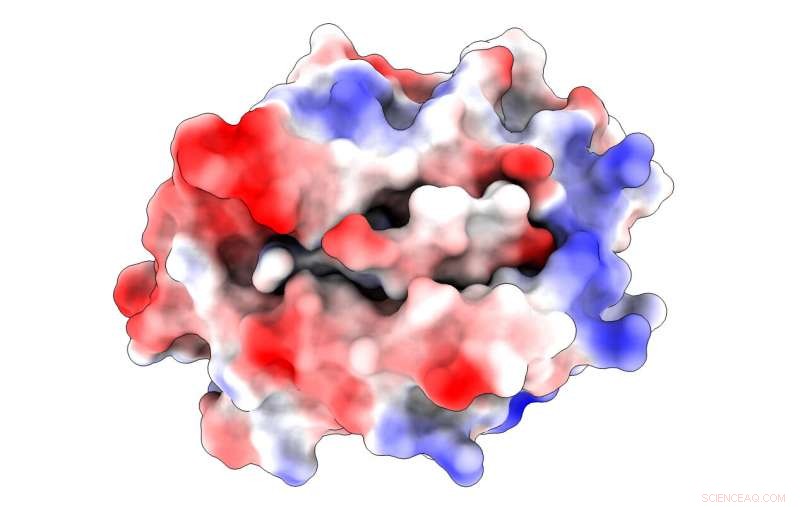
En illustration visar de elektrostatiska laddningarna (blått är positivt, röd negativ) i ett stort histokompatibilitetsprotein bundet till en peptid hämtad från ett SARS-CoV-virus. Komplexet hjälper till att utlösa infektionen av T-celler som är en del av immunsystemet. Rice University forskare upptäckte en icke-ankarbindande rest i peptiden som både kunde bidra till bindning och till T-cellsaktiveringen som behövs för att besegra viruset. Kredit:Kavraki Lab/Rice University
Forskarna använde sina simuleringar för att belysa detaljer om hur den intracellulära SARS-peptiden, QFKDNVILL, binder till ett MHC-receptorprotein som kallas HLA-A*24:02, primärt vid dominerande ankare på båda ändarna av peptiden (vid positionerna 2 och 9) och presenterar dem för inspektion för immunsystemets T-celler.
Stabil bindning av en peptid och MHC är en förutsättning för aktivering av T-celler, som letar efter peptider som normalt inte finns i friska celler. Om peptiden och proteinet inte binder, T-cellen uppmanas inte att attackera.
"Så mycket var känt från tidigare studier av de bundna och obundna tillstånden för många sådana komplex, ", sa Kavraki. "Vad de inte fångade var de mellanliggande tillstånden och övergångarna som leder från ett tillstånd till ett annat, speciellt det obindande.
"Jag tror att det här är den enda analysen som visar avbindningen av peptider från MHC med atomär upplösning, Kavraki sa. "Andra peptider har liknande egenskaper och vi tror att de skulle ha liknande beteenden."
Alla dessa interaktioner avslöjades i detalj genom Markov-tillståndsmodeller som analyserar hur system förändras över tiden. I detta fall, modellerna avslöjade vikten av sekundära ställen som stödjer peptidens primära ankare. Det var där position 4 stack ut.
"Det finns de viktigaste, kanoniska ankare som folk känner, men det finns dessa sekundära interaktioner som bidrar till bindningen och stabiliteten, " sa Antunes. "De här är svårare att fånga, men i denna studie, det verkar som om position 4 spelar en mycket viktig roll. När du muterar det, det påverkar beteendet hos peptiden när den frigörs från molekylen."
Forskarna modellerade mutationer av MHC för att se hur de skulle påverka bindningen och fann att de stödde betydelsen av position 4 för komplexets stabilitet.
Elektrostatiska potentiella kartor skapade från modeller av Rice University visar ett stort histokompatibilitetsprotein bundet till en peptid från SARS-CoV-virus. Sådana dynamiska komplex kan utlösa aktiveringen av T-celler som är en del av immunsystemet. Rice-teamet upptäckte den stabiliserande inverkan av en alternativ bindningsrest (vid position 4) gemensam för både peptiden de studerade, överst, och en associerad med SARS-CoV-2, på botten, ansvarig för covid-19-sjukdomen. Kredit:Kavraki Lab/Rice University
"Vår beräkningsmetod kunde göra förutsägelser om effekten av mutationer som sedan experimentellt verifieras, " sa medförfattaren Cecilia Clementi, en före detta risprofessor som nyligen blev Einstein-professor i fysik vid Free University of Berlin.
Forskarna utvecklade en tvåstegsprocess för att förenkla beräkningskomplexiteten i atomskalaanalys av stora molekyler. Det första steget använde en teknik som kallas paraplyprovtagning för att påskynda den initiala utforskningen av molekylerna. Den andra, utforskande skede använde adaptiv sampling, där simuleringar drivs för att påskynda konstruktionen av Markov-modellen.
"Utmaningen är att dessa MHC är ganska stora system för beräkningskemister att simulera, sa Abella, vars forskning i ämnet utgjorde mycket av hans doktorsavhandling. "Vi var tvungna att göra några uppskattningar och utnyttja framsteg i dessa klasser av metoder för att gå vidare.
"Vi är inte de första att studera obindande, men det som kännetecknar vårt arbete framför andra är att vi håller full atomupplösning i våra simuleringar, " sade han. "Andra verk använder en teknik som kallas en Markov-kedja Monte Carlo, medan vi använder molekylär dynamik, som låter oss införliva tid i vår beräkning för att fånga kinetiken."
Deras metoder kan tillämpas på andra peptid-MHC-komplex med befintliga 3-D-modeller. "Detta var, på något vis, en genomförbarhetsstudie för att visa att vi kan använda molekylär dynamik och bygga en Markov-tillståndsmodell av ett system av denna storlek, sa Abella.
Forskarna noterade också studiens relevans för den nuvarande kampen mot COVID-19, som SARS-peptiden de såg, QFKDNVILL, är mycket lik NFKDQVILL-peptiden i SARS-CoV-2, med samma bindningsfickor i position 2, 4 och 9.
"Dessa resultat tyder på att båda peptiderna kan binda till HLA-A*24:02 och tillhandahålla mål för antivirala T-cellssvar, som är av stort intresse i ljuset av den nuvarande pandemin, " sa medförfattaren Gregory Lizée, en professor vid avdelningen för melanom medicinsk onkologi vid MD Anderson. "Men dessa resultat kastar också ljus över många andra potentiella immunmål, inklusive de från andra virus och till och med mänskliga cancerformer."
Kavraki noterade att experimentellt arbete av den långvariga samarbetspartnern Lizée och Kyle Jackson, en forskarassistent vid Lizées labb som producerade mutantproteinerna, var avgörande för att validera sina simuleringar. Kavrakis eget labb vann ett Rapid Response Research-anslag från National Science Foundation (NSF) för att hjälpa till att identifiera fragment av SARS-CoV-2 virala proteiner som möjliga mål för vaccinutveckling.