Intrinsically disordered proteiner (IDP) finns i stor utsträckning i eukaryoternas proteomer och spelar nyckelroller i livsprocesser som transkription av genetisk information och signalering. Förutom att de vanligtvis är mycket repetitiva, hydrofila och elektriskt laddade samt kodar för enkla sekvenser av gener, utmärker sig IDP:er också i sin naturliga överflöd och strukturella aspekter, som blir grunden för proteinernas "störningsfunktionsparadigm".
Under de senaste två decennierna har internflyktingars roll i mänskliga sjukdomar och som läkemedelsmål aktivt studerats, medan hur man karakteriserar de mycket flexibla och heterogena konformationerna hos internflyktingar med hög upplösning förblir en nyckelfråga på detta område.
Mer än 15 % av IDP-molekylerna är membranbundna i celler, och deras interna dynamik och övergripande (translationella och roterande) rörelser inom fosfolipiddubbelskikten är nära relaterade till deras fysikalisk-kemiska egenskaper och biologiska funktioner, men dessa dynamiska processer är svåra att fånga och kvantitativt karakteriserad av konventionella strukturanalysmetoder.
En grupp forskare under ledning av prof. Long Dong från University of Science and Technology of China (USTC) har utvecklat en IDP-spektroskopimetod baserad på membranparamagnetic relaxation enhancement-teknik (mPRE), som framgångsrikt har uppnått högprecisionsmodellering av interna konformationer, orienteringar och nedsänkningsdjup hos IDP:er. Resultaten har publicerats i Journal of the American Chemical Society .
I detta arbete undersökte forskarna i detalj flexibiliteten och rörligheten hos spinnprobmolekylerna i membranet för korrekt tolkning av mPRE-spektraldata, och föreslog en viktad tredimensionell (3D) rutnätsmodell baserad på allatomsimuleringar för kvantitativt skildrar effekten av spinprobens dynamik på membranets paramagnetiska avslappningsförstärkningshastighet.
Genom att dra nytta av modellens höga beräkningseffektivitet utvecklade forskarna en algoritm som är detaljerad i Supporting Information (SI) för att optimera orienteringen av de globala och interna rörelsefrihetsgraderna för membranbundna IDP:er via superposition av z- Endast koordinater, skräddarsydda för mPRE-dataanalysen, konstruerar en allatom-ensemblemodell av IDP i en implicit membranmiljö.
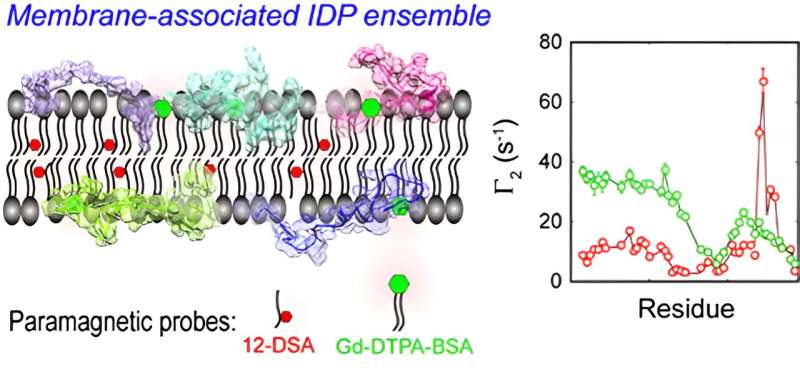
CD3ε är en komponent i T-cellsreceptorkomplexet (TCR) som ansvarar för igenkänning av T-cellsantigen. CD-skivan3ε cytoplasmatisk domän (CD3εCD ) innehåller immunoreceptortyrosinbaserade aktiveringsmotiv (ITAMs), och det bildar ett flummigt komplex med lipiddubbelskikt i ett i sig oordnat tillstånd, och reglerar signalaktiviteten hos fuzzy komplex genom att använda dynamisk membranavskärmning av viktiga tyrosinställen.
Forskarna löste ensemblen baserat på molekylär dynamik hos CD3εCD i lipiddubbelskikt genom att applicera en all-atom ensemble modelllösning av IDP i en implicit membranmiljö.
Ensemblen från mPRE experimentella parametrar kartlägger den dynamiska fördelningen av CD3εCD i olika regioner av membranet på atomnivå och avslöjar viktiga skillnader i membraninteraktionerna mellan olika tyrosinställen på ITAM, vilket ger en ny mekanistisk förklaring till monofosforyleringsmönstret för ITAM.
Den mPRE-spektroskopiska analysmetoden som etablerats i detta arbete förväntas i stor utsträckning underlätta atomupplösningsstudier av olika funktionella membran-IDP.
Mer information: Hong Jin et al, Quantitative Ensemble Interpretation of Membrane Paramagnetic Relaxation Enhancement (mPRE) for Studying Membrane-Associated Intrinsically Disordered Proteins, Journal of the American Chemical Society (2023). DOI:10.1021/jacs.3c10847
Journalinformation: Tidskrift för American Chemical Society
Tillhandahålls av University of Science and Technology i Kina