
Utdrag från time-lapse-filmen för miRNA-detektion på 1 timme med hjälp av miRacles-analys. Kredit:Science Advances, doi:10.1126/sciadv.aau9443
MikroRNA (miRNA) är korta icke-kodande regulatoriska RNA som kan undertrycka genuttryck post-transkriptionellt och används därför alltmer som biomarkörer för sjukdomar. Att upptäcka miRNA kan vara svårt och dyrt eftersom de kräver amplifiering, märkning och radioaktiva sonder. I en nyligen publicerad rapport om Vetenskapens framsteg , Arun Richard Chandrasekaran och medarbetare vid RNA-institutet och Institutionen för biologiska vetenskaper, vid University of Albany, State University of New York, rapporterade i ett steg, icke-enzym mikroRNA-detektionsanalys med användning av DNA-nanoswitchar som inte svarar på konformationen.
Forskarna kallade analysen "mirakel, ' för att förkorta 'mikro-RNA-aktiverad villkorad looping av konstruerade switchar'. Analysen visade subattomol- och enkelnukleotidspecificitet med användning av en agarosgelelektroforesavläsning. I experimenten, de upptäckte cellulära mikroRNA från RNA-extraktioner erhållna från differentierande muskler på nanogramskalan. Forskarna presenterade en kostnadseffektiv experimentell uppsättning för att upptäcka miRNA inom en timmes tidsram för att tillhandahålla ett övertygande alternativ till de befintliga metoderna för kvantitativ polymeraskedjereaktion (qPCR) och Northern blotting för att kvantifiera de reglerande genetiska materialen.
miRNA kan reglera många biologiska processer under normal fysiologisk utveckling och sjukdom genom att påverka cellproliferation, differentiering och apoptos in vivo. Uttrycket av miRNA kan kvantifieras i vävnader, celler och kroppsvätskor som stabila biomarkörer för cellulära händelser och sjukdomsdiagnostik, betonar vikten av att de upptäcks. Ändå, miRNA-detektion är utmanande på grund av låg förekomst, liten storlek och likheter i följd. Biomolekylerna utgör cirka 0,01 procent av det totala RNA-innehållet och individuella mRNA-kopior per cellområde kan variera kraftigt. Dessutom, miRNA i en familj kan skilja sig åt med en enda nukleotid, medan specifika miRNA kan regleras under sjukdom och regelbunden cellulär funktion. Som ett resultat, Detektionsstrategierna för miRNA kräver hög specificitet och förmågan att korrekt identifiera ett fåtal molekyler från ett prov som är rikligt med dominerande RNA-molekyler.
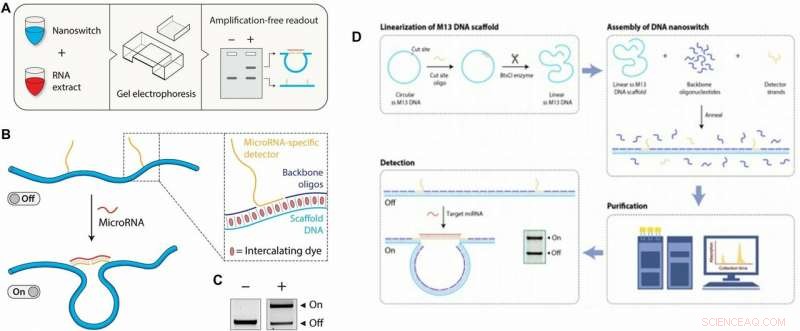
Till vänster:Koncept och arbetsflöde för miracles-analysen. (A) Arbetsflöde för miRacles-analysen:Anpassade DNA-nanoswitchar blandas med mål-miRNA-prov, inkuberade, och kör på en agarosgel för detektion. (B) DNA nanoswitchar genomgår en konformationell förändring från ett linjärt "av" tillstånd till ett loopat "på" tillstånd när de är bundna till ett mål miRNA. Infälld:Nanoswitchen är sammansatt av en enkelsträngad M13-ställning, ryggradsoligonukleotider, och enkelsträngade förlängningar (detektorer) komplementära till mål-miRNA. Interkalerande färgämnen som är inneboende i elektroforesprocessen ger signalen för att visualisera nanoswitcharna. (C) De två konformationerna är lösbara i en standard agarosgel. Till höger:Fördjupad undersökning av DNA-nanoswitchkonstruktionsprocessen. (D) Linjärisering:Framställning av en linjär enkelsträngad M13-ställning fortsätter genom att hybridisera en oligonukleotid för att möjliggöra klyvning vid det dubbelsträngade stället av ett restriktionsenzym (BtsCI i detta fall). Montering:DNA-nanoswitchmontering utförs genom att blanda ett 10x molärt överskott av ryggradsoligonukleotider och detektorsträngar (specifika för målet) till den enkelsträngade M13 och applicera en kylramp från 90˚C till 25˚C vid 1˚C/min. . Rening:För att ta bort överskott av oligonukleotider, en vätskekromatografimetod används. Detektering:För att använda nanoswitcharna, de blandas med en lösning som innehåller mikroRNA-mål och körs på en gel för att lösa på och av tillstånden som visas i insättningen. Kredit:Science Advances, doi:10.1126/sciadv.aau9443
Traditionella metoder för miRNA-detektion inkluderar Northern blotting, kvantitativ omvänd transkriptionspolymeraskedjereaktion (qRT-PCR), nästa generations sekvensering och mikroarraybaserad hybridisering för att separera miRNA-signalen från brus. Av de angivna metoderna, Northern blotting kan direkt identifiera infödda miRNA, medan andra förlitar sig på ytterligare märkningsmetoder eller stegvis amplifiering, lägga till kostnaden, detektionens komplexitet och prestanda. Till exempel, innovativa DNA-nanostrukturer kan användas för miRNA-detektion, där olika forskargrupper tidigare har kombinerat nanostrukturer med nanopartiklar, hybridiseringskedjereaktioner och övergångsmetall dikalkogenid nanosheets för att möjliggöra processen.
I detta arbete, Chandrasekaran et al. utvecklat en relativt enkel DNA-baserad enhet för att lösa en komplex biomedicinsk utmaning. I mirakelanalysen, forskarna använde ett "smart reagens" bestående av rationellt utformade DNA-nanoswitchar för enkel och kostnadseffektiv infödd miRNA-detektion utan att använda specialutrustning i labbet. DNA-nanoswitchar designades ursprungligen som verktyg för biofysikexperiment med en molekyl och erkändes senare för sin förmåga att detektera och kvantifiera biomolekylära interaktioner med hjälp av gelelektrofores. Tidigare forskningsinsatser från samma forskargrupp fokuserade på molekylär detektion för att kvantifiera proteinnivåer och detektera syntetiska DNA-sekvenser som ett bevis på konceptet.
Det nuvarande arbetet utökade med preliminära studier och koncept för att producera användarklar multiplexerad miRNA-detektion och kvantifiering. Forskarna analyserade nanogram av cellulära RNA-extrakt inom en kort tidsram med hjälp av en experimentell uppsättning byggd med vanliga laboratoriematerial. De designade DNA-nanoswitchen som en linjär duplex som bildade en slinga i närvaro av mål-miRNA-molekylen. För att konstruera nanoswitchen, Chandrasekaran et al. använde DNA-origami-metoden genom att hybridisera korta oligonukleotider som är komplementära till en enkelsträngad DNA-ställning.
De designade två avlägsna "detektor"-strängar med överhäng som är komplementära till olika segment av mål-miRNA. När miRNA kände igen och band till konstruktionen, omkopplaren omkonfigurerad från det linjära "av"-tillståndet till det loopade "på"-tillståndet. De kvantifierade de två tillstånden med hjälp av standard agarosgelelektrofores för att detektera signalen som härrör från den loopade nanoswitchen. Signalen förstärktes endast av ett enda miRNA av intresse, resultaten jämfördes positivt med tekniken för fluorescensresonansenergiöverföring (FRET).

Validering av miRacles-analysen. (A) Specificitet för DNA-nanoswitcharna med detektorer utformade för let-7b. Så låg som 1-nt missmatch mellan detektorerna och målet miRNA eliminerar signalen. (B) Detektionsgräns för analysen. NC, negativ kontroll. AU, godtyckliga enheter. (C) Analysens tidsförlopp för ett mål med låg koncentration. (D) Dynamiskt område för analysen vid olika reaktionstider. Kredit:Science Advances, doi:10.1126/sciadv.aau9443.
För att validera konceptet, forskarlaget valde ett let-7b-mål miRNA på grund av dess mycket konserverade familj av mer än ett dussin-relaterade miRNA som varierade med en eller flera nukleotider. Dessa miRNA var lämpade på grund av deras kritiska roll i biologiska funktioner och dysreglering i flera mänskliga sjukdomar. För att eliminera överhörning och brus mellan nanoswitchen och målet – vilket ledde till minskad signalintensitet jämfört med en perfekt matchning, forskarna gjorde rationellt om nanoswitcharna. För att uppnå perfekt specificitet, de destabiliserade interaktionen på den sida som innehöll obalansen. Resultaten av studien illustrerade hög specificitet hos analysen som sålunda utvecklats, ger ett svar på en nyckelutmaning i miRNA-detektion som kulminerade från ett högt signal/brusförhållande.
Den låga mängden miRNA krävde också hög känslighet för detektion, vilket forskarna uppnådde genom att optimera protokollet. De utförde sedan liknande experiment för två andra varianter av miRNA (miR-15 a och miR-206), vilket resulterar i detekteringsnivåer av subattomol till enatomol. Till exempel, för en låg koncentration av ett målprov (6 pM), signalen ökade i 4 timmar, med liten förändring utöver den tidsramen.
miRNA-detektion från differentierande myoblastceller. (A) Schematisk som visar myoblastceller, skördas under odling i GM och på differentieringsdag 1 och 4, bearbetas för att ge totala och små RNA-fraktioner. En tidig myogen differentieringsmarkör, Myog, och en sen myogen differentieringsmarkör, MHC, mättes med (B) Western blotting och (C) med immuncytokemi för att bekräfta differentiering. Både Myog och MHC uppreglerades i DM1 och DM4. GAPDH (glyceraldehyd-3-fosfatdehydrogenas) fungerade som kontroll i (B). (D) Chandrasekaran et al. upptäckte miR-206 i de differentierade proverna med 50 ng små RNA och (E) med 500 ng totalt RNA. Kvantifiering av gelintensiteter visar en kraftig progressiv uppreglering under differentiering, liknande i både (F) små RNA- och (G) totala RNA-prover. Från DM4-prover, vi noterar detektion från så lite som (H) 200 pg små RNA och (I) 500 pg totalt RNA. Kredit:Science Advances, doi:10.1126/sciadv.aau9443. 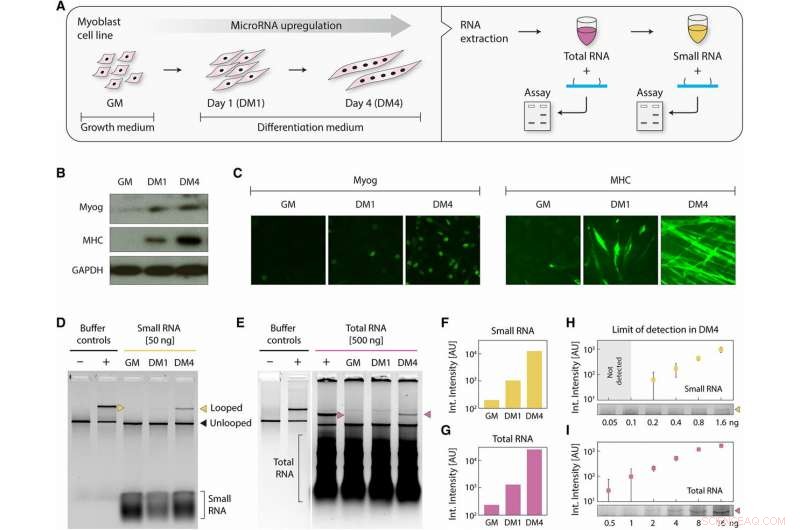
Eftersom flera mRNA också kan ändra sitt uttryck under olika cellulära eller sjukdomsstadier, fenomenet krävde ytterligare detekteringsmöjligheter. För att åstadkomma detta i experimentuppställningen, Chandrasekaran et al. använde nanoswitchars programmerbarhet och utvecklade ett multiplexeringssystem som kan detektera flera miRNA från samma prov. De placerade detektorsträngarna på önskade platser i DNA-ställningen, vilket resulterar i slingor av olika storlekar när de binds till mål-miRNA. Slingstorleken på nanoswitchen bestämde sålunda gelmigreringen, vilket resulterar i ett unikt band på gelén för exakt detektering. För experimentet, forskarna valde fyra miRNA som finns i muskelceller; miR-206, miR-125b, miR-24 och miR-133-3p och ett negativt kontrollmiRNA; miR-39 specifikt för arten Caenorhabditis elegans.
Inom 50 ng av små RNA, forskarna upptäckte fyra miRNA vid olika uttrycksnivåer, samtidigt som ingen detektering bekräftas med den negativa kontrollen. Multiplexeringsstrategin gjorde det möjligt för forskarna att direkt jämföra miRNA-nivåer i ett enda prov, utan märkning eller förstärkning. Totalt, arbetet gav ytterligare ett steg i riktning mot att utöka genomströmningen av miRacles-analysen. Förmågan kan utökas för att rymma fler miRNA per switch också.
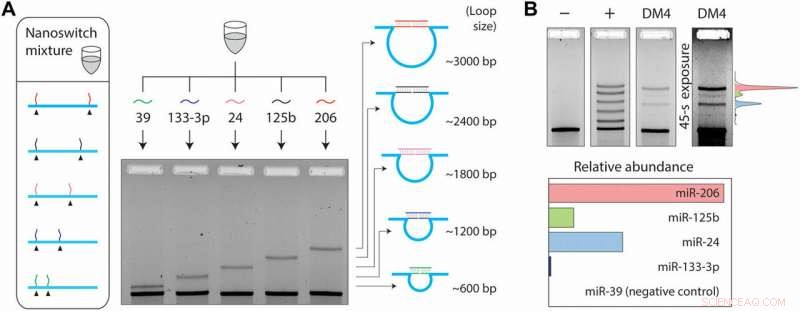
Fem-kanals multiplexeringsinställning. (A) Multiplexering möjliggör detektering av olika miRNA med olika loopstorlekar. (B) En multiplexerad nanoswitchblandning visar fem band med liknande intensitet i en positiv kontroll bestående av alla fem mål-miRNA. I 50 ng av DM4 små RNA, fyra olika miRNA detekteras vid olika uttrycksnivåer, med miR-39 (ett C. elegans-specifikt miRNA) som inte detekteras.
På det här sättet, Chandrasekaran et al. avsevärt gått framåt från deras preliminära proof-of-concept-detektion av syntetiska DNA-sekvenser; att etablera, karakterisera och optimera en användningsfärdig miRNA-detektionsanalys med biologiska extrakt. Det visade arbetet var ett första exempel på att använda DNA-nanoswitchar för att detektera miRNA från ett äkta biologiskt prov. Medan prestandan för miRNA-analysen var konkurrenskraftig jämfört med andra vanliga tekniker, selektiviteten för 1 nukleotid som ses i föreliggande arbete var svår att åstadkomma med befintliga metoder. Miracles känslighet överträffade också Northern blotting och mikroarrayer. Analysen kunde mäta miRNA utan att behöva amplifiering, med enklare protokoll och utan det extra felet med extra provbearbetning. Protokollet blandade helt enkelt nanoswitcharna med provvätskan för gelelektrofores, att producera högkvalitativa resultat i labbet. Forskningsarbetet är potentiellt överförbart från biologiska till kliniska prover för att diagnostisera och övervaka sjukdom.
Mer viktigt, föreliggande arbete är i linje med det bredare begreppet sparsam vetenskap; en lovande vision om kostnadseffektiv vetenskap som redan har producerat lågkostnadslösningar för blodcentrifugering och vattenreningstekniker inom biomedicinsk teknik. Chandrasekaran et al. sträva efter att fortsätta att bidra till den framväxande trenden inom vetenskap, genom att störa det befintliga kostnads/prestandaförhållandet för att ge bredskalig tillgång till enkla miRNA-detektionsmetoder med smarta reagenser som blandas och läs.
© 2019 Science X Network