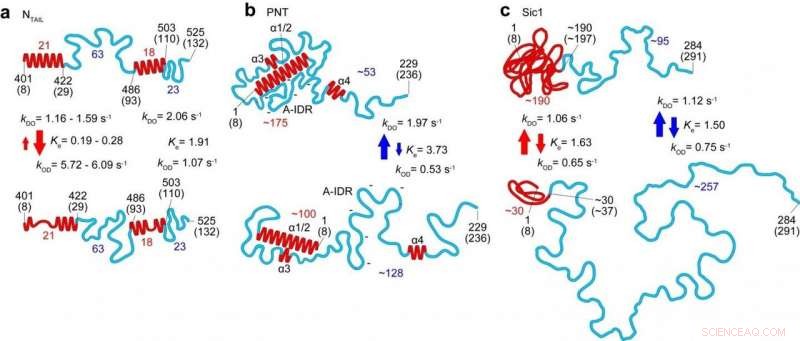
Strukturella och dynamiska egenskaper hos tre IDP:er (N SVANS , PNT och Sic1) avslöjas av HS-AFM-bildbehandling. De övre och nedre panelerna motsvarar de mer ordnade och mindre ordnade tillstånden, respektive. Siffrorna i rött och blått representerar antalet aminosyror som finns i respektive vikta regioner och helt oordnade regioner, respektive. De röda och blå pilarna indikerar den kinetiska karaktären hos förändringar i höjden av vikta områden och avstånd från ände till ände för helt oordnade områden, respektive. Kredit:Kanazawa University
Vår förståelse av biologiska proteiner korrelerar inte alltid med hur vanliga eller viktiga de är. Hälften av alla proteiner, molekyler som spelar en integrerad roll i cellprocesser, är i sig störda, vilket innebär att många av standardteknikerna för att sondera biomolekyler inte fungerar på dem. Nu har forskare vid Kanazawa University i Japan visat att deras egenodlade höghastighetsteknik för atomkraftsmikroskopi kan ge information inte bara om strukturerna hos dessa proteiner utan också om deras dynamik.
Att förstå hur ett protein är sammansatt ger värdefulla ledtrådar till dess funktioner. Utvecklingen av proteinkristallografi på 1930- och 1950-talen gav för första gången flera proteinstrukturer i synen, men det blev gradvis uppenbart att en stor del av proteiner saknar en enda struktur som gör dem svårbehandlade för röntgenkristallografi. Eftersom de är för tunna för elektronmikroskopi, de enda genomförbara alternativen för många av dessa intrinsically disordered proteiner (IDP) är kärnmagnetisk resonanstomografi och röntgenspridning med liten vinkel. Data som samlats in från dessa tekniker är medelvärde över ensembler och ger därför ingen tydlig indikation på individuella proteinkonformationer eller hur ofta de förekommer. Atomkraftsmikroskopi å andra sidan är kapabel till biologisk avbildning i nanoskala upplösning i hög hastighet, så det kan fånga dynamik såväl som proteinstrukturer.
I detta senaste arbete forskare vid Kanazawa University tillsammans med medarbetare i Japan, Frankrike och Italien tillämpade tekniken för att studera flera internflyktingar och identifierade parametrar som definierar formen, storlek och kedjelängd av proteinregioner, samt en maktlag som relaterar proteinstorleken till proteinlängden, och en kvantitativ beskrivning av glimmerytans effekt på proteindimensioner. Dynamiken i proteinkonformationerna som fångats tack vare teknikens höghastighetskapacitet avslöjade kulor som dyker upp och försvinner, och transformationer mellan helt ostrukturerade och löst vikta konformationer i segment upp till 160 aminosyror långa.
Studier av nukleoproteinet mässlingvirus i synnerhet hjälpte till att identifiera inte bara formen och dimensionerna utan också egenskaperna hos ordningsstörningsövergångarna i regionen som är ansvarig för molekylär igenkänning, vilket gör att virus kan identifiera värdfaktorer så att de kan reproducera sig. De skulle också kunna bestämma strukturer i större skala av virusets fosfoprotein som inte är tillgängliga för kärnmagnetisk resonans (vilket bara kan ge en indikation på avstånd mellan aminosyror separerade med mindre än 2 nm). Forskarna föreslår att bildandet av vissa kompakta former som observerats kan förklara motståndet mot proteolys - proteinnedbrytning.
I sin rapport om arbetet, forskarna framhåller att såväl som ett kraftfullt verktyg i sin egen rätt, "När alla molekylära egenskaper som avslöjas av HS-AFM kombineras med den vikta lokala strukturen som ges av NMR, den kombinerade informationen möjliggör en kvantitativ avgränsning av de strukturella och dynamiska karaktärerna hos internflyktingar, på ett mer realistiskt sätt jämfört med bilderna som avbildas individuellt, som visats för PNT [fosfoprotein från mässlingvirus]."
Höghastighets atomkraftsmikroskopi
Atomkraftsmikroskopi utvecklades på 1980-talet och förde upplösningen i atomskala som uppnåddes genom skanning av tunnelmikroskopi (som vann Nobelpriset i fysik 1986) till icke-ledande prover. Det fungerar med en liten konsol med en nanoskala spets i slutet, som antingen känns ytan ungefär som en vinylskivnål eller knackar på den. Oavsett om du justerar spetshöjden eller resonansfrekvensen för tappningen, the interactions between tip and surface provide a signal that can be used to generate an image.
While AFM images brought huge benefits to biological research, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
Intrinsically disordered proteins
The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.