Forskare från Griffith University har spelat en nyckelroll i att använda DNA-"origami"-mallar för att kontrollera hur virus samlas.
Det globala teamet bakom forskningen, med titeln "DNA-origami-directed virus capsid polymorphism", publicerad i Nature Nanotechnology , utvecklat ett sätt att styra sammansättningen av viruskapsider – proteinskalet hos virus – vid fysiologiska förhållanden på ett exakt och programmerbart sätt.
Dr Frank Sainsbury och Dr Donna McNeale från Griffith Institute for Drug Discovery var en del av forskargruppen och sa att det att tvinga virus att samlas på DNA som vikas till olika former "som origami" var en fråga som detta projekt besvarade.
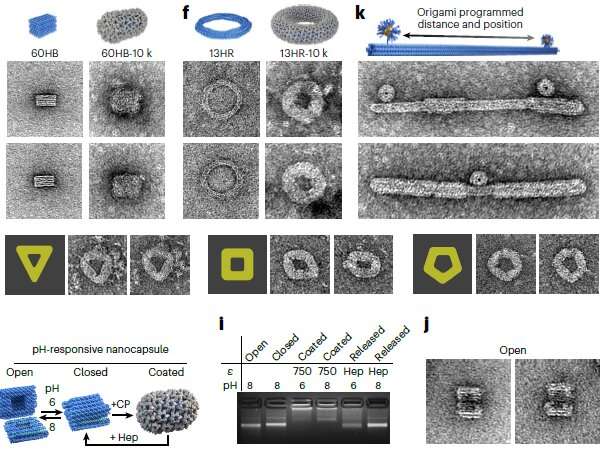
"Vi uppnådde kontroll över virusproteinets form, storlek och topologi genom att använda användardefinierade DNA-origami nanostrukturer som bindnings- och sammansättningsplattformar, som blev inbäddade i kapsiden," sa Dr. Sainsbury.
"Virusproteinbeläggningarna skulle kunna skydda den inkapslade DNA-origamin från nedbrytning.
"Denna aktivitet är mer som att slå in en present - virusproteinerna lägger sig ovanpå den olika formen som definieras av DNA-origamiformen.
"Och olika virusproteiner är som olika omslagspapper, vilket skulle vara relevant för olika användningar av den belagda DNA-origami."
Exakt kontroll över storleken och formen på virusproteiner skulle ha fördelar i utvecklingen av nya vacciner och leveranssystem.
"Men nuvarande verktyg för att styra monteringsprocessen på ett programmerbart sätt var svårfångade," sa Dr. McNeale.
"Vårt tillvägagångssätt är inte heller begränsat till en enda typ av viruskapsidproteinenhet och kan också tillämpas på RNA-DNA-origamistrukturer för att bana väg för nästa generations lastskydd och inriktningsstrategier."
För närvarande arbetar Dr. Sainsbury och hans team på att få en mer djupgående förståelse för hur olika virus självbildar sig och hur de kan användas för att kapsla in olika laster.
Detta kommer att tillåta dem att designa och modifiera ytterligare virusliknande partiklar för en rad olika användningsområden. Till exempel upptäckte de att ett virus som finns i möss kan transportera proteinlaster genom ogästvänliga miljöer och in i ett specifikt subcellulärt fack i mänskliga celler.
"Med det enorma befintliga designutrymmet bland virus som skulle kunna användas som bärare finns det fortfarande mycket att lära av att studera dem. Vi kommer att fortsätta att tänja på gränserna för hur virusliknande partiklar kan samlas och vad som kan läras av att använda dem som medicintransportörer, vacciner och biokemiska reaktionskärl," sa Dr. Sainsbury.
Nästa steg i GRIDD-teamets forskning kommer att använda detta tillvägagångssätt för att undersöka varför virus inte själva samlas i olika former.
Mer information: DNA-origami-riktad viruskapsidpolymorfism, Nature Nanotechnology (2023). DOI:10.1038/s41565-023-01443-x, https://www.nature.com/articles/s41565-023-01443-x
Journalinformation: Nanoteknik i naturen
Tillhandahålls av Griffith University