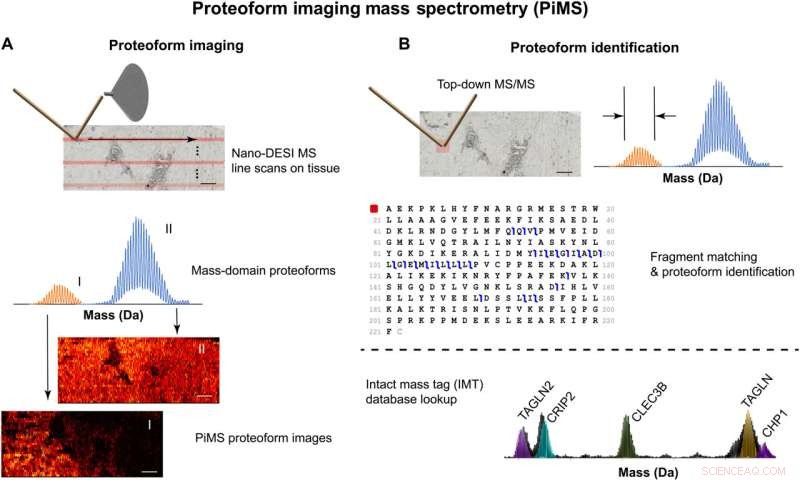
Illustration av PiMS-arbetsflödet för proteoformavbildning och identifiering. (A) Skanningsmetod (överst), detektion av proteoformer i massdomänen (mitten) och bildrekonstruktion (nederst). (B) Två tillvägagångssätt för att identifiera proteoformer med användning av antingen direkt fragmentering av proteoformjoner och spektral avläsning av individuell jon MS/MS (överst) eller databasuppslagning av exakta massvärden (IMT, botten). Skalstänger, 1 mm. Kredit:Science Advances (2022). DOI:10.1126/sciadv.abp9929
Utredare ledda av Neil Kelleher, Ph.D., professor i medicin vid avdelningen för hematologi och onkologi och för biokemi och molekylär genetik, har utvecklat en ny avbildningsteknik som ökar detektionen av intakta proteoformer med fyra gånger jämfört med nuvarande proteinavbildningsmetoder .
Bildtekniken, detaljerad i en nyligen publicerad artikel publicerad i Science Advances , tillhandahåller högupplöst bildåtergivning med hög genomströmning av proteoformer eller alla modifierade versioner av proteiner. Viktigt är att tekniken är "etikettfri", kräver inga antikroppar och kan identifiera hela proteoformer direkt från någon ofixerad vävnad. Tekniken kan för närvarande detektera ungefär 1 000 proteoformer och lokaliserar proteoformer med en rumslig upplösning på 40 till 70 mikron.
Flera tekniker används vanligtvis för att avbilda proteiner i mänsklig vävnad, men mycket få är kapabla att avbilda proteoformer. De som kan avbilda hela proteoformer gör det genom att separera proteoformen från vävnad och jonisera dem för masspektrometri. Dessa tekniker erbjuder emellertid låg molekylär specificitet.
För att lösa detta problem utvecklade Kellehers team proteoform imaging masspektrometri (PiMS). Tekniken fungerar genom att extrahera proteoformer från vävnaden med nanodroppar, "väga" de extraherade proteoformerna för att identifiera dem och sedan använda dessa data för att konstruera proteoformbilder av den skannade vävnaden.
"Den verkliga innovationen med PiMS är att den kombinerar en robust befintlig teknik för att extrahera och jonisera proteoformer, nanoDESI, med en banbrytande teknologi för individuell jonmasspektrometri som uppfanns tillsammans av Thermo Fisher Scientific och Northwestern Proteomics. Jämfört med vanliga detektionstekniker, Individuell jonmasspektrometri erbjuder upp till 500 gånger mer känslighet och 20 gånger mer upplösningsförmåga. Det ökar kraften i tekniken avsevärt och PiMS upptäcker större, sällsyntare proteoformer och utvidgar gränserna för proteomtäckning avsevärt, säger Kelleher, som också är chef för Northwesterns Proteomics Center of Excellence, Robert H. Lurie Comprehensive Cancer Centers Proteomics Core Facility och Northwesterns Chemistry of Life Processes Institute.
För att demonstrera PiMS kapacitet använde Kellehers team tekniken för att avbilda proteoformer från funktionella enheter i den mänskliga njuren. Dessa bilder avslöjade distinkta rumsliga lokaliseringar av proteoformer från olika anatomiska regioner och funktionella vävnadsenheter som njurbarken kontra medulla.
PiMS ökade proteomtäckning öppnar också dörren för bredare tillämpningar inom molekylär vävnadskartläggning, identifiering av nya biomarkörer och förbättrar sjukdomsdiagnostik, enligt Kelleher.
"Nyligen har det skett ett stort tryck inom genomik och proteomik för encellig biologi:att bättre fånga sjukdomars heterogenitet genom att använda rumsliga eller encelliga tillvägagångssätt som bevarar de många olika signalerna istället för bulkmetoder som blandar alla celltyper och Det rumsliga tillvägagångssättet i synnerhet tillför en mycket större precision för proteinavbildning och vi driver för närvarande på att identifiera tusentals proteoformer med encellsupplösning, säger Kelleher. + Utforska vidare