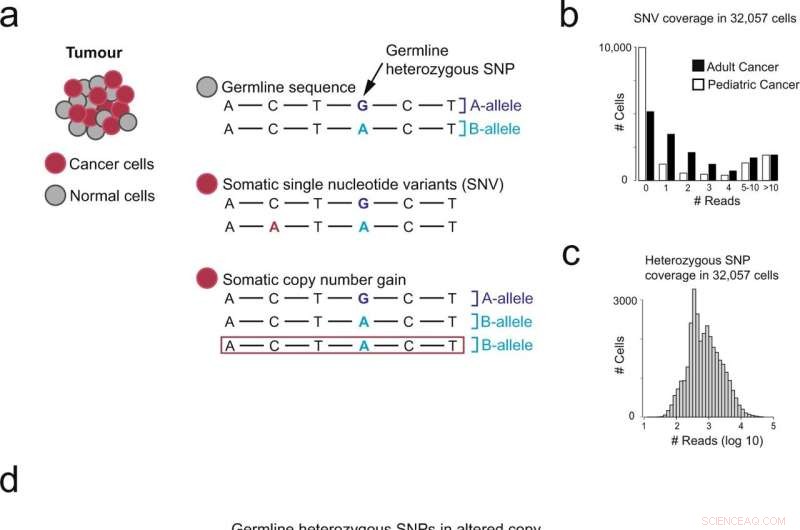
Översikt över olika metoder för att identifiera cancerhärledda celler. a Genomiska förändringar som finns i cancergenom. b Antal celler (y-axel) med N-avläsningar som täcker punktmutationer (x-axel), åtskilda av låg (NB neuroblastom) och hög (RCC njurcellskarcinom) mutationsbörda. c Antal celler (y-axel) med N-avläsningar som täcker heterozygota enkelnukleotidpolymorfismer (SNP) (x-axel). d Översikt över användning av allelskiften som representerar kopienummerändringar för att detektera cancerceller. Kredit:Communications Biology (2022). DOI:10.1038/s42003-022-03808-9. https://www.nature.com/articles/s42003-022-03808-9
En ny metod för att separera cancerceller från icke-cancerceller har utvecklats av forskare vid Wellcome Sanger Institute, i ett uppsving för dem som arbetar för att bättre förstå cancerbiologin genom att använda encellig mRNA-sekvensering.
Studien, publicerad idag i Communications Biology , förbättrar befintliga metoder för att identifiera vilka celler i ett prov som är cancerösa och tillhandahåller avgörande data om tumörernas mikromiljö. Programvaran är öppet tillgänglig för forskare runt om i världen att tillämpa på sina egna data, vilket förbättrar effektiviteten av encellssekvensering för att förstå cancer.
Encellig mRNA-analys av cancerceller är en av de ledande teknikerna som används för att bättre förstå cancerbiologin. Data som genereras kan användas för att försöka störa cancer med droger eller räkna ut hur cancer uppstår i första hand.
Ett grundläggande steg i denna process är att separera cancerceller och icke-cancerceller, men detta är inte alltid en lätt uppgift. Förutom de många typerna av cancer kommer det också att finnas molekylära skillnader mellan cancerceller av samma typ inom en enda tumör.
För närvarande är den bästa metoden att göra detta att mäta det genomsnittliga genuttrycket av celler i provet, med högre eller lägre uttryck som används för att skilja cancerceller från friska celler. Men den här metoden kan leda till falska slutsatser.
I denna nya studie utförde forskare vid Wellcome Sanger Institute helgenomsekvensering och encellig mRNA-sekvensering på prover som samlats in av Great Ormond Street Hospital (GOSH).
Genom att lokalisera obalanser av alleler i dessa data, som indikerar förändringar i antal kopior i genomet, kunde teamet identifiera cancerceller mer tillförlitligt än med tidigare metoder. Detta tillvägagångssätt kommer i första hand att vara användbart för att validera nya cancercelltyper och bättre förstå mikromiljön i tumörvävnad.
"Att kunna veta hur transkriptomet skiljer sig i celler med avvikande genom, som de som finns i cancer, är värdefull kunskap och kommer att utöka de frågor som vi kan svara på med encellssekvensering", säger Dr Matt Young.
Metoden, som heter alleleIntegrator, är tillgänglig som ett mjukvarupaket för forskare över hela världen att använda. + Utforska vidare