Grafiskt abstrakt. Kredit:Cell (2022). DOI:10.1016/j.cell.2022.08.003
Nyckelstudier under det senaste decenniet har visat att tarmmikrobiomet, samlingen av hundratals bakteriearter som lever i det mänskliga matsmältningssystemet, påverkar neural utveckling, svar på cancerimmunterapier och andra hälsoaspekter. Men dessa samhällen är komplexa och utan systematiska sätt att studera beståndsdelarna, de exakta cellerna och molekylerna kopplade till vissa sjukdomar förblir ett mysterium.
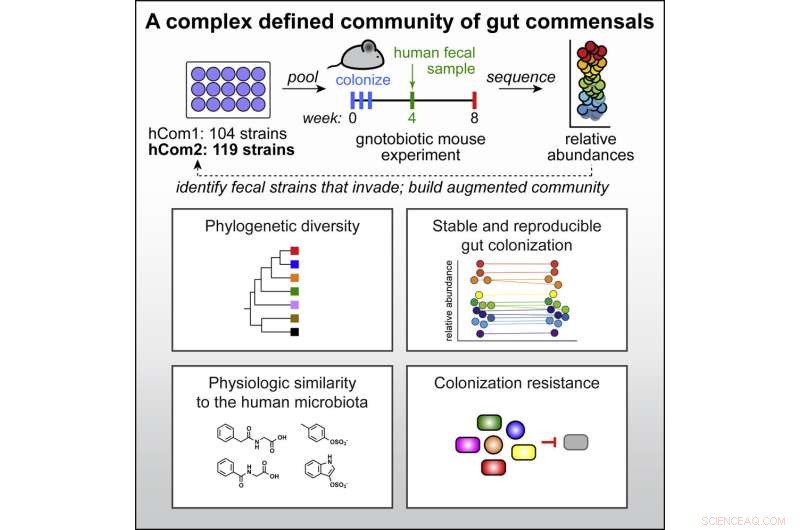
Forskare vid Stanford University har byggt det mest komplexa och väldefinierade syntetiska mikrobiomet och skapat en gemenskap av över 100 bakteriearter som framgångsrikt transplanterats till möss. Möjligheten att lägga till, ta bort och redigera enskilda arter kommer att göra det möjligt för forskare att bättre förstå kopplingarna mellan mikrobiomet och hälsan och så småningom utveckla förstklassiga mikrobiomterapier.
Många viktiga mikrobiomstudier har gjorts med fekala transplantationer, som introducerar hela den naturliga mikrobiomen från en organism till en annan. Medan forskare rutinmässigt tystar en gen eller tar bort ett protein från en specifik cell eller till och med en hel mus, finns det ingen sådan uppsättning verktyg för att ta bort eller modifiera en art bland de hundratals i ett givet fekalt prov.
"Så mycket av det vi vet om biologi, vi skulle inte veta om det inte vore för förmågan att manipulera komplexa biologiska system bitvis", säger Michael Fischbach, Institute Scholar vid Sarafan ChEM-H och motsvarande författare på studien, publicerad i Cell den 6 september.
Fischbach, som är docent i bioteknik och i mikrobiologi och immunologi, och andra såg en lösning:Bygg ett mikrobiom från grunden genom att odla individuellt och sedan blanda dess ingående bakterier.
Bygg arken
Varje cell i mikrobiomet upptar en specifik funktionell nisch och utför reaktioner som bryter ner och bygger upp molekyler. För att bygga ett mikrobiom var teamet tvunget att se till att den slutliga blandningen inte bara var stabil, upprätthöll en balans utan att någon enskild art övermannade resten, utan också funktionell, och utförde alla handlingar av en komplett, naturlig mikrobiom. Att välja arter att inkludera i deras syntetiska samhälle var också svårt med tanke på den naturliga variationen mellan individer; två personer som valts ut slumpmässigt delar mindre än hälften av sina mikrobiella gener.
Forskarna bestämde sig för att bygga sin koloni från de vanligaste bakterierna och vände sig till Human Microbiome Project (HMP), ett initiativ från National Institutes of Health för att sekvensera hela mikrobiella genomen från över 300 vuxna.
"Vi letade efter Noaks ark av bakteriearter i människans tarm, och försökte hitta de som nästan alltid fanns där hos någon individ", sa Fischbach.
De valde ut över 100 bakteriestammar som fanns i minst 20 % av HMP-individerna. Genom att lägga till några arter som behövs för några efterföljande studier kom de till 104 arter, som de odlade i individuella bestånd och sedan blandade till en kombinerad kultur för att göra vad de kallar mänskligt samhälle till ett, eller hCom1.
Även om de var nöjda med att stammarna kunde samexistera i labbet, var det sanna testet om deras nya koloni skulle slå rot i tarmen. De introducerade hCom1 till möss som är noggrant utformade för att inte ha några bakterier närvarande. hCom1 var anmärkningsvärt stabilt, med 98 % av de ingående arterna som koloniserade tarmen hos dessa bakteriefria möss, och de relativa förekomstnivåerna för varje art förblev konstant under två månader.
Utländsk invasion
För att göra sin koloni mer komplett ville forskarna se till att alla vitala mikrobiomfunktioner skulle utföras av en eller flera arter. They relied on a theory called colonization resistance, which explains that any bacterium, when introduced into an existing colony, will only survive if it can fill a niche not already occupied.
By introducing a complete microbiome, in the form of a human fecal sample, to their colony and tracking any new species that took up residence, they could build a more complete community.
Some were skeptical that this would work. "The bacterial species in hCom1 had lived together for only a few weeks," said Fischbach. "Here we were, introducing a community that had coexisted for a decade. Some people thought they would decimate our colony."
Remarkably, hCom1 held its own, and only about 10% of cells in the final community came from the fecal transplant.
They found over 20 new bacterial species that inserted themselves in at least two of their three fecal transplant studies. Adding those to their initial community and removing those that failed to take root in mouse guts gave them a new community of 119 strains, dubbed hCom2. This second iteration, still made from individually growing and then mixing the constituents, made mice even more resistant to fecal challenges than the first.
Final challenge
To demonstrate the utility of their synthetic microbiome, the team took hCom2-colonized mice and challenged them with a sample of E. coli. These mice, like those that were colonized with a natural microbiome, resisted infection.
Prior studies have shown that a healthy natural microbiome leads to protection, but Fischbach and colleagues could take this a step further by iteratively eliminating or modifying certain strains to determine which ones specifically conferred protection. They found several key bacteria and plan to conduct further studies to narrow down to the most critical species.
Fischbach believes that hCom2, or future versions of it, will enable similar reductionist studies that reveal the bacterial agents involved in other areas, like immunotherapy responses.
"We built this consortium for the broader research community. We want to get this into as many hands as possible to have an impact on the field," said Fischbach.
He also envisions that this method of building a microbiome from the ground up will make engineered microbiome-based therapies possible in the future. As the director of the Stanford Microbiome Therapies Initiative (MITI), an initiative launched in 2019 by Sarafan ChEM-H and the Department of Bioengineering, he aims to construct engineered communities that could one day be transplanted into people to treat or prevent a variety of diseases. + Utforska vidare