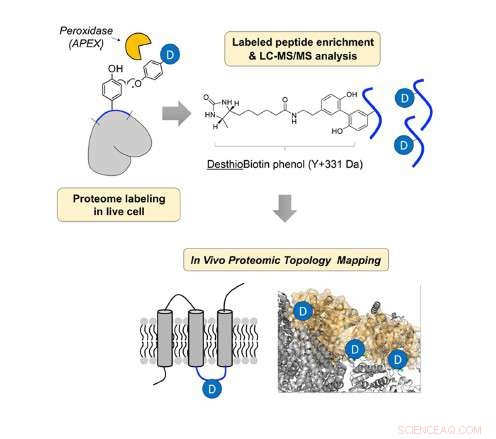
In vivo strukturell identifiering av mitokondrieprotein, använder peroxidas och destiobiotin-fenol. Kredit:UNIST
Membranproteiner utgör ungefär en fjärdedel av alla genprodukter och är måltavlor för över 50 procent av alla moderna läkemedel. Det inre mitokondriella membranet (IMM) proteomet spelar en central roll för att upprätthålla mitokondriell fysiologi och cellulär metabolism. Trots deras betydelse, det har inte funnits någon metod för att avslöja topologin för mitokondriella membranproteiner i levande celler, tills nu.
En nyligen genomförd studie, knuten till UNIST har utvecklat en ny teknik för att förstå de korrekta arkitekturerna för IMM-proteiner, med hjälp av speciella kemiska verktyg. Genom att lösa det svåraste stadiet i utvecklingen av nya läkemedel, deras arbete kommer att hjälpa till att påskynda utvecklingen av nya terapier och botemedel.
Denna forskning har letts av teamet av professor Hyun-Woo Rhee i kemi vid UNIST i samarbete med professor Jong-Seo Kim vid Center från Center for RNA Research, inom Institute for Basic Science (IBS) vid Seoul National University och professor Jeong-Kon Seo vid UNIST Central Research Facilities (UCRF). Resultaten av studien har visats i den 15 mars upplagan av Journal of the American Chemical Society ( JACS ).
IMM är en av de mest aktiva platserna för cellulär metabolism och den är djupt relaterad till olika mänskliga metabola sjukdomar inklusive cancer och neurodegenerativa sjukdomar. Därför, det är avgörande att förstå den korrekta arkitekturen för IMM-proteomet i levande celler för framgångsrik och effektiv utveckling av mitokondrierinriktade terapier.
I studien, Professor Lee och hans forskargrupp avslöjade den in vivo topologiska riktningen för 135 IMM-proteiner, med användning av en in situ-genererad destiobiotin-fenoxyl-radikalsond med genetiskt riktat peroxidas (APEX).
"Bestämning av membranproteinstruktur är en av de mest utmanande uppgifterna i proteinstrukturanalys, " säger professor Lee. "Vår identifiering av strukturell information om mitokondriernas innermembranproteom kan ge värdefulla insikter för arkitekturen och kopplingen av IMM-proteomet i levande celler."
Forskargruppen designade en ny kemisk sond, destiobiotin-fenol och applicerade det på IMM-proteinerna i levande celler. Sedan, de identifierade strukturen av membranproteiner via masspektrometri (MS).
Peroxidas kan reagera med väteperoxid för att göra fenoxylradikalen. Sedan, fenoxylradikalen kan reagera med tyrosinresten på det proximala proteinet och bilda en kovalent bindning. I studien, forskargruppen fick topologiinformationen genom att analysera det märkta tyrosinstället i membranproteinet.
Majoriteten av proteinsekvensanalys idag använder masspektrometri (MS), som smälter proteinprovet till peptider med hjälp av ett lämpligt enzym. Tidigare analyser, som använde genetiskt riktat askorbatperoxidas (APEX), kunde inte lösa strukturell identifiering eftersom dessa analyser baserades på omärkt peptiddetektion. Dock, endast den märkta peptiden kan ge användbar strukturell information, enligt forskargruppen.
Till skillnad från biomolekyler som är märkta med biotin-fenol, proteiner och andra mål som är märkta med destiobiotin-fenol kan elueras utan hårda, denaturerande förhållanden. Dessutom, när antalet tillgängliga membranproteinstrukturprover som erhålls via MS ökar, effektiviteten av strukturell identifiering av membranproteiner ökar också.
På grund av den korta livslängden för fenoxylradikaler som genereras in situ av submitokondriella riktade APEX och IMM:s ogenomtränglighet för små molekyler, de lösningsmedelsexponerade tyrosinresterna på både matris- och intermembranrymden (IMS) sidorna av IMM-proteiner märktes exklusivt med radikalsonden i levande celler av Matrix-APEX och IMS-APEX, och identifieras med masspektrometri.
Genom denna analys, forskargruppen bekräftade 58 IMM-proteintopologier och fastställde den topologiska riktningen för 77 IMM-proteiner vars topologi vid IMM inte helt har karakteriserats.