Kredit:University of Pennsylvania
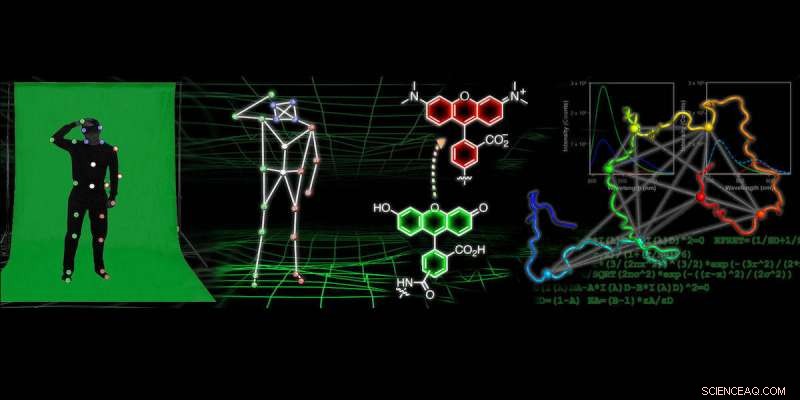
I många moderna animerade filmer, Knepet för att uppnå realistiska rörelser för enskilda karaktärer och objekt ligger i motion-capture-teknik. Denna process innebär ofta att någon bär en träningsdräkt täckt av små, färgade bollar medan en kamera fångar positionen för de färgade bollarna, som sedan används för att representera hur personen rör sig.
Forskare vid University of Pennsylvania utvecklar en liknande teknik för att få "filmer" med atomupplösning som spårar hur proteiner viker sig och ändrar form. För att skapa dessa filmer, forskarna märker proteinet med sonder på många positioner och observerar rörelsen av dessa märken. Fluorescensdata om probernas relativa positioner kan sedan användas för att konstruera beräkningsmodeller av proteinstrukturen i atomär detalj. Denna forskning kan leda till förbättringar av läkemedel som används för att behandla neurodegenerativa sjukdomar, samt nya metoder för avbildning som kan leda till att de upptäcks tidigare.
Forskningen var multidisciplinär ansträngning ledd av E. James Petersson. Tidningen publicerades i Biofysisk tidskrift .
"En av de stora grundläggande frågorna inom biokemi är hur proteiner viker sig till en viss form, sa Petersson, "och detta dikteras av sekvensen av aminosyror i proteinet. Informationen i alla interaktioner mellan aminosyrasidokedjorna leder på något sätt till att den viker sig till en riktig form."
I hälsosamma scenarier, Petersson sa:att rätt form gör att proteinet kan ha olika funktioner, som att transportera syre i blodet eller att bli "molekylära maskiner" som i slutändan leder till muskelrörelser, som de som krävs för att gå och springa.
Men i vissa sjukdomstillstånd, särskilt vid neurodegenerativa sjukdomar som Alzheimers och Parkinsons, proteinerna felveckas till en ohälsosam form, vilket kan få flera kopior av proteinerna att aggregera till "spaghettiliknande tovor eller långa fibriller". Dessa fibriller, Petersson sa:är giftiga för neuroner, som ligger till grund för Alzheimers och Parkinsons sjukdom.
"Eftersom alla dessa involverar proteinveckning till en viss form, sedan vad vi skulle vilja göra är att spåra förändringar i form av proteiner, " sa han. "Det finns ett antal olika tekniker som kan användas för att göra detta, men vi gillar fluorescens eftersom du kan få fluorescensdata tillräckligt snabbt för att du faktiskt kan se proteiner vikas i realtid. I slutändan skulle vi vilja försöka se proteiner som viker sig i celler."
För att få information om proteinform med hjälp av fluorescerande prober, forskare använder en teknik som kallas fluorescensresonansenergiöverföring, vilket kräver att de mäter många avstånd mellan olika punkter på proteinet och sedan använder den informationen för att förstå dess form, liknande motion-capture-teknik.
I det här pappret, forskarna gjorde cirka 30 mätningar av olika avstånd inom proteinet alfa-synuklein under olika tillstånd där det ändrar form. De använde sedan den samlingen av avståndsmätningar i kombination med komplex beräkningsmodellering för att få atomupplösningsstrukturer av proteinets form.
Ferrie fick ett stipendium från Parkinsons Disease Foundation för att tillbringa sommaren i David Bakers laboratorium vid University of Washington, där Rosetta, ett av de mest använda programmen för att modellera proteiner, var utvecklad.
"Rosetta är designad för att modellera stabila välvikta proteiner, sa Petersson, "inte oordnade proteiner som kan ändra form, så Jack fick göra en hel del omskrivning av koden själv för att kunna modellera dessa oregerliga proteiner."
Ferrie och Yoon gjorde en serie experimentella mätningar som kunde användas för att styra proteinveckningen så att den överensstämmer med de experimentella mätningarna, vilket gjorde det möjligt för honom att modellera proteinform.
Eftersom det var ett nytt tillvägagångssätt, forskarna ville visa att strukturerna som kommer ut ur beräkningsmodellerna stämde överens med verkligheten. För att göra detta, de genomförde tre typer av experiment för att matcha verkliga data med modellerna som kommer ut från dessa fluorescensexperiment.
De samarbetade med Rhoades grupp för att validera de modellerade strukturerna med hjälp av enstaka molekylfluorescensmätningar gjorda av Pan. Arbetar i Fakhraiis grupp, Lin, använde en annan typ av teknik som kallas atomkraftsmikroskopi för att avbilda proteinet och validera modellerna. Chris Dobson, professor i kemi vid Cambridge University, delade kärnmagnetisk resonansdata om alfa-synuklein som gav ytterligare bekräftelse på datamodellernas noggrannhet.
Uppsatsen representerar ett av de största biblioteken av proteiner märkta med syntetiska fluoroforer som hittills rapporterats. Enligt Petersson, forskarna behövde göra många avståndsmätningar över olika regioner av proteinet för att ha tillräckligt med data för att generera beräkningsmodeller. Ferrie och Haney var tvungna att komma på en strömlinjeformad metod för att fästa olika uppsättningar av sonder som skulle fungera över olika avståndsområden.
Forskarna arbetar nu med att tillämpa denna teknik för att modellera proteinstruktur i de aggregerade former som är giftiga för neuroner och att modellera dess svar på läkemedel som skulle få det att ändra form, förhindra denna aggregering.
"Förmågan att se ett protein när det ändrar form, sa Petersson, "och att faktiskt få ut strukturer ur det är ett riktigt viktigt grundläggande vetenskapsmål som vi har arbetat mot i 10 år. Det har skett några mycket imponerande genombrott för att få strukturer av proteiner i neurodegenerativa sjukdomar, men fluorescenstekniken har potential att göra det i levande celler, som ingen annan teknik har förmågan att göra."
Enligt Petersson, proteiner som antar flera former, stapla olika kopior på varandra och aggregera, som de gör vid neurodegenerativa sjukdomar, är ett viktigt grundläggande biokemiproblem som andra strukturbiologiska tekniker inte riktigt kan hantera. A better understanding of how that works and what those shapes are has the potential to make an impact on diseases such as Parkinson's and to give researchers an opportunity to figure out how drug or diagnostic models interact with the protein.
"We're working on being able to generate model structures that actually show what is the effect of these drugs, " Petersson said. "We take the protein with the fluorescent labels, add the drug, allow the protein to change shape, make fluorescence measurements and then take those back to the computational modeling so we can actually see the structural effect of these drugs. Hopefully this will lead to more of a rational understanding so that better second and third generation drugs can be made."
The researchers are also collaborating with Robert Mach, the Britton Chance Professor of Radiology in Penn's Perelman School of Medicine. Mach's group is interested in developing positron emission tomography imaging probes that can be used to bind to the aggregated forms of proteins and image them in patients.
"There are some promising drugs for treating neurodegenerative diseases such as Alzheimer's and Parkinson's, that could block this formation of aggregates, " Petersson said, "but the problem is that, by the time people show cognitive or motor-tremor symptoms, it's too late to use these drugs because there's already too much neurodegeneration. If you're getting aggregates in your brain, even if you're not showing any behavioral changes or learning deficits, these probes could noninvasively image the aggregates. By achieving a rational understanding of what the protein structure is, we hope we can help with that work moving forward."