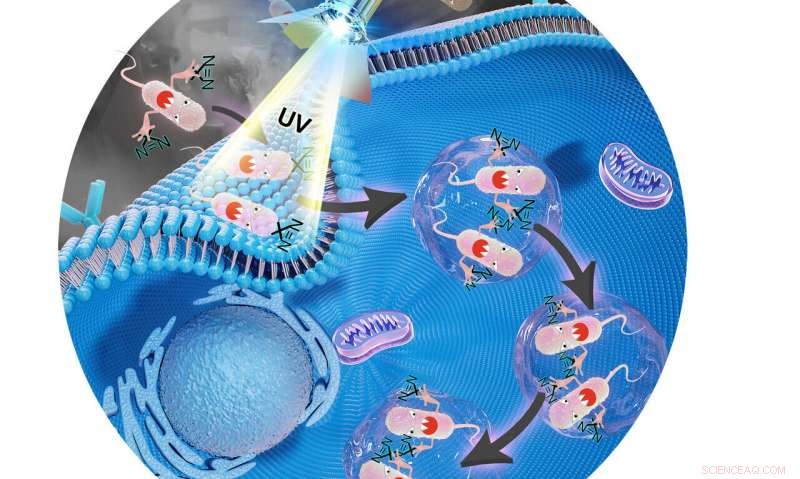
W. Andy Tao och kollegor har utvecklat en metod för att implantera en kemisk etikett som fungerar som en GPS-spårare i levande salmonellabakterier. Väl inne i bakterierna, sonden kan fångas när som helst, visar i realtid proteinerna som interagerar med bakterierna. Kredit:W. Andy Tao
När bakterier som salmonella infekterar och sjuka människor, de kapar en persons cellproteiner för att utveckla ett försvar mot ett immunsvar. Att förstå hur det fungerar och att utveckla metoder för att försvara sig mot dessa bakterier är svårt eftersom forskare inte har kunnat spåra de hundratals proteiner som är involverade i realtid.
Nu, W. Andy Tao, en Purdue University professor i biokemi, och kollegor vid Purdue och Fudan University i Kina, har utvecklat en kemisk metod - värd och patogen temporal interaktionsprofilering, eller HAPTIP – för att märka en levande bakterie och spåra den när den invaderar en värdcell. Deras resultat, publiceras i tidskriften Angewandte Chemie , kan hjälpa till att förbättra förståelsen för bakterieinfektioner och leda till utvecklingen av nya läkemedel.
"Interaktionen mellan värdceller och patogener är mycket dynamiska och komplexa med många frågor som ska besvaras. Det är oerhört värdefullt att ge en dynamisk bild av sådana interaktioner under infektionsprocessen, " skriver författarna. "Det är tänkbart att den allmänna strategin för HAPTIP kan tillämpas på många bakterier eller virus, vilket bidrar till upptäckten och förståelsen av värd-patogen-interaktioner i flera infektionssystem."
Salmonellabakterier avvärjer en cells immunförsvar genom att skapa en ficka i cellen, kallas en salmonella-innehållande vakuol, att gömma sig i. Bakterierna kapar och använder hundratals av cellens proteiner för att göra det, göra identifiering av dessa proteiner nyckeln till att motverka bakterierna.
HAPTIP-metoden går ut på att märka salmonellabakterierna med en diaziringrupp, en kemisk grupp som skapar kovalenta bindningar mellan Salmonellaproteiner och värdcellsproteiner när ett ultraviolett ljus lyser på cellen. En kemisk sond berikar alla tvärbundna proteiner och isolerar dem från de andra cellextrakten. Forskare kan sedan använda masspektrometri för att identifiera proteinerna.
En av metodens styrkor är att den kan fungera när som helst efter att salmonella har introducerats i den friska cellen. I sina fynd, forskarna testade metoden efter 15 minuter, en timme och sex timmar efter att salmonella infekterade en cell och identifierade mer än 400 proteiner som interagerar med salmonellabakterierna.
"Du kan designa vilken tidpunkt som helst baserat på när du väljer att lysa UV-ljuset på cellerna, " Sa Tao. "Genom att titta på vilka proteiner som interagerar med bakterierna vid de olika tidpunkterna, vi kan bestämma metoden som bakterierna använder för att kapa cellen, som kommer att skilja sig åt med tiden."
Att utveckla strategier för att behandla livsmedelsburna sjukdomar som härrör från bakterier som salmonella och E. coli kan få betydande inverkan globalt. Världshälsoorganisationen uppskattar att det finns 600 miljoner globala fall av livsmedelsburna sjukdomar varje år, vilket resulterar i 420, 000 döda.