Forskare med anknytning till det internationella mjukvaruprojektet MZmine, som leds av Dr. Robin Schmid och Dr. Tomáš Pluskal från Institute of Organic Chemistry and Biochemistry vid den tjeckiska vetenskapsakademin, har kommit fram till en ny mjukvara som avsevärt snabbar upp och förenklar identifiering av kemikalier i vävnader.
Det gör det möjligt för forskare att känna igen kemikalier och visualisera deras distribution i organ. Jämfört med etablerade arbetsflöden kräver den nya pipelinen mindre arbete i laboratoriet för att få liknande insikter om den kemi som sker inom till exempel tumörer och inflammatoriska lesioner. Artikeln som presenterar denna programvara för det globala forskarsamhället har just publicerats i tidskriften Nature Communications .
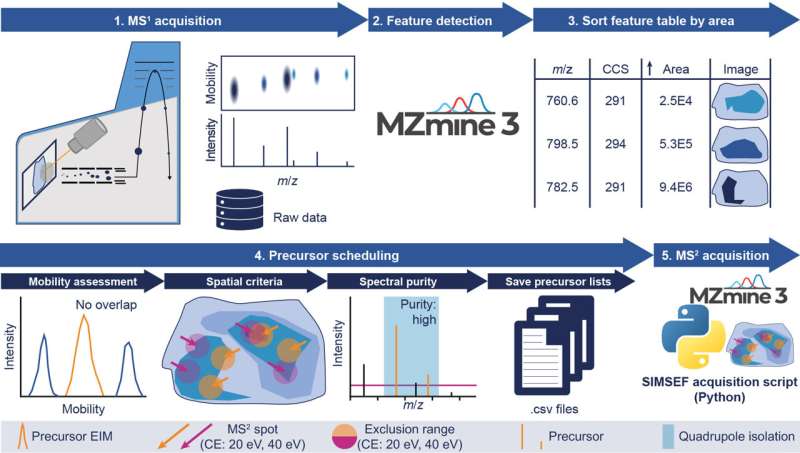
På IOCB Prag är Dr. Robin Schmid mest involverad i att utveckla programvaran SIMSEF (spatial ion mobility-scheduled exhaustive fragmentation) tillsammans med sina kollegor från gruppen av Prof. Dr. Karst vid universitetet i Münster, Tyskland, där han tidigare var baserad.
Arbetet görs i samarbete med utvecklarna av timsTOF fleX state-of-the-art masspektrometer från Bruker Daltonics, som möjliggör belysning på hög nivå av sammansättningen av molekyler genom att mäta jonernas rörlighet.
"Hittills har forskare kunnat säga med en masspektrometer att detta var formeln för molekylen de tittade på, men när de sedan tittade på databasen och försökte identifiera detta ämne var det väldigt svårt. Anledningen är att biologiska prover kan innehålla många distinkta lipider och kombinationer av molekyler som ofta är biologiskt mycket olika."
"Nu, tack vare den nya algoritmen, är det möjligt att titta inuti molekylen, lära sig vad den består av och till och med jämföra bilder med varandra", förklarar Robin Schmid. Detta kan ge viktig information, till exempel att en specifik del av hjärnan innehåller en annan typ av lipid än en annan del av detta väsentliga organ och att just denna lipid saknas i annan vävnad.
SIMSEF-algoritmen utvecklades i samarbete med medicinska experter från tyska och schweiziska universitet. Av avgörande betydelse för läkare är upptäckten av kliniska biomarkörer för diagnostiska syften också betydligt snabbare. Att lära sig mycket snabbt, till exempel om de har att göra med en lipid eller metabolit som bara finns i cancervävnad, spelar en avgörande roll för att besluta om fortsatt behandling. Det behöver inte sägas att detta har stora konsekvenser för resultatet.
Den nya algoritmen är en del av mjukvarupaketet MZmine med öppen källkod, som har hjälpt experter över hela världen att analysera masspektrometridata sedan 2005. Steffen Heuckeroth från MZmine-teamet och Institute of Inorganic and Analytical Chemistry vid University of Münster har varit huvudutvecklare av SIMSEF-algoritmen och är den första författaren till artikeln som nu publicerats i Nature Communications .
Han tillägger, "Artikeln i fråga innehåller ingen specifik upptäckt inom biologin. Men tack vare vår nya metod kan många forskare förbättra och påskynda sitt arbete, vilket öppnar utrymme för nya upptäckter."
"Jag tycker alltid att det är fantastiskt när vi gör det möjligt för andra människor att förbättra arbetsvillkoren för sin forskning och även att kommunicera det till andra forskare. Tack vare feedbacken från forskarsamhället som använder MZmine driver själva programmet direkt deras forskning framåt", tillägger Robin Schmid från IOCB Prag.
Direktören för IOCB Prag Prof. Jan Konvalinka påpekar, "Alla pratar om maskininlärning och artificiell intelligens nuförtiden. Våra kollegor från Dr. Pluskals team använder det för att lösa mycket specifika problem inom forskning såväl som i klinisk praxis."
Den tredje generationen av MZmine, rapporterad våren 2023 i tidskriften Nature Biotechnology , kan behandla tusentals prover per timme. Algoritmerna som nyligen utvecklats av detta internationella team förbättrar därmed befintliga möjligheter att länka olika typer av data, inklusive att kombinera data från analys- och bildbehandlingsmetoder, vilket hittills inte har varit möjligt med någon akademisk eller kommersiell programvara.
Mer information: Steffen Heuckeroth et al, On-tissue dataset-beroende MALDI-TIMS-MS2 bioimaging, Nature Communications (2023). DOI:10.1038/s41467-023-43298-9
Journalinformation: Nature Communications , Naturbioteknik
Tillhandahålls av Institutet för organisk kemi och biokemi vid Tjeckiska vetenskapsakademin (IOCB Prag)