
Teleskoparkitektur:jobbansvarig samlar in jobbinformation genom att ansluta till målklustret via sin anslutningshanterare. Telescopes lokala databas håller register över denna information, som renderas av användargränssnittet till en (mobilvänlig) webbsida. Kredit:Brito et al.
Ett team av forskare vid UCLA, universitetet i São Paulo, Federal University of São Carlos och University of Southern California har nyligen utvecklat ett interaktivt verktyg för att hantera storskaliga bioinformatiska analyser i realtid och från bärbara enheter. Detta nya verktyg, kallas Teleskop, presenterades först i en tidning som förpublicerades den arXiv .
Teamet som utvecklade Telescope inkluderar postdoktorer från UCLA Collaboratory, ett forskningslabb vid universitetets Institute for Quantitative and Computational Biosciences. Att arbeta på UCLA Collaboratory tillåter dessa forskare att interagera med andra studenter och fakultetsmedlemmar vid universitetet som har olika nivåer av expertis inom beräkningsbiologi. Dessa interaktioner är det som i slutändan inspirerade teamet att utveckla ett användarvänligt verktyg för att hantera storskaliga bioinformatiska analyser.
"De flesta biovetenskapliga och biomedicinska studier riktar sig idag mot effekter så subtila och svåra att upptäcka att stora mängder data och beräkningskraft har blivit det nya status quo, " berättade forskarna för TechXplore via e-post. "Ofta, dock, Forskare beskriver det överdrivna beroendet av kommandoradsverktyg som besvärligt och mycket ointuitivt för dem som saknar formell beräkningsutbildning."
Specialiserade utbildningsinitiativ och workshops har visat sig vara särskilt effektiva för att uppmuntra forskare att börja använda kraftfulla metoder för storskaliga bioinformatiska analyser. Dock, de flesta tekniker för att komma åt dessa metoder på beräkningsanläggningar är inte särskilt väl integrerade med våta labbmiljöer, där de flesta biologiska experiment utförs. Med detta i åtanke, forskarna ville utveckla ett verktyg som skulle möjliggöra bättre integration mellan bioinformatikverktyg och våtlabbforskning.
"Teleskop föreslogs för att främja integration och närmare samarbete mellan våtlabbforskning och bioinformatik, minska inlärningskurvan som traditionellt krävs för att köra komplexa bioinformatiska analyser i beräkningsanläggningar, " berättade teamet för TechXplore. "Utmaningen, dock, garanterade att den slutliga lösningen verkligen skulle ta itu med luckorna och vara intuitiv för de flesta forskare."
För att presentera sin lösning för både tekniskt kunniga forskare och de med begränsade beräkningsfärdigheter, forskarna beslutade att introducera Telescope under II Hackathon-evenemanget i början av 2018. Hackathon samlar forskare med IT- och kodningskunskaper, erbjuda dem möjlighet att lära sig IT-kunskaper som grupp, till exempel, utforska hur man bygger skript och tillämpar bioinformatikverktyg på datauppsättningar.
"Vår idé lockade både nybörjare som ville smutsa ner händerna och arbeta direkt med data så snabbt som möjligt och teknikkunniga som bara ville ha ett enklare sätt att kontrollera sitt arbete från sina telefoner, ", förklarade forskarna. "Teleskopet designades alltså från och för samhället."
De flesta forskare som utför experiment i våta labbmiljöer behöver möta betydande tidskrav. Telescope är designat för att hjälpa dem att hantera sin arbetsbelastning bättre, övervaka och justera bioinformatikanalyser i realtid.
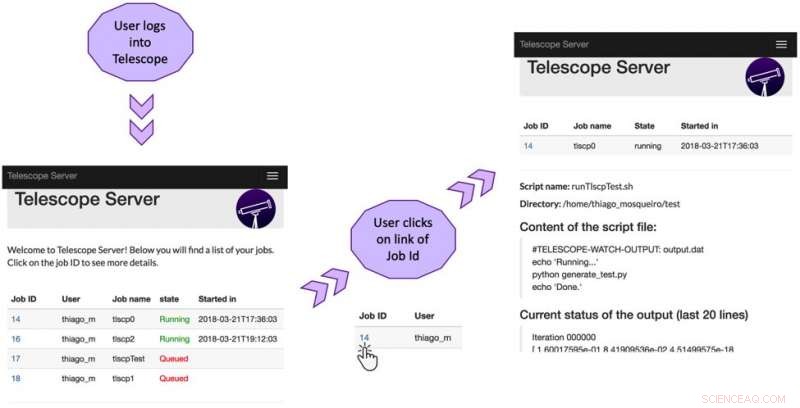
Teleskopanvändargränssnitt:Den första skärmen visar status för jobben i klustret. Nästa skärm visar detaljerad information om det först listade jobbet:källkatalog, namn och innehåll i skriptfilen, och sista raderna av aktuell uppgiftsutgång. Kredit:Brito et al.
Väsentligen, Telescope stärker relationen mellan våtlabbexperiment och verktyg för att analysera stora mängder biologisk data. Den gör detta genom att tillhandahålla en säker och användarvänlig plattform där användare kan kickstarta sin forskning och hantera uppgifter som är relevanta för bioinformatikjobb som körs i högpresterande kluster.
"Telescope använder vanliga webbramverk som Twitters Bootstrap för att ge en ögongodis användarupplevelse och stödja ett utbyggbart gränssnitt, ", sa forskarna. "Att stödja både nya och erfarna användare, Telescopes kärna hanterar och lagrar användaråtkomstnycklar med hjälp av industristandarder."
Åtkomstnycklarna som lagras av Telescope används för att ansluta högpresterande beräkningskluster genom ett säkert skal för att ta emot de senaste jobbstatusuppdateringarna och utfärda nya kommandon när det behövs (t.ex. radera gamla uppgifter etc.). Telescope gör det möjligt för forskare i våtlabb att spåra status för sitt arbete och enkelt få tillgång till preliminära resultat av sina bioinformatiska analyser direkt på sina mobiltelefoner. Det innebär att de också tidigt kan identifiera potentiella problem med sina analyser och avbryta dem utan att behöva använda en dator.
"Jämfört med den vanliga metoden att använda kommandorader på Linux-terminaler, Telescope är ett mer intuitivt och interaktivt verktyg, " förklarade forskarna. "Till exempel, att dela analysresultat är lika enkelt som att dela en länk, som du skulle göra på Twitter eller Facebook. Ändå, vi känner att vår mest meningsfulla prestation var engagemanget med den traditionellt icke-beräkningsbaserade life science-gemenskapen i början av utvecklingen."
Detta nya verktyg för att hantera bioinformatikanalyser tog biologiforskarnas behov i beaktande. Faktiskt, teamet utnyttjade Hackathon-evenemanget för att samla in input och feedback från biologiexperter med olika grader av IT-kompetens och erfarenhet av att använda beräkningsanalysverktyg.
"Vi samlade in input från nya datoranvändare, eftersom vi ville skapa ett verktyg som tar itu med de mest utmanande punkterna med att använda ointuitiva kommandorader, " sa forskarna. "Vi utvecklade sedan ett enkelt och minimalt webbgränssnitt som kan köras på olika enheter, inklusive mobiltelefoner, med noll inställning. Utmaningen var att byta ut enkelhet med säkerhet, och vi uppnådde en bra balans genom att använda industristandardprotokoll, som att lagra nycklar med PBDKS."
Verktyget som utvecklats av denna grupp forskare, som nu är tillgänglig på GitHub, kan hjälpa biologiexperter i deras arbete, hjälpa dem att hantera sina bioinformatiska analyser på ett enklare och mer intuitivt sätt. Förutom att tillåta biomedicinska forskare att få tillgång till kraften i stora beräkningsanläggningar från sina bärbara enheter, Telescope är en anpassningsbar och utbyggbar programvara. Detta innebär att användare kan lägga till nya funktioner till den eller kan hjälpa till att utveckla den ytterligare.
"Vårt projekt kommer nu att underhållas av Mangul Lab vid USC som programvara med öppen källkod, ", sa forskarna. "Vi planerar också att integrera Telescope i de flesta av vårt labbs analyspipelines för att utvärdera användarupplevelsen ytterligare. Som nästa steg, dessa tester kommer att utökas till en omröstning av betatestare från ett bredare samhälle. Our overarching goal is to roll out Telescope for the users of USC's high-performance cluster in the future. Throughout this process, the Mangul lab will thrive to engage with the community to drive adoption and ensure that Telescope remains relevant."
© 2019 Science X Network