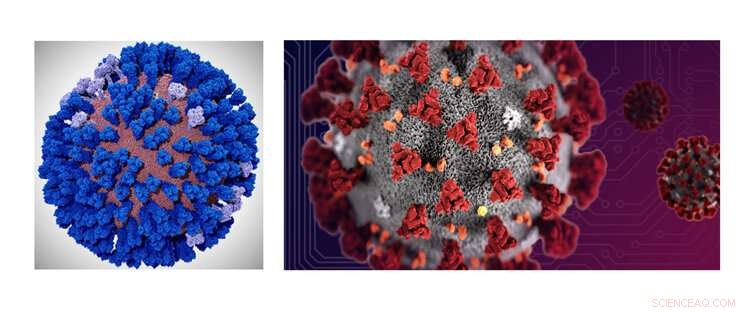
En coronavirus-kuvert-atom-datormodell utvecklas av Amaro Lab i UC San Diego på den NSF-finansierade Frontera-superdatorn för TACC vid UT Austin. Biokemisten Rommie Amaro hoppas kunna bygga vidare på sin senaste framgång med all-atom i? Uenza-virussimuleringar (vänster) och tillämpa dem på coronaviruset (höger). Upphovsman:Lorenzo Casalino (UCSD), TACC
Forskare förbereder en massiv datormodell av coronaviruset som de förväntar sig kommer att ge insikt om hur det smittar i kroppen. De har tagit de första stegen, testa de första delarna av modellen och optimera koden på Frontera -superdatorn vid University of Texas vid Austin's Texas Advanced Computing Center (TACC). Kunskapen från hela modellen kan hjälpa forskare att utforma nya läkemedel och vacciner för att bekämpa coronaviruset.
Rommie Amaro leder ansträngningar för att bygga den första kompletta all-atom-modellen av SARS-COV-2 coronavirushöljet, dess yttre komponent. "Om vi har en bra modell för hur partikelns utsida ser ut och hur den beter sig, Vi kommer att få en bra bild av de olika komponenterna som är involverade i molekylär igenkänning. "Molekylär igenkänning innebär hur viruset interagerar med angiotensinomvandlande enzym 2 (ACE2) -receptorer och eventuellt andra mål i värdcellmembranet. Amaro är professor i kemi och biokemi vid University of California, San Diego.
Coronamodellen förväntas av Amaro innehålla ungefär 200 miljoner atomer, ett skrämmande företag, eftersom interaktionen mellan varje atom med varandra måste beräknas. Hennes teams arbetsflöde tar en hybrid, eller integrerande modelleringsmetod.
"Vi försöker kombinera data i olika upplösningar till en sammanhängande modell som kan simuleras på ledningsanläggningar som Frontera, "Amaro sa." Vi börjar i princip med de enskilda komponenterna, där deras strukturer har lösts med atom- eller nära atomupplösning. Vi får försiktigt igång var och en av dessa komponenter och till ett tillstånd där de är stabila. Sedan kan vi introducera dem i de större kuvertsimuleringarna med angränsande molekyler. "
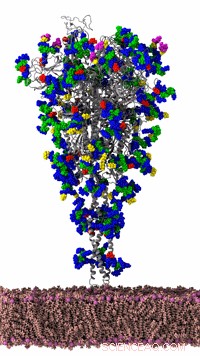
SARS-CoV-2 spikprotein av coronaviruset simulerades av Romaro Lab i UC San Diego på den NSF-finansierade Frontera-superdatorn för TACC vid UT Austin. Det är det huvudsakliga virala proteinet som är involverat i värdcell-coronavirusinfektion. Fysikbaserade molekylära dynamiksimuleringar kan förutsäga hur coronavirusets molekylära maskin rör sig, som ger forskare insikt i dess sårbarhet för potentiella vacciner och läkemedel. Coronavirus Spike -huvud baserat på pdb 6vsb; spikstjälk med hjälp av homologimodellering; glykoprofil utformad enligt Walls et al. 2019 Cell, och Watanabe et al. 2020 bioRxiv. Upphovsman:Rommie Amaro, UCSD
Den 12-13 mars, 2020, Amaro Lab körde molekylära dynamiksimuleringar på upp till 4, 000 noder, eller cirka 250, 000 bearbetningskärnor, på Frontera. Frontera, världens #5 bästa superdator och #1 akademiska superdator enligt rankningen av Top500 -organisationen i november 2019, är högpresterande datorsystem i ledarklass som stöds av National Science Foundation.
"Simuleringar av den storleken är bara möjliga att köra på en maskin som Frontera eller på en maskin möjligen på energidepartementet, "Sade Amaro." Vi kontaktade direkt Frontera -teamet, och de har varit mycket nådiga med att ge oss prioritetsstatus för benchmarking och försöka optimera koden så att dessa simuleringar kan köras så effektivt som möjligt, när systemet faktiskt är igång. "
"Det är spännande att arbeta med en av dessa helt nya maskiner, Säkert. Vår erfarenhet hittills har varit mycket bra. De första riktmärkena har varit riktigt imponerande för detta system. Vi kommer att fortsätta att optimera koderna för dessa extremt stora system så att vi i slutändan kan få ännu bättre prestanda. Jag skulle säga att arbetet med teamet på Frontera också har varit fantastiskt. De är redo att hjälpa till och har varit extremt lyhörda under detta kritiska tidsfönster. Det har varit en mycket positiv upplevelse, "Sade Amaro.
"TACC är stolta över att stödja denna kritiska och banbrytande forskning, "sa Dan Stanzione, Verkställande direktör för TACC och huvudutredare för Frontera -superdatorprojektet. "Vi kommer att fortsätta att stödja Amaros simuleringar och annat viktigt arbete relaterat till att förstå och hitta ett sätt att besegra detta nya hot."

Den NSF-finansierade Frontera-superdatorn från Texas Advanced Computing Center i UT Austin är rankad som #5 snabbast i världen och #1 för akademiska system, enligt topplistorna i november 2019. Upphovsman:TACC
Amaros arbete med coronaviruset bygger på hennes framgång med en all-atom-simulering av influensavirushöljet, publicerad i ACS Central Science , Februari 2020. Hon sa att influensaarbetet kommer att ha ett anmärkningsvärt antal likheter med vad de nu bedriver med coronaviruset.
"Det är ett strålande test av våra metoder och våra förmågor att anpassa sig till ny data och för att få detta igång direkt, "Amaro sa." Det tog oss ett år eller mer att bygga influensavirushöljet och få det igång på de nationella superdatorer. För influensa, vi använde Blue Waters superdator, som på något sätt var föregångaren till Frontera. Arbetet, dock, med coronaviruset går uppenbarligen mycket, mycket snabbare tempo. Detta är aktiverat, delvis på grund av det arbete som vi gjorde på Blue Waters tidigare. "
Sa Amaro:"Dessa simuleringar kommer att ge oss ny inblick i de olika delarna av coronaviruset som krävs för smittsamhet. Och varför vi bryr oss om det är för att om vi kan förstå dessa olika funktioner, forskare har en bättre chans att designa nya läkemedel; för att förstå hur nuvarande läkemedel fungerar och potentiella läkemedelskombinationer fungerar. Den information som vi får från dessa simuleringar är mångfacetterad och mångdimensionell och kommer att vara till nytta för forskare på frontlinjerna omedelbart och även på längre sikt. Förhoppningsvis kommer allmänheten att förstå att det finns många olika komponenter och aspekter av vetenskapen att driva fram för att förstå detta virus. Dessa simuleringar på Frontera är bara en av dessa komponenter, men förhoppningsvis en viktig och en vinstrik. "