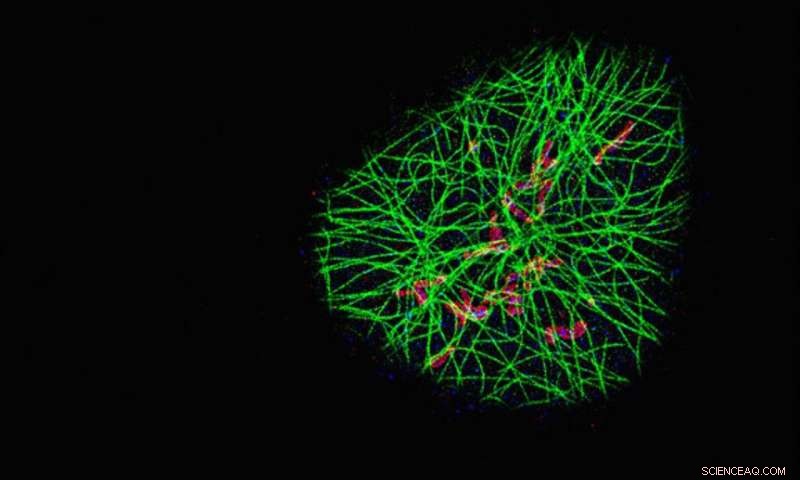
Forskarna använde sin SDC-PAINT-metod för att visualisera nätverket av cytoskeletala mikrotubuli-filament (grönt) och deras närhet med ytterligare två proteiner som kallas TOM20 (röd) och HSP60 (blå). Varje bild visar proteinerna i ett annat plan i cellen från toppen, och de förstorade bilderna på undersidan jämför upplösningen med SDC-PAINT (vänster) med den som är möjlig med konventionell konfokalmikroskopi (höger). Upphovsman:Florian Schueder, MPI/LMU
Cellbiologer använder traditionellt fluorescerande färgämnen för att märka och visualisera celler och molekylerna inom dem under ett mikroskop. Med olika superupplösta mikroskopimetoder, de kan till och med lysa upp enskilda molekyler och deras komplexa interaktioner med varandra. Dock, mikroskopimaskinvaran som gör att de kan göra detta är högspecialiserad och dyr och därför relativt sällsynt i laboratorier runt om i världen, och driften av sådana mikroskop är skrämmande, eftersom det kräver unika färdigheter.
Ralf Jungmann, Ph.D., en alumn vid Harvards Wyss Institute for Biologically Inspired Engineering och för närvarande professor vid Ludwig Maximilian University (LMU) och Max Planck Institute (MPI) of Biochemistry i Tyskland och Wyss Institute Core Fakultetsmedlem Peng Yin, Ph.D., har utvecklat DNA-PAINT, en kraftfull molekylär bildteknik som involverar övergående DNA-DNA-interaktioner för att exakt lokalisera fluorescerande färgämnen med superupplösning. Dock, även om forskarna har visat DNA-PAINTs potential genom att visualisera enstaka biomolekyler, såsom proteiner, i fasta celler på ett fast nära avstånd, tekniken kunde ännu inte tillämpas för att undersöka molekyler djupt inuti cellerna.
Nu, Jungmanns och Yins team rapporterar gemensamt en lösning för att övervinna denna begränsning. I deras nya studie, de anpassade DNA-PAINT-tekniken till mikroskop som är utbredda bland cellbiologilaboratorier, kallas konfokala mikroskop, och som används av forskare för att avbilda hela celler och tjockare vävnader vid lägre upplösning. MPI/Wyss Institute -teamet visar att metoden kan visualisera en mängd olika molekyler, inklusive kombinationer av olika proteiner, RNA och DNA genom hela djupet av hela celler vid superupplösning. Publicerad i Naturkommunikation , tillvägagångssättet kan öppna dörren för detaljerade enkelmolekylära lokaliseringsstudier inom många områden inom cellforskning.
DNA-PAINT-tillvägagångssättet fäster en DNA "ankarsträng till molekylen av intresse. Därefter fäster en färgmärkt DNA" imager-sträng "med en komplementär sekvens övergående till ankaren och producerar en fluorescerande signal, som inträffar som en definierad blinkande händelse vid enstaka molekylära platser. Eftersom "blinkande" är exakt inställbar, molekyler som bara är nanometer från varandra kan särskiljas-vid den högre upplösningsänden av superupplösningen.
"Vår nya strategi, SDC-FÄRG, integrerar de mångsidiga superupplösningsfunktionerna hos DNA-PAINT med de optiska snittfunktionerna i konfokala mikroskop. Vi skapade alltså medel för att utforska hela cellens djup, och för att visualisera molekylerna i den i nanometerskala, "sa Jungmann. Teamet kartlade förekomsten av olika kombinationer av proteiner i hela celler och gick sedan utöver det." Genom att diversifiera våra märkningsmetoder, vi visualiserade också olika typer av enskilda biomolekyler i den kromosomhaltiga kärnan, inklusive sekvenser i DNA, proteiner bundna till DNA eller membranet som omger kärnan, liksom kärn -RNA, "tillägger Yin, som också är ledare för Wyss Institute's Molecular Robotics Initiative, och professor i systembiologi vid Harvard Medical School. .
I princip, Konfokalmikroskop använder så kallade hål för att eliminera oönskad fluorescens som inte är i fokus från bildplan ovanför och under fokalplanet. Genom att skanna igenom provet, plan efter plan, forskare kan samla fluorescenssignalerna från molekylbundna färgämnen över hela djupet. Specifikt, MPI/Wyss Institute -teamet utvecklade tekniken för "Spinning Disk Confocal" (SDC) mikroskop som detekterar fluorescenssignaler från ett helt plan på en gång genom att känna av dem genom en roterande skiva med flera hål. Dessutom, "för att uppnå 3D-superupplösning, vi placerade en extra lins i detekteringsvägen, vilket gör att vi kan arkivera sub-diffraktionsbegränsad upplösning i den tredje dimensionen ", säger författaren Florian Schueder, en doktorand som arbetar med Jungmann som också arbetade med Yins Wyss Institute -team som en del av sin masteruppsats.
"Detta tillägg kan enkelt anpassas av tillverkare av SDC-mikroskop, så vi implementerar i princip superupplösande mikroskopi utan komplexa hårdvaruändringar i mikroskop som är allmänt tillgängliga för cellbiologer från alla biomedicinska forskningsplatser. Tillvägagångssättet har således potential att demokratisera super -upplösningsavbildning genom hela celler och vävnader, sa Jungmann.
"Med detta viktiga framsteg, superupplöst mikroskopi och DNA-PAINT kan bli mer tillgängligt för biomedicinska forskare, påskynda vår insikt om enskilda molekylers funktion och de processer som de kontrollerar i celler, "sade Wyss Institute grundande direktör Donald Ingber, M.D., Ph.D., som också är Judah Folkman professor i vaskulär biologi vid HMS och Vascular Biology Program på Boston Children's Hospital, samt professor i bioingenjör vid SEAS.