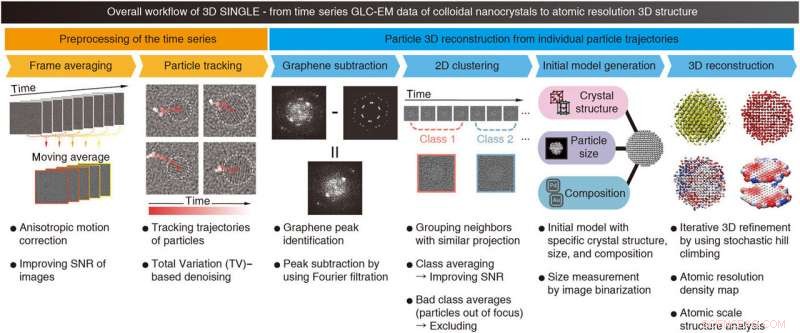
Övergripande arbetsflöde för SINGLE. Övergripande arbetsflöde och beskrivningar av varje steg i SINGLE visas. SINGLE består av två huvudsteg:Förbearbetning av tidsserien (orange), inklusive (i) medelvärdesberäkning av tidsfönster med anisotrop rörelsekorrigering och (ii) spårning av partikelbana med användning av total variation (TV)-baserad avbrusning, och partikel 3D-rekonstruktion från individuella partikelbanor (blå), inklusive (i) identifiering och subtraktion av grafenbakgrund, (ii) tidsbegränsad 2D-klustring med uteslutning av bilder som inte är i fokus, (iii) inledande modellgenerering, och (iv) 3D-rekonstruktion och strukturanalys i atomskala. Kredit:Science Advances, doi:10.1126/sciadv.abe6679
Materialforskare använder vanligtvis lösningsfastransmissionselektronmikroskopi (TEM) för att avslöja de unika fysiokemiska egenskaperna hos tredimensionella (3-D) strukturer hos nanokristaller. I en ny rapport om Vetenskapens framsteg , Cyril F. Reboul och ett forskarlag vid Monash University, Australien, Seouls nationella universitet, Sydkorea, och Lawrence Berkeley National Laboratory U.S., utvecklat en Brownsk 3D-rekonstruktionsmetod med en partikel. För att åstadkomma detta, de avbildade ensembler av kolloidala nanokristaller med hjälp av grafen-vätskecelltransmissionselektronmikroskopi. Teamet fick projektionsbilder av olika roterade nanokristaller med hjälp av en direkt elektrondetektor för att få en ensemble av 3D-rekonstruktioner. I det här arbetet, de introducerade beräkningsmetoder för att framgångsrikt rekonstruera 3D nanokristaller vid atomupplösning och åstadkom detta genom att spåra individuella partiklar genom tiden, samtidigt som den störande bakgrunden subtraheras. Metoden kan också identifiera/avvisa bilder av låg kvalitet för att underlätta skräddarsydda strategier för 2-D/3-D anpassning som skilde sig från dem i biologisk kryo-elektronmikroskopi. Teamet gjorde utvecklingen tillgänglig genom ett mjukvarupaket med öppen källkod som kallas SINGLE. Den kostnadsfria programvaran finns tillgänglig på GitHub.
Använder SINGLE för kristallografi
Forskare har upprätthållit framsteg inom kristallografi under de senaste 50 åren för att förändra den befintliga förståelsen av kemi och biologi. Ändå, vissa mål inklusive solubiliserade nanokristaller förblir svårhanterliga för traditionella kristallografiska metoder. Till exempel, kolloidala nanokristaller innehåller tiotals till hundratals atomer och har en mängd olika tillämpningar inom tvärvetenskapliga områden inklusive elektronik, katalys och biologiska sensorer. Mångsidigheten härrör från den höga känsligheten hos nanokristallegenskaper för storlek, kemisk sammansättning och andra variabler under syntesen. Vanligtvis, forskare använder en partikel, 3D-rekonstruktion i strukturbiologi för att bestämma strukturen av proteiner. Tekniken är relativt ny för in-situ 3D-rekonstruktion av solubiliserade individuella nanokristaller. I det här arbetet, Reboul et al. utvecklat ENKEL; en metod som förlitade sig på den oberoende 3D-rekonstruktionen av solubiliserade individuella nanokristaller inklusive Brownsk rörelse. Tekniken är ett första framsteg i studien för att lösa 3D-atomstrukturer av nanokristaller direkt från lösningsfasen.
Spårning av individuella nanokristallbanor. Spåra resultat av partikel 1 (A och B) och 2 (C och D) genom hela filmen (blått till rött). Representativa tidsmedelvärden för råa ojusterade partikelbilder (50 bildrutor) visas (B och D). Skala staplar, 1 nm. Kredit:Science Advances, doi:10.1126/sciadv.abe6679 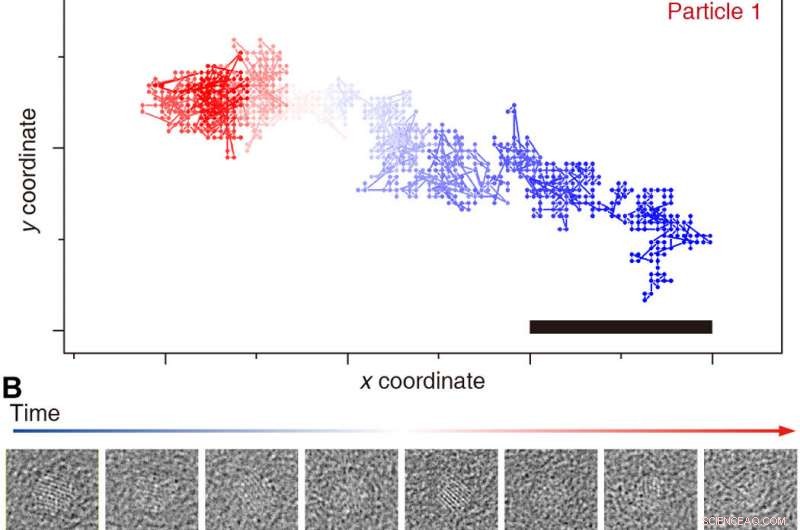
Forskarna introducerade nya förbehandlingsmetoder för att förbättra signal-till-brus-förhållandet (SNR) för att spåra partikelbanorna samtidigt som grafeninducerad bakgrundssignal tas bort. Avancerade beräkningsmetoder skulle framgångsrikt kunna rekonstruera 3D från in-situ grafen flytande cell (GLC) transmissionselektronmikroskopdata. Jämfört med befintliga tekniker, arbetet presenterade användbarheten av en aldrig tidigare skådad beräkningsmetod för att erhålla 3D-rekonstruktioner vid atomupplösning för nanokristaller dispergerade i lösning. De delade in ENKEL arbetsflöde i två huvudsteg (1) förbearbetning och (2) partikel 3D-rekonstruktion. Forskarna strävade efter att ge högsta möjliga prestanda och effektivitet på alla CPU-hårdvara, inklusive superdatorer till arbetsstationer eller till och med bärbara datorer.
I början, teamet tog ett genomsnitt av tidsfönstret över flera bildrutor med anisotropisk rörelse för att förbättra signal-brusförhållandet, vilket leder till synliga partiklar och en förbättrad grafensignal. Teamet identifierade sedan partikelpositionerna manuellt i det första tidsfönstrets genomsnitt. Därefter, teamet utvecklade en startmodell baserad på den förväntade kristallografiska strukturen, partikeldiameter och ingående element och producerade 3D-rekonstruktioner med inpassade atomkoordinater för strukturanalys i atomär skala.
Tidsbegränsad 2D-klustring. (A) Bråkdel av vinkelförändring genom tidsserien. Röd streckad linje är trendlinjen. Projektionsriktningarna ändras snabbt i områdena mellan ramar 1500 till 1600 (orange), ramar 3800 till 3900 (grön), och ramar 5600 till 5700 (blå). Insättningar är diagram som visar vinkelskillnad i projektionsriktning i dessa regioner. (B) Schematisk skildring av tidsbegränsad 2D-klassmedelvärde. (C) Plot som visar tilldelade klasser för individuella ramar i 1 till 400-regionen. Insättning är en plot som visar tilldelade klasser över alla ramar. (D) Klassmedelvärden erhållna med tidsbegränsad 2D-klustring och inriktning. Kredit:Science Advances, doi:10.1126/sciadv.abe6679 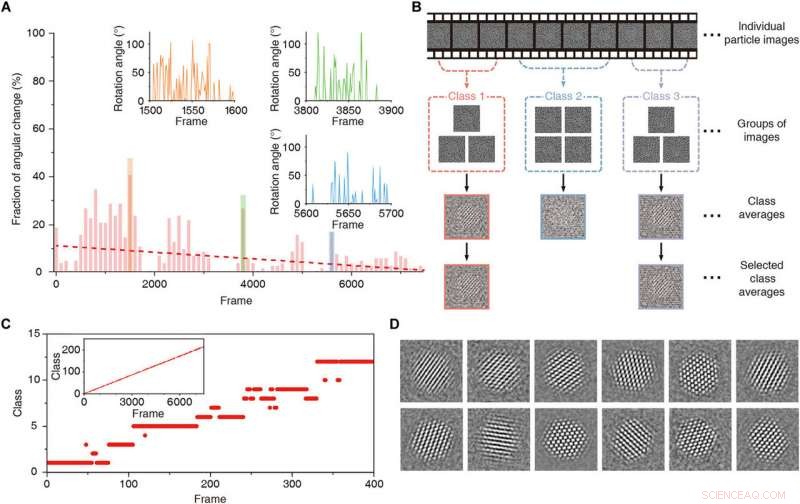
Reboul et al. introducerade en ny spårningsmetod som använder snabba Fourier-transformationer och faskorrelationen för att identifiera ett korrelationsmaximum med subpixelnoggrannhet. Teamet avböjde extraktionstidsfönstret med hjälp av total variation (TV)-baserad denoising och kombinerad deoising och tidsgenomsnitt för att tillhandahålla en robust metod för att spåra rörelsen hos individuella nanokristaller genom hela provet. Metoden gjorde det möjligt för dem att urskilja den övergripande formen på nanokristallerna och/eller deras kristallina egenskaper – vilket vittnade om spårningsalgoritmens robusthet. Med hjälp av metoden, de återhämtade också tidigare utmanande banor för att erhålla 3D-rekonstruktioner och använde en bakgrundssubtraherad partikelbana i alla bildbehandlingssteg för grafensubtraktion av GLC (grafen flytande cell). Teamet karakteriserade vidare karaktären av nanokristallrotationer i det mycket begränsade utrymmet i grafenvätskecellen. Metoden var icke-trivial på grund av den probabilistiska karaktären hos 3-D-rekonstruktionsalgoritmen. Teamet införlivade därför en deterministisk metod för att förbättra klustrets noggrannhet, samtidigt som signal-brusförhållandet förbättras jämfört med de enskilda bildrutorna.
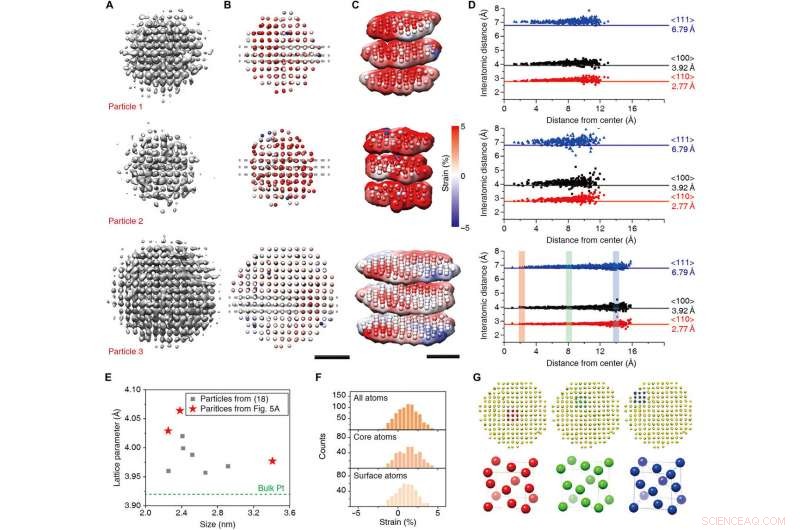
3D-rekonstruktionsresultat och strukturanalys på atomnivå. (A till C) 3D-densitetskartor (A), radiella töjningskartor från inpassade atomkoordinater (B), och deras skivrepresentation (C). (D) Interatomiska avstånd i riktningarna för <110> (röd), <100> (svart), och <111> (blå) för tre nanokristaller. (E) Inpassade gitterparametrar för tidigare rapporterade nanokristaller (18) (grå rutor) och nya resultat (röda stjärnor). (F) Histogram för radiell töjning av alla atomer (överst), kärnatomer (mitten), ytatomer (botten) av partikel 3. (G) Enhetscellstruktur av kärna (röd), mitten (grön), och yta (blå) av partikel 3. Skalstaplar, 1 nm. Kredit:Science Advances, doi:10.1126/sciadv.abe6679
Genererar modeller
Forskarna utvecklade sedan en startmodell med hjälp av kunskapen att partiklar har en ungefärlig kubisk atomposition. De simulerade atomdensiteterna med hjälp av 5-Gaussiska atomspridningsfaktorer. 2-D-projektionerna av den simulerade 3-D-densiteten representerade karaktären av projektioner i kärnan av nanokristallen, för att övervinna problem relaterade till translationell symmetri och en störande bakgrundssignal. 3D-förfiningsmetoden som används för biologisk kryoelektronmikroskopi kunde inte enkelt tillämpas på tidsseriedata för nanokristaller; därför, Reboul et al. infört kritiska ändringar. De använde ett tvåstegsförfiningsschema för att fastställa den korrekta formen på nanokristallen för att tillåta atomer och deras former att driva 3D-inriktning. Forskarna valde tre nanokristaller av varierande storlek som inte tidigare rekonstruerades för benchmarking, sedan använda atomkartor framtagna med metoden, Reboul et al. erhållit mikroskopiska strukturella detaljer på atomnivå. Arbetet underlättade också atomkartor som beskriver stamanalys och enhetscellstrukturanalys.
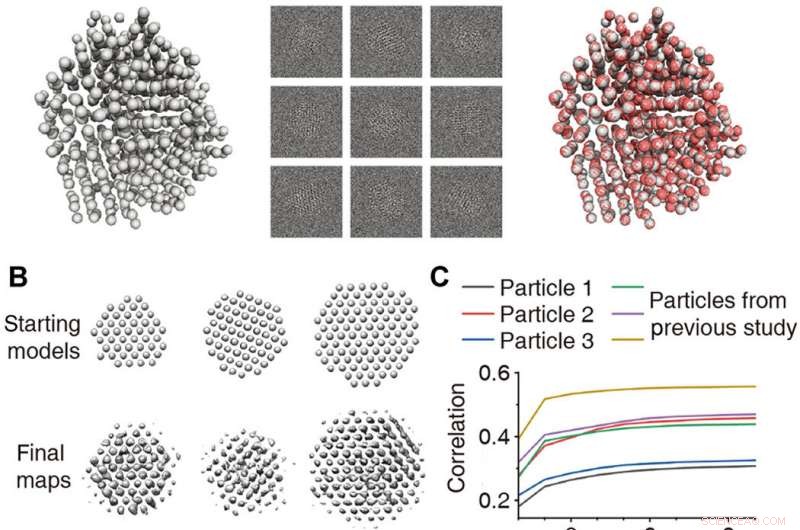
Validering av 3D-rekonstruktioner. (A) 3D-rekonstruktion av simulerad oordnad partikel med känd atomstruktur. Till vänster:Modell av en oordnad nanokristall erhållen genom simulering av molekylär dynamik. Mellan:5000 simulerade bilder med flera skivor med brus lagt till för att ge ett SNR =0,1. Till höger:Atomkarta (röd) som ett resultat av 3D-rekonstruktion överlagd med grundsannningsmodellen (grå). (B) Starta 3D-modeller och slutliga 3D-densitetskartor erhållna från experimentella data. (C) Korrelation mellan reprojektioner av den raffinerade 3D-densitetskartan och de experimentella partikelvyerna plottade som en funktion av iteration för det första steget av 3D-förfining. Partiklar som presenteras i denna artikel (svarta, röd, och blå färg) och presenterades i en tidigare studie (18) (grön, lila, och ockra färg) plottas. (D) Jämförelse av klassmedelvärden (anges som projektion) med omprojektioner för validering av de tre strukturerna. (E) Tidsberoende atomrepresentation av projektionsriktningarna för de tre strukturerna:vit (början) till rosa (mitten) till röd (slut). Röd, gul, och blå pilar indikerar x, y, och z-axlar, respektive. Kredit:Science Advances, doi:10.1126/sciadv.abe6679
Validerar 3D-rekonstruktionerna
Forskarna genererade vidare en modell av en oordnad nanokristall med hjälp av simuleringar av molekylär dynamik för att förstå tillämpbarheten av SINGLE på mycket oordnade nanokristaller. Använda simuleringar med flera skivor, de tillämpade translationell rörelse och slumpmässiga defokusvariationer för att representera realistisk partikelrörelse. De erhöll sedan en 3D-densitetskarta av den oordnade nanokristallen från 500 simulerade bilder med ett signal-brusförhållande på 0,1 och en startmodell med perfekt kristallin ordning för att överensstämma utmärkt med de ursprungliga partiklarna. Teamet fick fördelningen av projektionsriktningarna för de roterande nanokristallerna för att validera kvaliteten på 3D-rekonstruktionen och kommer att kräva ytterligare studier för att förstå hur de faktiska atomstrukturerna hos nanokristaller påverkar rotationsdynamiken.
På det här sättet, Cyril F. Reboul och kollegor demonstrerade beräkningsmetoder i SINGLE för att erhålla atomär upplösning nanokristalldensitetskartor. Genom att använda en avancerad vätskecellkonfiguration som grafenvätskeceller med ordnade nanokammare, teamet tillät kontroll av vätsketjockleken för att utöka tillämpbarheten av SINGLE för effektiv datainsamling. SINGLE-sviten tillhandahöll en första-i-studie effektiv analytisk plattform för att förstå det strukturella ursprunget till de unika fysikaliska och kemiska egenskaperna hos nanokristaller i deras ursprungliga lösningsfas.
© 2021 Science X Network