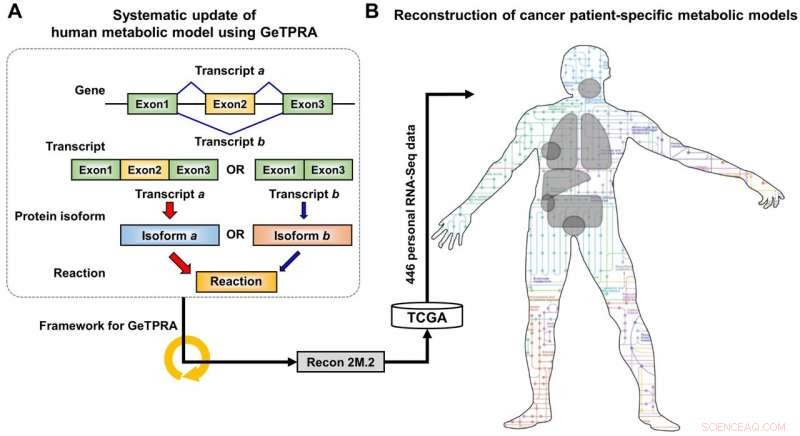
Schema för Recon 2M.2-utveckling och dess användning vid rekonstruktion av personliga metaboliska modeller i genomskala (GEM). (A) Vid utvecklingen av Recon 2M.2, ett koncept med alternativ splitsning av mänskliga gener övervägdes, och införlivas i modellering genom beräkningsramverket för Gene-Transcript-Protein-Reaction Associations (GeTPRA). (B) Vid utvecklingen av Recon 2M.2, cancerpatientspecifika GEM kan rekonstrueras med hjälp av personliga biologiska data (RNA-Seq-data). I den här studien, patientspecifika RNA-Seq-data erhölls från The Cancer Genome Atlas (TCGA; https://cancergenome.nih.gov/). Kredit:KAIST
Ett koreanskt forskarlag från KAIST utvecklade ett beräkningsramverk som möjliggör rekonstruktion av en omfattande beräkningsmodell av mänsklig metabolism, vilket möjliggör en exakt förutsägelse av personliga metaboliska egenskaper (eller fenotyper).
Genom att förstå personliga metaboliska fenotyper kan vi utforma effektiva terapeutiska strategier för olika kroniska och infektionssjukdomar. En mänsklig beräkningsmodell som kallas genome-scale metabolic model (GEM) innehåller information om tusentals metaboliska gener och deras motsvarande reaktioner och metaboliter, och har spelat en viktig roll för att förutsäga metaboliska fenotyper. Även om flera versioner av mänskliga GEMs har släppts, de hade utrymme för vidareutveckling, speciellt när det gäller att införliva biologisk information som kommer från en mänsklig genetikmekanism som kallas "alternativ splitsning". Alternativ splitsning är en genetisk mekanism som gör att en gen kan ge upphov till flera reaktioner, och är starkt förknippad med patologi.
För att ta itu med detta problem, Jae Yong Ryu (en doktorand), Dr. Hyun Uk Kim (forskarstipendiat), och framstående professor Sang Yup Lee, alla från Institutionen för kemi- och biomolekylär teknik vid KAIST, utvecklat ett beräkningsramverk som systematiskt genererar metabola reaktioner, och lägger till dem till den mänskliga GEM. Den resulterande mänskliga GEM visades exakt förutsäga metaboliska fenotyper under olika miljöförhållanden. Forskningsresultaten publicerades online i Proceedings of the National Academy of Sciences ( PNAS ) den 24 oktober, 2017, under rubriken "Ramverk och resurs för mer än 11, 000 gen-transkript-protein-reaktionsföreningar i mänsklig metabolism."
Forskargruppen uppdaterade först det biologiska innehållet i en tidigare version av den mänskliga GEM. Det uppdaterade biologiska innehållet inkluderar metaboliska gener och deras motsvarande metaboliter och reaktioner. Särskilt, metaboliska reaktioner katalyserade av redan kända proteinisoformer inkorporerades dessutom i den mänskliga GEM; proteinisoformer är flera varianter av proteiner som genereras från individuella gener genom den alternativa splitsningsprocessen. Varje proteinisoform är ofta ansvarig för funktionen av en metabolisk reaktion. Även om flera proteinisoformer genererade från en gen kan spela olika funktioner genom att ha olika uppsättningar av proteindomäner och/eller subcellulära lokaliseringar, sådan information beaktades inte korrekt i tidigare versioner av mänskliga GEM.
Efter den första uppdateringen av den mänskliga GEM, heter Recon 2M.1, forskargruppen implementerade därefter ett beräkningsramverk som systematiskt genererar information om Gene-Transcript-Protein-Reaction Associations (GeTPRA) för att identifiera proteinisoformer som tidigare inte identifierats. Detta ramverk utvecklades i denna studie. Som ett resultat av implementeringen av ramverket för GeTPRA, fler än 11, 000 GeTPRA förutspåddes automatiskt, och grundligt validerad. Ytterligare metabola reaktioner lades sedan till Recon 2M.1 baserat på den förutspådda GeTPRA för de tidigare okarakteriserade proteinisoformerna; Recon 2M.1 bytte namn till Recon 2M.2 från denna uppgradering. Till sist, Recon 2M.2 integrerades med 446 uppsättningar av personliga biologiska data (RNA-Seq-data) för att bygga patientspecifika cancermodeller. Dessa patientspecifika cancermodeller användes för att förutsäga cancermetabolismaktiviteter och anticancermål.
Utvecklingen av en ny version av mänskliga GEMs tillsammans med beräkningsramverket för GeTPRA förväntas öka studierna i grundläggande mänsklig genetik och medicin. Modellfiler av de mänskliga GEMs Recon 2M.1 och 2M.2, en fullständig lista över GeTPRA och källkoden för beräkningsramverket för att förutsäga GeTPRA är alla tillgängliga som en del av publiceringen av denna studie.
Den framstående professor Lee sa, "Den förutspådda GeTPRA från beräkningsramverket förväntas fungera som en riktlinje för framtida experiment på mänsklig genetik och biokemi, medan den resulterande Recon 2M.2 kan användas för att förutsäga läkemedelsmål för olika mänskliga sjukdomar."