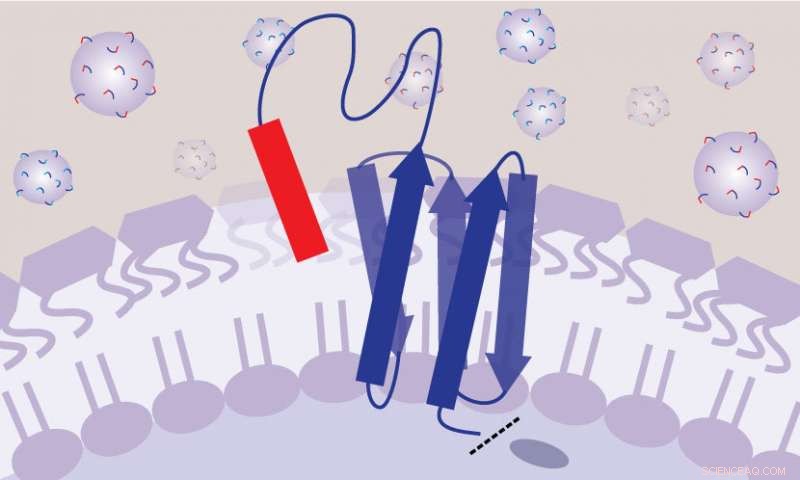
I SLAY -metoden, varje bakterie är genetiskt konstruerad för att producera en molekyl på sin cellyta som är en del peptid och en del tether-som en tetherball på en lekplats. Detta arrangemang tillåter peptiderna att efterlikna fritt flytande läkemedel i människokroppen. Upphovsman:Ashley Tucker
Biologer vid University of Texas i Austin har utvecklat en metod för att snabbt screena hundratusentals potentiella läkemedel för att bekämpa infektioner, en innovation som lovar att bekämpa den växande gissel av antibiotikaresistenta bakterier. Metoden innebär att man konstruerar bakterier för att producera och testa molekyler som är potentiellt giftiga för sig själva.
En beskrivning av metoden visas i den 25 januari tryckta upplagan av tidskriften Cell .
Ingen ny antibiotikaklass har upptäckts på 40 år - många av de mest tillgängliga i naturen har redan hittats, och processen för att skapa och testa nya från grunden är långsam och mödosam - men modern medicin är i stort behov av dem. Enligt Världshälsoorganisationen, antibiotika har lagt till cirka 20 år till den genomsnittliga mänskliga livslängden. Men deras skyddande fördelar glider bort när bakterier utvecklar antibiotikaresistens.
I deras konceptbevis, UT Austin -teamet, ledd av Bryan Davies, visade cirka 800, 000 molekyler kallade peptider för att se om de hade antimikrobiella effekter, vilket betyder att de dödade skadliga bakterier. Av dem, flera tusen dödade E. coli -bakterier, vilket gör dem till potentiella ledare för antibiotika. Vissa antibiotika som för närvarande används är peptider. Uppföljningsforskning kommer att vara nödvändig för att avgöra vilken, om någon, av tusentals nya leads är verkligen effektiva och säkra hos människor, men forskarna visade att minst en sådan molekyl, kallad P7, dödar också andra former av patogena bakterier och är säker på möss.
Med denna metod, kallad SLAY (Surface Localized Antimicrobial Display), en person kan screena hundratusentals liknande peptider snabbare och mer kostnadseffektivt än befintliga metoder kan. Davies skulle vilja se att metoden blir ett standardverktyg i den globala jakten på nya antibiotika.
"Så tänk om vi har tusen grupper som alla använder detta system för att följa sina egna intressen och sina egna peptider?" sa Davies, biträdande professor i molekylär biovetenskap. "När du aktiverar en grupp av den storleken, då tror jag att du har större chans att faktiskt hitta ett nytt antibiotikum som fungerar. "
Ett viktigt framsteg i detta arbete var att ta reda på hur man får bakterier att producera molekyler som kan vara giftiga för dem själva och att kontrollera hur dessa molekyler interagerar med sina värdbakterier.
"Vi trodde, vore det inte bra om en bakterie kunde syntetisera föreningen för oss, eftersom bakterier är billiga och lätta att odla, och testa sedan föreningen på sig själv och rapportera tillbaka och berätta för oss, var det antimikrobiellt eller inte? "sa Davies.
Deras lösning var att genetiskt manipulera bakterierna för att producera en molekyl på cellytan som är en del peptid och en del tether - som en lekplats tetherball och dess tether - med ena änden fixerad till cellmembranet och den andra änden fri att flyta runt. Detta gör att peptiden kan röra sig och komma i kontakt med bakteriecellens yta, som om det var fritt flytande som ett läkemedel i blodet, men utan att interagera med andra närliggande bakterier.
Genom att se till att varje version av tetherball bara interagerar med bakterierna som producerade den, forskarna kunde då göra ett stort steg i effektivitet. De kan skapa hundratusentals bakteriestammar - var och en genetiskt konstruerad för att producera en något annorlunda version av tetherballen - och sätta alla dessa stammar i samma provrör för att växa. Genom att köra hundratusentals experiment samtidigt, deras metod sparar enormt mycket utrymme, tid och kostnad.
En del av denna process bygger på en teknik som utvecklades av UT Austins George Georgiou på 1990 -talet som får bakterier att producera proteiner eller peptider på sina ytor.
För att ta reda på vilka tetherballs (peptider) som slår ut sina värdar, forskarna använder gensekvensering för att identifiera vilka versioner som produceras av bakterier i början och vilka som produceras i slutet.
Efter upptäckten att P7 dödar patogener, laget planerar nu att skapa tusentals subtila variationer av denna molekyl, kallas derivat, och kör dem genom samma screeningprocess för att söka efter en ännu mer effektiv version.
Postdoktor Ashley Tucker ledde det experimentella arbetet med att demonstrera användningen av plattformen.
Davies, Tucker och UT Austin har lämnat in patentansökningar för SLAY -metoden och för de specifika genetiska sekvenserna för de tusentals antimikrobiella peptider som de hittat hittills.