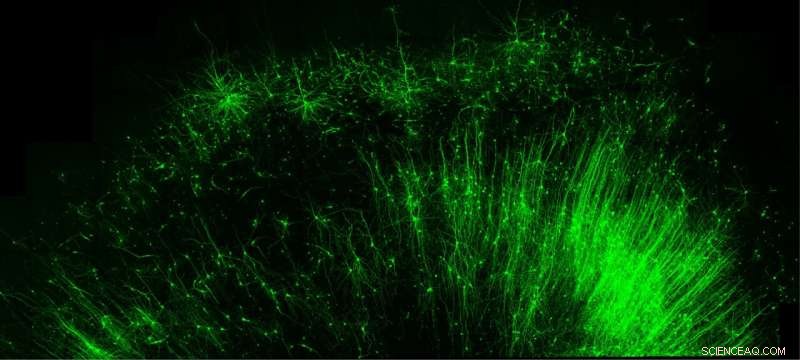
Märkning och belysning av endast de hämmande "broms"-cellerna (gröna) i mänsklig hjärnvävnad är bara en av många saker som det nya verktyget från Duke University, CellREADR, kan göra. Kredit:Derek Southwell, Duke University
Duke University-forskare har utvecklat ett RNA-baserat redigeringsverktyg som riktar sig mot enskilda celler, snarare än gener. Det är kapabelt att exakt rikta in sig på alla typer av celler och selektivt lägga till vilket protein som helst av intresse.
Forskare sa att verktyget kan göra det möjligt att modifiera mycket specifika celler och cellfunktioner för att hantera sjukdomar.
Med hjälp av en RNA-baserad sond, ledde ett team av neurobiologen Z. Josh Huang, Ph.D. och postdoktor Yongjun Qian, Ph.D. visat att de kan införa fluorescerande taggar i celler för att märka specifika typer av hjärnvävnad; en ljuskänslig på/av-knapp för att tysta eller aktivera neuroner som de väljer; och till och med ett självförstörande enzym för att exakt radera vissa celler men inte andra. Verket visas 5 oktober i Nature .
Deras selektiva cellövervaknings- och kontrollsystem bygger på ADAR-enzymet, som finns i varje djurs celler. Även om det här är tidiga dagar för CellREADR (Cell access through RNA sensing by Endogenous ADAR), verkar de möjliga tillämpningarna vara oändliga, sa Huang, liksom dess potential att arbeta över hela djurriket.
"Vi är glada eftersom detta ger en förenklad, skalbar och generaliserbar teknik för att övervaka och manipulera alla celltyper i alla djur," sa Huang. "Vi skulle faktiskt kunna modifiera specifika typer av cellfunktioner för att hantera sjukdomar, oavsett deras ursprungliga genetiska predisposition," sa han. "Det är inte möjligt med nuvarande terapier eller medicin."
CellREADR är en anpassningsbar sträng av RNA som består av tre huvudsektioner:en sensor, en stoppskylt och en uppsättning ritningar.
Först bestämmer forskargruppen vilken specifik celltyp de vill undersöka och identifierar ett mål-RNA som är unikt producerat av den celltypen. Verktygets anmärkningsvärda vävnadsspecificitet bygger på det faktum att varje celltyp tillverkar signatur-RNA som inte syns i andra celltyper.
En sensorsekvens utformas sedan som mål-RNA:s komplementära sträng. Precis som stegpinnarna på DNA är uppbyggda av komplementära molekyler som är naturligt attraherade av varandra, har RNA samma magnetiska potential att länka till ett annat RNA-stycke om det har matchande molekyler.
Efter att en sensor tagit sig in i en cell och hittar sin mål-RNA-sekvens, glom båda bitarna ihop för att skapa en bit dubbelsträngat RNA. Denna nya RNA-mashup utlöser enzymet ADAR för att inspektera den nya skapelsen och sedan ändra en enda nukleotid av dess kod.
The ADAR enzyme is a cell-defense mechanism designed to edit double-strand RNA when it occurs, and is believed to be found in all animal cells.
Knowing this, Qian designed CellREADR's stop sign using the same specific nucleotide ADAR edits in double-stranded RNA. The stop sign, which prevents the protein blueprints from being built, is only removed once CellREADR's sensor docks to its target RNA sequence, making it highly specific for a given cell type.
Once ADAR removes the stop sign, the blueprints can be read by cellular machinery that builds the new protein inside the target cell.
In their paper, Huang and his team put CellREADR through its paces. "I remember two years ago when Yongjun built the first iteration of CellREADR and tested it in a mouse brain," Huang said. "To my amazement, it worked spectacularly on his first try."
The team's careful planning and design paid off as they were then able to demonstrate CellREADR accurately labelled specific brain cell populations in living mice, as well as effectively added activity monitors and control switches where directed. It also worked well in rats, and in human brain tissue collected from epilepsy surgeries.
"With CellREADR, we can pick and choose populations to study and really begin to investigate the full range of cell types present in the human brain," said co-author Derek Southwell, M.D., Ph.D., a neurosurgeon and assistant professor in the department of neurosurgery at Duke.
Southwell hopes CellREADR will improve his and others' understanding of the wiring diagram for human brain circuits and the cells within them, and in doing so, help advance new therapies for neurological disorders, such as a promising new method to treat drug-resistant epilepsy he is piloting.
Huang and Qian are especially hopeful about CellREADR's potential as a "programmable RNA medicine" to possibly cure diseases—since that's what drew them both to science in the first place. They have applied for a patent on the technology.
"When I majored in pharmacology as an undergraduate, I was very naïve," Qian said. "I thought you could do a lot of things, like cure cancer, but actually it's very difficult. However, now I think, yes maybe we can do it." + Utforska vidare