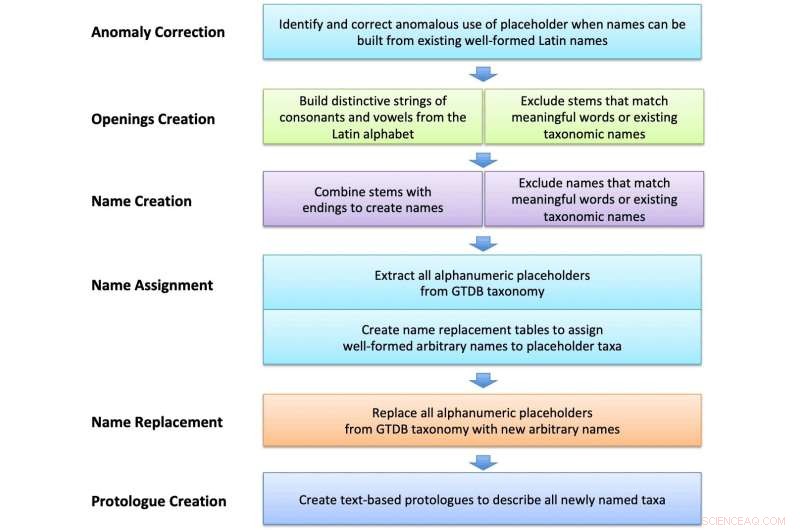
Schematiskt arbetsflöde för att skapa och tilldela godtyckliga latinska namn. Strängar byggda av latinska bokstäver valdes ut och kurerades för att utesluta oönskade komponenter och meningsfulla ord. Dessa kombinerades med latinska suffix eller ord för att skapa namn, som sedan användes för att namnge platshållartaxa i GTDB-filer och skapa textbaserade protologer. Kredit:International Journal of Systematic and Evolutionary Microbiology (2022). DOI:10.1099/ijsem.0.005482
I en artikel som nyligen publicerades i International Journal of Systematic and Evolutionary Microbiology , har forskare i Storbritannien och Österrike namngett mer än 65 000 olika typer av mikrober. Studien, ledd av professor Mark Pallen vid Quadram Institute i Norwich, bygger på en lång tradition av att skapa välformade men godtyckliga latinska namn för nya arter, men tillämpar detta tillvägagångssätt i en skala som saknar motstycke i taxonomins historia.
Prof. Pallen hävdar att mikrobiologi är ett offer för sin egen framgång, med tiotusentals nya arter som upptäckts under de senaste åren – men de flesta förblir namnlösa. Tidigare har bakterier fått beskrivande namn eller fått namn efter personer eller platser. Detta tillvägagångssätt levererar för närvarande omkring tusen nya artnamn per år. Men med en eftersläpning på>50 000 välklassificerade men icke namngivna arter, med denna framstegshastighet, skulle det ta minst ett halvt sekel att namnge alla dessa icke namngivna bakterier – då skulle forskare möta problemet med att namnge de miljontals fler arterna upptäckt under tiden.
Pallen och kollegor har antagit en effektiv, hög genomströmning, big-data-strategi, med hjälp av ett datorprogram för att generera tiotusentals distinkta men lättanvända namn som inte liknar några befintliga ord. För att uppfylla kravet att namn på bakterier ska vara på latin, kombinerade teamet godtyckliga bokstäversträngar från det latinska alfabetet med grammatiskt välformade feminina suffix. Resultatet är en uppsättning namn som påminner om latinets förtrogenhet och gravitas, även om de saknar någon meningsfull stamtavla. Exempel är Dupisella tifacia för en bakterie från fårtarmen, Hopelia gocarosa för en bakterie som lever i svenskt grundvatten eller Saxicetta apufaria från en rysk saltsjö.
Även om detta kan tyckas radikalt, har namnbildningen på ett godtyckligt sätt en lång tradition, som sträcker sig tillbaka till den första taxonomiska nomenklaturkoden 1869 och till och med tillbaka till taxonomins fader, den svenske naturforskaren Linnéa. Vad som är annorlunda här är den häpnadsväckande känslan av skala, med en katalog med namn som sträcker sig över tio tusen sidor i vad som representerar den största namngivningen av arter någonsin i en enda publikation.
Eftersom de har applicerats på arter som upptäckts genom DNA-sekvensering men ännu inte odlats i labbet, förblir de nya namnen för närvarande provisoriska ganska permanenta. Men eftersom nomenklaturkoden insisterar på att bakteriologer strävar efter stabilitet i namn och inga konkurrerande tillvägagångssätt för namngivning i skala är på gång, verkar det troligt att den stora majoriteten av de nya namnen kommer att användas i år eller till och med århundraden framöver.
Avslutningsvis säger Pallen "Jag var medlem i arbetsgruppen som gav oss grekiska bokstäver för COVID-varianter, som snabbt antogs av forskarsamhället. Jag hoppas att de namn som föreslås här också snabbt antas och används i stor utsträckning. Detta är bara det första steget. Den mikrobiella upptäcktens tidsålder är långt ifrån förbi, men det kommer att vara lätt att skapa framtida namn en masse med hjälp av de principer vi har etablerat här." + Utforska vidare