
Proteiner designade med ett ultrasnabbt mjukvaruverktyg som heter ProteinMPNN var mycket mer benägna att vikas ihop som avsett. Kredit:Ian Haydon, UW Medicine Institute for Protein Design
Under de senaste två åren har maskininlärning revolutionerat förutsägelsen av proteinstruktur. Nu, tre uppsatser i Science beskriva en liknande revolution inom proteindesign.
I de nya tidningarna visar biologer vid University of Washington School of Medicine att maskininlärning kan användas för att skapa proteinmolekyler mycket mer exakt och snabbare än vad som tidigare varit möjligt. Forskarna hoppas att detta framsteg kommer att leda till många nya vacciner, behandlingar, verktyg för kolavskiljning och hållbara biomaterial.
"Proteiner är grundläggande i biologin, men vi vet att alla proteiner som finns i varje växt, djur och mikrob utgör mycket mindre än en procent av vad som är möjligt. Med dessa nya mjukvaruverktyg borde forskare kunna hitta lösningar på långa – Stående utmaningar inom medicin, energi och teknologi, säger seniorförfattaren David Baker, professor i biokemi vid University of Washington School of Medicine och mottagare av ett genombrottspris 2021 i biovetenskap.
Proteiner kallas ofta för "livets byggstenar" eftersom de är avgörande för strukturen och funktionen hos allt levande. De är involverade i praktiskt taget alla processer som äger rum inuti celler, inklusive tillväxt, delning och reparation. Proteiner är uppbyggda av långa kedjor av kemikalier som kallas aminosyror. Sekvensen av aminosyror i ett protein bestämmer dess tredimensionella form. Denna invecklade form är avgörande för att proteinet ska fungera.
Nyligen har kraftfulla maskininlärningsalgoritmer inklusive AlphaFold och RoseTTAFold tränats för att förutsäga de detaljerade formerna av naturliga proteiner baserat enbart på deras aminosyrasekvenser. Maskininlärning är en typ av artificiell intelligens som gör att datorer kan lära sig av data utan att vara explicit programmerad. Maskininlärning kan användas för att modellera komplexa vetenskapliga problem som är för svåra för människor att förstå.
För att gå utöver de proteiner som finns i naturen delade Bakers teammedlemmar upp utmaningen med proteindesign i tre delar och använde nya mjukvarulösningar för var och en.
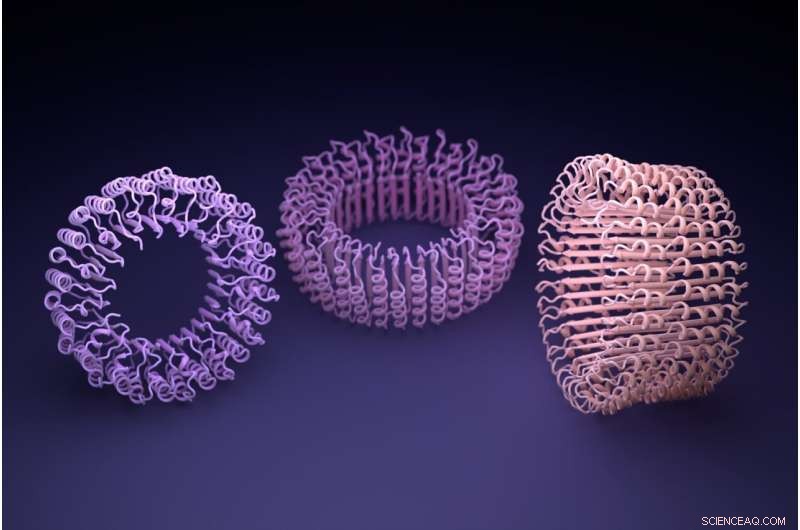
Artificiell intelligens hallucinerade dessa symmetriska proteinsammansättningar, på ett sätt som liknar andra A.!. generativa verktyg som producerar utdata baserat på enkla uppmaningar. Kredit:Ian Haydon, UW Medicine Institute for Protein Design
Först måste en ny proteinform genereras. I en artikel publicerad 21 juli i tidskriften Science , visade teamet att artificiell intelligens kan generera nya proteinformer på två sätt. Den första, kallad "hallucination", är besläktad med DALL-E eller annan generativ A.I. verktyg som producerar utdata baserat på enkla uppmaningar. Den andra, kallad "inpainting", är analog med autoslutförandefunktionen som finns i moderna sökfält.
För det andra, för att påskynda processen, tog teamet fram en ny algoritm för att generera aminosyrasekvenser. Beskrivs i numret av 15 september av Science , detta mjukvaruverktyg, kallat ProteinMPNN, körs på ungefär en sekund. Det är mer än 200 gånger snabbare än den tidigare bästa mjukvaran. Dess resultat är överlägsna tidigare verktyg, och programvaran kräver ingen expertanpassning för att köras.
"Neurala nätverk är lätta att träna om du har massor av data, men med proteiner har vi inte så många exempel som vi skulle vilja. Vi var tvungna att gå in och identifiera vilka egenskaper i dessa molekyler som är de viktigaste. Det var lite trial and error", säger projektforskaren Justas Dauparas, postdoktor vid Institutet för Proteindesign
För det tredje använde teamet AlphaFold, ett verktyg utvecklat av Alphabet's DeepMind, för att självständigt bedöma om aminosyrasekvenserna de kom fram till sannolikt skulle vikas till de avsedda formerna.
"Programvara för att förutsäga proteinstrukturer är en del av lösningen men den kan inte komma med något nytt på egen hand", förklarade Dauparas.
"ProteinMPNN är att designa proteiner vad AlphaFold var för förutsägelse av proteinstruktur", tillade Baker.

Detalj av ett protein designat med hjälp av ett snabbt verktyg som kallas ProteinMPNN, ytterligare ett framsteg i användningen av artificiell intelligens och maskininlärning i proteindesign. Kredit:Ian Haydon, UW Medicine Institute for Protein Design
I en annan tidning som visas i Science 15 september bekräftade ett team från Baker-labbet att kombinationen av nya verktyg för maskininlärning på ett tillförlitligt sätt kunde generera nya proteiner som fungerade i laboratoriet.
"Vi fann att proteiner gjorda med ProteinMPNN var mycket mer benägna att vikas upp som avsett, och vi kunde skapa mycket komplexa proteinsammansättningar med dessa metoder", säger projektforskaren Basile Wicky, en postdoktor vid Institutet för Proteindesign.
Bland de nya proteinerna som tillverkades fanns ringar i nanoskala som forskarna tror kan bli delar till anpassade nanomaskiner. Elektronmikroskop användes för att observera ringarna, som har en diameter som är ungefär en miljard gånger mindre än ett vallmofrö.
"Detta är själva början av maskininlärning inom proteindesign. Under de kommande månaderna kommer vi att arbeta med att förbättra dessa verktyg för att skapa ännu mer dynamiska och funktionella proteiner", säger Baker.
Datorresurser för detta arbete donerades av Microsoft och Amazon Web Services. + Utforska vidare