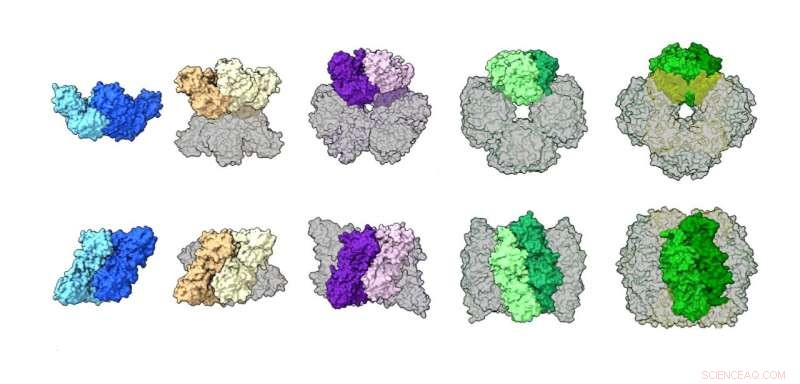
En jämförelse av rubisco-aggregat från olika arter, som illustrerar ett brett utbud av strukturer inklusive en dimer, tetramer, hexamer, oktamer och hexadecamer (16 enheter protein). Kredit:Shih Lab/Berkeley Lab
När du tänker på proteiner – enzymer, signalmolekyler och strukturella komponenter i allt levande – kanske du tänker på enstaka strängar av aminosyror, organiserade som pärlor på ett snöre. Men nästan alla proteiner består av flera strängar hopvikta och bundna till varandra och bildar komplicerade 3D-överbyggnader som kallas molekylära sammansättningar. Ett av nyckelstegen för att förstå biologi är att upptäcka hur ett protein gör sitt jobb, vilket kräver kunskap om dess strukturer ner till atomnivå.
Under det senaste århundradet har forskare utvecklat och implementerat fantastiska teknologier som röntgenkristallografi och kryoelektronmikroskopi för att bestämma proteinstruktur och därmed svarat på otaliga viktiga frågor. Men nytt arbete visar att förståelsen av proteinstruktur ibland kan vara mer komplicerad än vi tror.
En grupp forskare från Lawrence Berkeley National Laboratory (Berkeley Lab) som studerar världens mest förekommande protein, ett enzym som är involverat i fotosyntesen som kallas rubisco, visade hur evolution kan leda till en överraskande mångfald av molekylära sammansättningar som alla utför samma uppgift. Resultaten, publicerade idag i Science Advances , avslöjar möjligheten att många av de proteiner vi trodde att vi visste faktiskt existerar i andra, okända former.
Historiskt sett, om forskare löste en struktur och fastställde att ett protein var dimeriskt (bestod av två enheter), till exempel, skulle de kunna anta att liknande proteiner också existerade i en dimer form. Men liten provstorlek och provtagningsbias – oundvikliga faktorer med tanke på att det är mycket svårt att omvandla naturligt flytande proteiner till fasta, kristalliserade former som kan undersökas via röntgenkristallografi – fördunklade verkligheten.

Albert Lui (till vänster) och Patrick Shih visar hur de studerade strukturen och utvecklingen av rubisco, enzymet som växter använder för att utnyttja CO2 för att skapa socker, i Koshland Hall vid UC Berkeley. Kredit:Marilyn Sargent/Berkeley Lab
"Det är som att om du gick utanför och såg någon gå ut med sin hund, om du aldrig hade sett en hund förut och sedan såg en wienerhund, skulle du tänka," OK, så här ser alla hundar ut. Men vad du behöver göra är att gå till hundparken och se all mångfald av hundar som finns där, säger huvudförfattaren Patrick Shih, en fakultetsforskare inom biovetenskapsområdet och chef för växtbiosystemdesign vid Joint BioEnergy Institute (JBEI). "En del av detta papper som går utöver rubisco, till alla proteiner, är frågan om huruvida vi ser det verkliga utbudet av strukturer i naturen eller inte, eller om dessa fördomar får det att verka som att allt ser ut som en wienerhund."
I hopp om att utforska alla de olika rubisco-arrangemangen i den metaforiska hundparken och lära sig var de kom ifrån, samarbetade Shihs labb med strukturbiologiexperter från Bioscience Area med hjälp av Berkeley Labs avancerade ljuskälla. Tillsammans studerade teamet en typ av rubisco (form II) som finns i bakterier och en delmängd av fotosyntetiska mikrober med hjälp av traditionell kristallografi - en teknik som kan upplösas på atomnivå - kombinerad med en annan strukturlösningsteknik, röntgenspridning med små vinklar (SAXS), som har lägre upplösning men kan ta ögonblicksbilder av proteiner i deras ursprungliga form när de är i flytande blandningar. SAXS har den ytterligare fördelen med hög genomströmningskapacitet, vilket innebär att den kan bearbeta dussintals individuella proteinsammansättningar i snabb följd.
Tidigare arbeten hade visat att den bättre studerade typen av rubisco som finns i växter (form I) alltid har en "oktamerisk kärna" sammansättning av åtta stora proteinenheter ordnade med åtta små enheter, medan form II troddes existera mestadels som en dimer med en några sällsynta exempel på sex-enhets hexamerer. Efter att ha använt dessa komplementära tekniker för att undersöka prover av rubisco från en mängd olika mikroberarter, observerade författarna att de flesta rubiscoproteiner av form II faktiskt är hexamerer, med en och annan dimer, och de upptäckte en aldrig tidigare sett tetramer (fyra enheter) hopsättning.
Genom att kombinera dessa strukturella data med respektive proteinkodande gensekvenser tillät teamet att utföra rekonstruktion av förfäders sekvenser - en datorbaserad molekylär evolutionsmetod som kan uppskatta hur förfäders proteiner såg ut baserat på sekvensen och utseendet på moderna proteiner som utvecklats från dem.
Albert Liu kontrollerade några odlingsplattor av E. coli med generna för att göra form II rubisco. Kredit:Albert Liu
Rekonstruktionen tyder på att genen för form II rubisco har förändrats under sin evolutionära historia för att producera proteiner med en rad strukturer som omvandlas till nya former eller går tillbaka till äldre strukturer ganska lätt. Däremot ledde selektiva tryck under evolutionens gång till en serie förändringar som låste form I rubisco på plats – en process som kallas strukturell förankring – vilket är anledningen till att den oktameriska sammansättningen är det enda arrangemanget vi ser nu. Enligt författarna antogs det att de flesta proteinsammansättningar var förankrade över tiden av selektivt tryck för att förfina sin funktion, som vi ser med form I rubisco. Men denna forskning tyder på att evolution också kan gynna flexibla proteiner.
"Det stora fyndet från det här dokumentet är att det finns mycket strukturell plasticitet", säger Shih, som också är biträdande professor vid UC Berkeley. "Proteiner kan vara mycket mer flexibla, över hela fältet, än vi har trott."
Efter att ha slutfört den förfäders sekvensrekonstruktionen genomförde teamet mutationsexperiment för att se hur förändring av rubisco-aggregatet, i det här fallet att bryta en hexamer till en dimer, påverkade enzymets aktivitet. Denna inducerade mutation producerade oväntat en form av rubisco som är bättre på att använda sin målmolekyl, CO2 . All naturligt förekommande rubisco binder ofta O2 av samma storlek molekyl vid olycka, vilket sänker enzymets produktivitet. Det finns ett stort intresse för att genetiskt modifiera rubisco i jordbruksväxtarter för att öka proteinets affinitet för CO2 , för att producera mer produktiva och resurseffektiva grödor. Däremot har det varit mycket fokus på proteinets aktiva plats – den region av proteinet där CO2 eller O2 binda.
"Detta är en intressant insikt för oss eftersom det tyder på att för att få mer fruktbara resultat som konstruerar rubisco, kan vi inte bara titta på det enklaste svaret, regionen av enzymet som faktiskt interagerar med CO2 ", sa förstaförfattaren Albert Liu, en doktorand i Shihs labb. "Kanske finns det mutationer utanför den aktiva platsen som faktiskt deltar i denna aktivitet och som potentiellt kan förändra proteinfunktionen på ett sätt som vi vill. Så det är något som verkligen öppnar dörrar till framtida forskningsvägar."
Medförfattare Paul Adams, Associate Laboratory Director for Biosciences och Vice President for Technology på JBEI tillade:"Blandningen av tekniker som används och teamets tvärvetenskapliga karaktär var en verklig nyckel till framgång. Arbetet belyser kraften i att kombinera genomisk data och strukturbiologiska metoder för att studera ett av de viktigaste problemen inom biologi och nå några oväntade slutsatser." + Utforska vidare