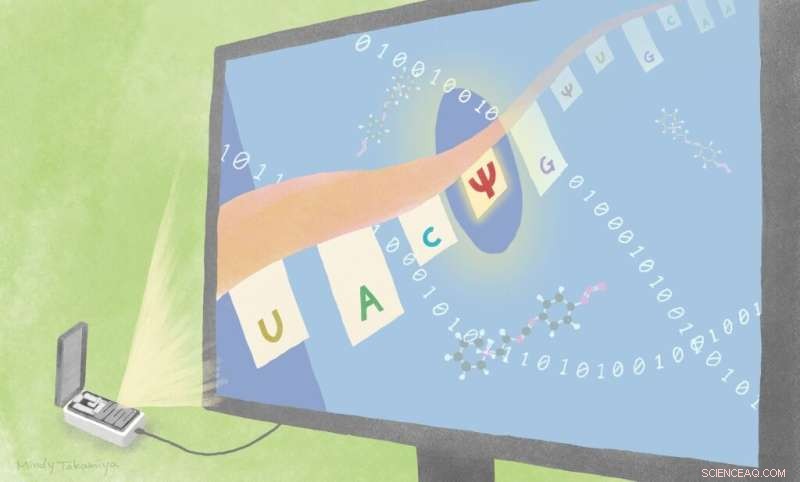
Forskare hittade två nya sätt att identifiera substitutioner från uracil till pseudouridin, genom att kombinera befintlig sekvenseringsteknologi med olika tillvägagångssätt. En är med algoritmbaserad beräkning och en är med en kemisk sond CMC. Kredit:Mindy Takamiya/Kyoto University iCeMS
Två nya tillvägagångssätt kan hjälpa forskare att använda befintlig sekvenseringsteknik för att bättre särskilja RNA-förändringar som påverkar hur genetisk kod läses.
Kyoto-universitetets forskare närmar sig att hitta sätt att identifiera förändringar i RNA-sekvenser som påverkar proteinbildningen och kan orsaka sjukdomar. Deras tillvägagångssätt, publicerad i tidskriften Genomics , använder sannolikhetsalgoritmer tillsammans med en redan tillgänglig sekvenseringsenhet i fickstorlek.
"Modifieringar som finns i alla typer av biologiskt RNA påverkar genreglering, som i slutändan avgör hur olika celler fungerar i vår kropp", förklarar Ganesh Pandian Namasivayam från Kyoto Universitys Institute for Integrated Cell-Material Science (WPI-iCeMS). "Onormaliteter i dessa modifieringar kan leda till allvarliga sjukdomar, som diabetes, neuroutvecklingsstörningar och cancer. Att veta hur och var dessa RNA-modifieringar är är av största vikt ur en klinisk synvinkel", tillägger Soundhar Ramaswamy, studiens första författare.
Det finns redan sätt att identifiera RNA-modifieringar, men de är otillräckliga. Biofysiska tillvägagångssätt som kromatografi och masspektrometri kan bara bearbeta små mängder RNA åt gången. Sekveneringsmetoder med hög genomströmning, som kan bearbeta stora mängder RNA, involvera mödosam provberedning, kan inte kartlägga flera modifieringar samtidigt och är felbenägna.
Namasivayam, Hiroshi Sugiyama och kollegor vid Kyoto University testade och hittade två tillvägagångssätt som relativt framgångsrikt kan särskilja en välkänd och riklig RNA-modifiering som involverar ersättning av nukleotidbasen uracil med en annan som kallas pseudouridin.
I likhet med DNA bildas RNA av en sträng av olika kombinationer av fyra olika nukleotidbaser:uracil, cytosin, adenin och guanin. Hur dessa baser är ordnade bestämmer koden som signalerar vilket protein som är tänkt att göras. När pseudouridin ersätter uracil i RNA-ryggraden kan det leda till ökad proteinproduktion eller att koden ändras från en kod som signalerar avbrott i informationsöversättning till en som signalerar aminosyrabildning.
Teamets tillvägagångssätt innebär att använda en redan tillgänglig direkt RNA-sekvenseringsplattform utvecklad av Oxford Nanopore Technologies. I denna plattform passerar RNA-strängar genom små porer i ett membran. Störningar orsakas av strömmen som rör sig genom membranet beroende på ordningen på de olika RNA-baserna. Detta gör det möjligt för forskare att "läsa" sekvensen. Men forskare som använder detta tillvägagångssätt har ofta svårt att skilja olika typer av modifieringar från varandra.
Shubham Mishra, en gemensam förstaförfattare till denna studie, utvecklade algoritmer för att identifiera en hög sannolikhet för existensen av en pseudouridinsubstitution jämfört med möjligheten att det var en annan typ av basförändring.
En av deras strategier jämför korta RNA-körningar av fem nukleotidbaser där uracil, pseudouridin eller cytosin är omgivna på båda sidor av samma baser. Avläsningarna går sedan igenom algoritmer som beräknar sannolikheten för att mellanbasen är en av de tre. De använde sin strategi, kallad Indo-Compare (Indo-C), på konstruerade RNA-sekvenser och sedan på jäst och humant RNA och fann att den var bra på att skilja pseudouridinsubstitutionerna från de andra.
De kunde också identifiera pseudouridinsubstitutioner genom att blanda en kemisk sond med RNA-prover, som sedan selektivt fäster till dem. Detta ändrade sekvensavläsningarna på ett sätt som identifierar modifieringen.
"Vi tror att vårt arbete kommer att göra nanopore-sekvenseringsbaserade metoder mindre mödosamma för att upptäcka RNA-modifieringar och mer kapabla att karakterisera effekterna av dessa modifieringar på utveckling och sjukdom", säger Namasivayam.
Teamet syftar sedan till att optimera användningen av båda metoderna tillsammans för att mer exakt identifiera RNA- och DNA-modifieringar. Detta kommer att innebära tillverkning av nya kemiska sonder som motsvarar specifika förändringar. De planerar också att vidareutveckla avancerade maskininlärningsalgoritmer som kompletterar kemiska sondbaserade direkt RNA-sekvenseringsmetoder. + Utforska vidare