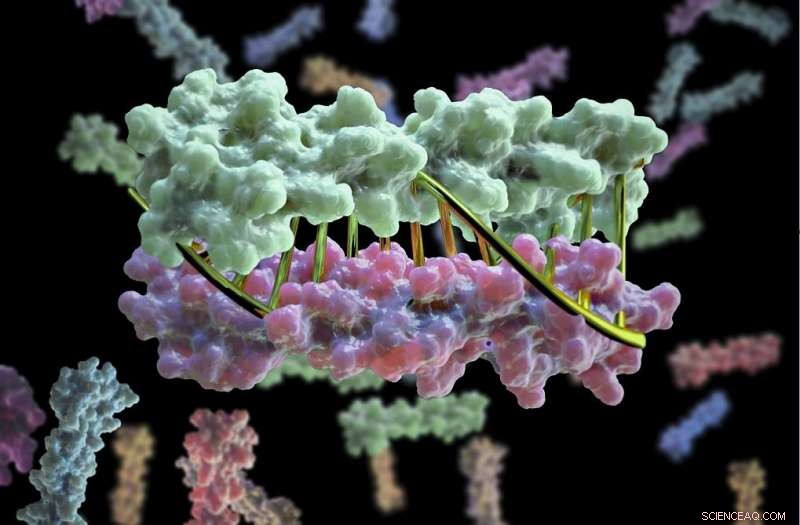
Proteiner som är utformade i laboratoriet kan nu zipas ihop på ungefär samma sätt som DNA -molekyler zipas upp för att bilda en dubbel helix. Tekniken kan möjliggöra design av proteinnanomaskiner som potentiellt kan hjälpa till att diagnostisera och behandla sjukdomar, möjliggöra mer exakt konstruering av celler och utföra en mängd andra uppgifter. Denna teknik ger forskare en exakt, programmerbart sätt att styra hur proteinmaskiner interagerar. Kredit:Institute for Protein Design
Proteiner har nu konstruerats i laboratoriet för att zip ihop på ungefär samma sätt som DNA -molekyler zip upp för att bilda en dubbel helix. Tekniken, vars utveckling leddes av forskare från University of Washington School of Medicine, kan möjliggöra utformning av proteinnanomaskiner som potentiellt kan hjälpa till att diagnostisera och behandla sjukdomar, möjliggöra mer exakt konstruering av celler och utföra en mängd andra uppgifter.
"För att alla maskiner ska fungera, dess delar måste sammanfalla exakt, "sa Zibo Chen, huvudförfattaren till uppsatsen och en UW -doktorand i biokemi. "Denna teknik gör det möjligt för dig att designa proteiner så att de kommer ihop exakt hur du vill att de ska."
Forskningen utfördes vid UW Medicine's Institute of Protein Design, regisserad av David Baker, professor i biokemi vid University of Washington School of Medicine och en Howard Hughes Medical Institute -utredare. Forskarna rapporterar sina fynd i tidningen 19 december Natur .
Förr, forskare som är intresserade av att designa biomolekylära nanomaskiner har ofta använt DNA som en viktig komponent. Detta beror på att DNA -strängar kommer ihop och bildar vätebindningar för att skapa DNA:s dubbla helix, men bara om deras sekvenser är komplementära.
Teamet utvecklade nya proteindesignalgoritmer som producerar kompletterande proteiner som exakt parar med varandra med samma kemiska DNA -språk.
"Det här är ett första genombrott i sitt slag, "Sa Chen." Det vi gör är att beräkningsdesigna dessa vätebindningsnätverk så att varje proteinpar har en unik komplementär sekvens. Det finns bara ett sätt för dem att komma ihop och de korsreagerar inte med proteiner från andra par. "
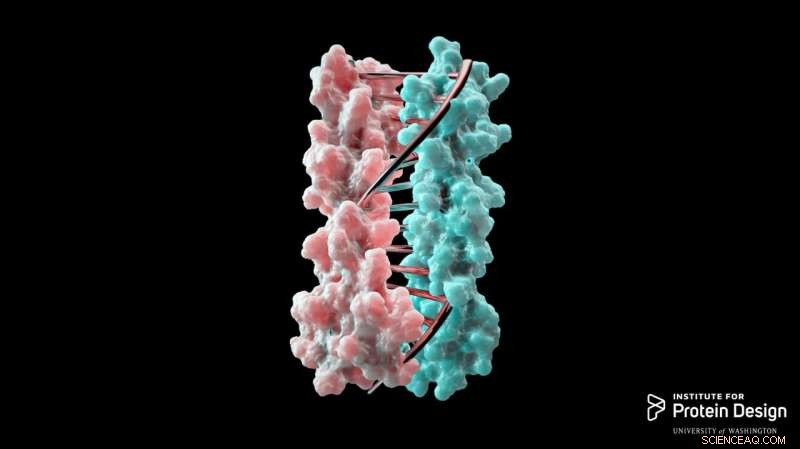
Proteiner som är utformade i laboratoriet kan nu zipas ihop på ungefär samma sätt som DNA -molekyler zipas upp för att bilda en dubbel helix. Tekniken kan möjliggöra design av proteinnanomaskiner som potentiellt kan hjälpa till att diagnostisera och behandla sjukdomar, möjliggöra mer exakt konstruering av celler och utföra en mängd andra uppgifter. Denna teknik ger forskare en exakt, programmerbart sätt att styra hur proteinmaskiner interagerar. Kredit:Institute for Protein Design
"Tekniska celler för att utföra nya uppgifter är framtiden för medicin och bioteknik, oavsett om det är att konstruera bakterier för att göra energi eller städa upp giftigt avfall eller skapa immunceller som angriper cancer, "sa Scott Boyken, en annan författare till uppsatsen och postdoktor vid Institute for Protein Design. "Denna teknik ger forskare en exakt, programmerbart sätt att styra hur proteinmaskiner interagerar, ett viktigt steg mot att uppnå dessa nya uppgifter. Vi har öppnat en stor dörr till proteinnanomaterialdesign. "
I deras studie, forskare använde ett datorprogram utvecklat i Baker -labbet som heter Rosetta. Programmet drar fördel av det faktum att den form en aminosyrakedja kommer att anta drivs av attraktionskrafterna och avstötningen mellan aminosyrorna i kedjan och vätskan i vilken kedjan är nedsänkt. Genom att beräkna den form som bäst balanserar dessa krafter så att kedjan uppnår sin lägsta totala energinivå, programmet kan förutsäga vilken form en given aminosyrakedja sannolikt kommer att ta.