Kredit:Srividya Kumar
Forskare vid Indian Institute of Science (IISc) har utvecklat en metod för att snabbt identifiera och kontrollera om en sjukdomsframkallande bakterie är levande eller död.
Den använder Raman -spektroskopi, en teknik som vanligtvis används för att identifiera kemiska bindningar i material, att känna igen bakterier och kontrollera deras livsduglighet, eller deras tillstånd att vara vid liv.
"Det unika med denna studie är metodens snabbhet och känslighet, och dess potential att modifieras till en sängkant, bordsenhet för diagnos, " säger Srividya Kumar, en tidigare doktorand student vid institutionen för oorganisk och fysikalisk kemi, och första författare till studien publicerad i tidskriften Analytisk och bioanalytisk kemi .
Snabb upptäckt av det sjukdomsframkallande medlet eller patogenen, samt att kontrollera om det är levande eller inte i patientprover är nyckeln till behandling av infektionssjukdomar. Att identifiera patogenens livsduglighet hjälper också läkare att bestämma dosen av antibiotika som ska förskrivas, och minskar risken för överförskrivning som kan leda till antibiotikaresistens.
Smittsamma bakterier identifieras vanligtvis genom tekniker som odling - odling av dem på ett näringsmedium i en petriskål. Det kan ta ungefär två till tre dagar att spåra deras tillväxt och bekräfta om de är döda eller levande. Dessutom, bakterier som är svåra att odla i labbkulturer blir ofta oupptäckta med konventionella metoder. Mer sofistikerade metoder som Polymerase Chain Reaction (PCR) skapar en genetisk profil av mikroben, men kan inte säga om den är levande eller död.
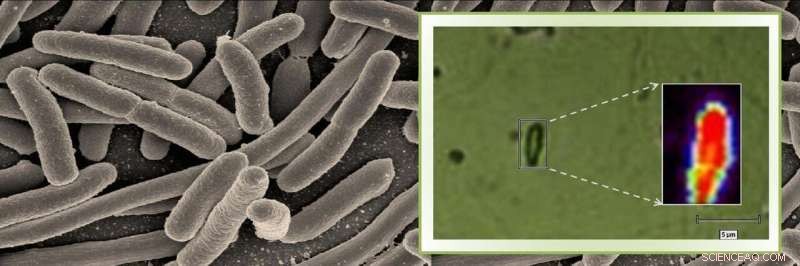
Ramanspektroskopi används i stor utsträckning inom kemiområdet för att undersöka strukturen hos molekyler. I den här studien, dock, Kumar och hennes kollegor har kombinerat detta tillvägagångssätt med avancerad mikroskopi för att kontrollera förekomsten av bakterier. De använde denna teknik för att upptäcka om tuberkulosbakterien (Mycobacterium tuberculosis) fanns i ett provmedium som liknar sputum. De testade också prover som innehöll fem andra mikrober.
Med hjälp av mikrospektroskopet, forskarna studsade en laserstråle från provet och fångade ljuset som spreds av provet i form av ett spektrum, som varierar med bakteriens biokemiska sammansättning. Varje bakterieart genererar ett unikt spektrum med specifika värden för intensitet och våglängdsposition för det spridda ljuset, beroende på vilken typ av kemiska bindningar som finns inuti bakteriecellen. Tekniken var tillräckligt känslig för att generera ett spektrum även för en enda bakteriecell.
Materialet som används för att montera bakterierna i denna teknik, känt som substratet, är också avgörande eftersom vissa substrat kan lägga till buller till den biokemiska profilen. Forskarna utvecklade en neutral, aluminiumbaserat tunnfilmssubstrat som inte genererar bakgrundssignaler som kan störa spektrat.
Med denna metod, forskarna kunde inte bara identifiera bakterien utan kunde också skilja mellan levande och döda bakterieceller - eftersom de två kemiska sammansättningen skiljer sig åt - inom två till tre timmar efter provtagning.
Eftersom varje bakterieart ger upphov till ett unikt spektrum, det kan vara möjligt att så småningom bygga en databas med patogener som kan användas i klinisk diagnos.
"Även om vi har validerat metoden för tuberkulos, metodiken kan utvidgas till alla typer av bakterieinfektioner, " säger Deepak Saini, docent vid Institutionen för molekylär reproduktion, Utveckling och genetik, IISc, och en av de äldre författarna till studien.