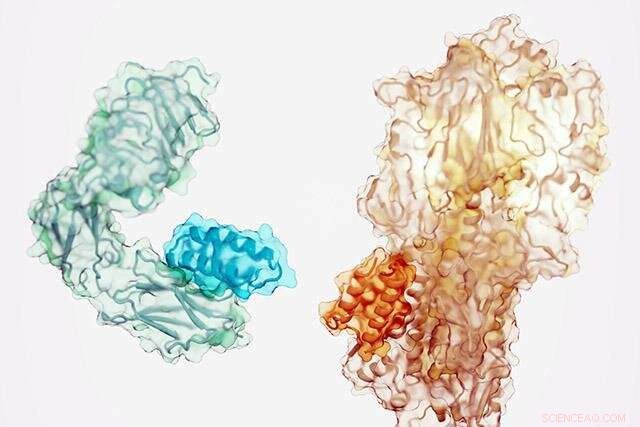
Små proteiner (mörkare nyans) utformade för att binda till insulinreceptorn (vänster) och en komponent av influensaviruset (höger). Kredit:Ian C. Haydon/Institute for Protein Design
Ett team av forskare har skapat en kraftfull ny metod för att generera proteinläkemedel. Med hjälp av datorer designade de molekyler som kan rikta in sig på viktiga proteiner i kroppen, som insulinreceptorn, samt sårbara proteiner på virusytan. Detta löser en långvarig utmaning inom läkemedelsutveckling och kan leda till nya behandlingar för cancer, diabetes, infektion, inflammation och mer.
Forskningen, som visas idag i tidskriften Nature , leddes av forskare i laboratoriet av David Baker, professor i biokemi vid University of Washington School of Medicine och mottagare av 2021 års Breakthrough Prize in Life Sciences.
"Förmågan att generera nya proteiner som binder tätt och specifikt till alla molekylära mål som du vill är ett paradigmskifte i läkemedelsutveckling och molekylärbiologi mer allmänt", säger Baker.
Antikroppar är dagens vanligaste proteinbaserade läkemedel. De fungerar vanligtvis genom att binda till ett specifikt molekylärt mål, som sedan antingen aktiveras eller inaktiveras. Antikroppar kan behandla ett brett spektrum av hälsorubbningar, inklusive covid-19 och cancer, men att skapa nya är en utmaning. Antikroppar kan också vara kostsamma att tillverka.
Ett team ledd av två postdoktorala forskare i Baker-labbet - Longxing Cao och Brian Coventry - kombinerade de senaste framstegen inom området för beräkningsproteindesign för att komma fram till en strategi för att skapa nya proteiner som binder molekylära mål på ett sätt som liknar antikroppar. De utvecklade mjukvara som kan skanna en målmolekyl, identifiera potentiella bindningsställen, generera proteiner som riktar sig mot dessa platser och sedan screena från miljontals bindande kandidatproteiner för att identifiera de som mest sannolikt kommer att fungera.
Teamet använde den nya programvaran för att generera bindande proteiner med hög affinitet mot 12 distinkta molekylära mål. Dessa mål inkluderar viktiga cellulära receptorer som TrkA, EGFR, Tie2 och insulinreceptorn, samt proteiner på ytan av influensaviruset och SARS-CoV-2 (viruset som orsakar COVID-19).
"När det kommer till att skapa nya läkemedel finns det lätta mål och det finns hårda mål", säger Cao, som nu är biträdande professor vid Westlake University. "I det här dokumentet visar vi att även mycket hårda mål är mottagliga för detta tillvägagångssätt. Vi kunde göra bindande proteiner till vissa mål som inte hade några kända bindningspartners eller antikroppar,"
Totalt producerade teamet över en halv miljon kandidatbindande proteiner för de 12 utvalda molekylära målen. Data som samlats in om denna stora pool av kandidatbindande proteiner användes för att förbättra den övergripande metoden.
"Vi ser fram emot att se hur dessa molekyler kan användas i ett kliniskt sammanhang, och ännu viktigare hur denna nya metod för att designa proteinläkemedel kan leda till ännu mer lovande föreningar i framtiden", säger Coventry. + Utforska vidare