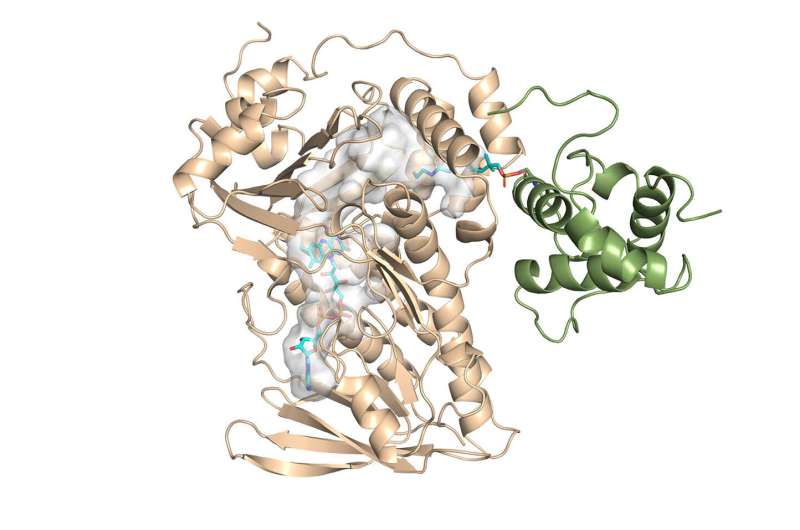
Kemister har för första gången fastställt kristallstrukturen för en nyckelkomponent i monensinenzymet och har låst upp mekanismen för dess reaktionsaktivitet.
"Huvudfyndet var den första kristallstrukturen för denna familj av enzymer", säger Chu-Young Kim, professor i biokemi vid University of Illinois Urbana-Champaign (UIUC), som ledde den experimentella sidan av studien. Han och kollegor löste kristallstrukturen för MonCI, ett nyckelenzym i jordbakterier som naturligt syntetiserar monensin.
Lela Vukovic, docent vid University of Texas, El Paso (UTEP), utförde beräkningsstudier av monensinforskningen, publicerade i Nature Communications .
Initiativet University of Texas Research Cyberinfrastructure (UTRC) tilldelade Vukovic superdatortilldelningar på Lonestar6-systemet vid Texas Advanced Computing Center (TACC) för att möta dessa utmaningar. UTRC tillhandahåller avancerade beräkningsmöjligheter till forskare från alla 14 UT System-institutioner.
TACC:s Lonestar6 hjälpte till att avslöja reaktionssekvensen som producerar monensin. Denna forskning öppnar dörren för framtida design av säkrare, effektivare antibiotika.
"Vi fann att MonCI används för att utföra tre avgörande epoxidationsreaktioner," sa Kim. "Detta är mycket ovanligt och har konsekvenser för hur vi kan konstruera bakterien för att producera nya antibiotika."
Kim, som nyligen lämnade UTEP för att gå med i UIUC, konsulterade Vukovics labb med strukturresultaten och full av nya frågor. Det han hittade var en intressant sekventiell reaktion inuti enzymet. Det var dock fortfarande experimentellt omöjligt att erhålla enzymets kristallstruktur med substratet inuti i dess aktiva läge.
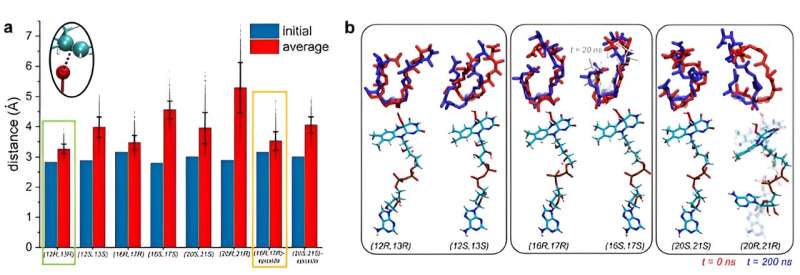
Det var då Kim och Vukovic bestämde sig för att modellera enzymet och substaten i simuleringar när substratet är stabilt. Om ett substrat är stabilt i en viss position, kan en reaktion inträffa för den positionen.
Vukovic och hennes elever Tara Nitka och Anju Yadav utvecklade fullständiga modeller av systemet med cirka 78 000 atomer baserade på de kristallstrukturer som bestämts av studieexperimentalisterna.
"De beräkningsmässiga utmaningarna uppstod från att undersöka flera system för att bestämma den position i vilken premonensin A och dess epoxiderade versioner är mest stabila och mest sannolikt att genomgå första, andra och tredje epoxidationsreaktioner," sa Vukovic.
"Superdatorer har varit mycket användbara för att karakterisera dessa biologiska molekyler som naturligt producerar antibiotika," tillade Vukovic. "Vi skulle inte kunna utföra beräkningsstudierna utan dem. Beräkningsstudier hjälper oss att upptäcka och förstå dessa komplexa sekventiella reaktioner som är viktiga för samhället."
Vukovic utförde sin postdoktorala forskning vid UIUC under avlidne Klaus Schulten, vars arv lever vidare i NAMD-mjukvaran som hans grupp utvecklade och användes i denna studie och otaliga andra.
"UIUC gör mycket arbete för att optimera NAMD för att köras på landets superdatorer, som Lonertar6 och Stampede2 vid TACC," sa hon. "NAMD tillät oss att zooma in på detta enzym och se vilka reaktioner som sker först, andra och tredje för att generera monensin."
"Monensin biosyntes kräver minst 14 olika enzymer, varav en är MonCI," tillade Kim. "Vi måste också undersöka alla andra enzymer. I framtiden förväntar vi oss att generera förbättrade versioner av monensin för att bättre ta hand om nötkreatur och fjäderfä. Dessutom är monensin giftigt för hästar och hundar, så dessa husdjur förgiftas ibland av misstag. och dödade. Därför behövs en giftfri monensin."
Författarna är Qian Wang, Tara A. Nitka, Anju Yadav och Lela Vukovic från University of Texas i El Paso; Ning Liu, Hongli Xiao, Hui Yang och Xi Chen från Northwest University, Kina; Irimpan I. Mathews från SLAC National Accelerator Laboratory; och Chu-Young Kim från University of Illinois Urbana-Champaign.
Mer information: Qian Wang et al, Triepoxidbildning genom ett flavinberoende monooxygenas i monensinbiosyntes, Nature Communications (2023). DOI:10.1038/s41467-023-41889-0
Tillhandahålls av University of Texas i Austin