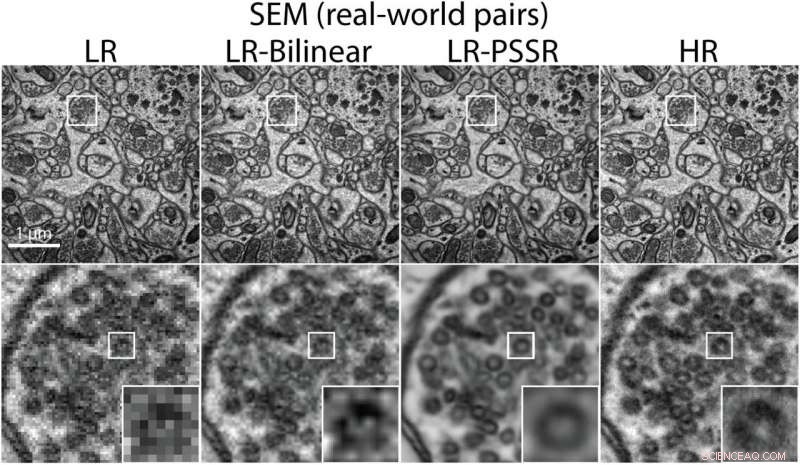
En bild som visar sida vid sida versioner av elektronmikroskopfångst. Kredit:Salk Institute
Läroboksbeskrivningar av hjärnceller får neuroner att se enkla ut:en lång ryggradsliknande central axon med förgrenade dendriter. Taget individuellt, dessa kan vara lätta att identifiera och kartlägga, men i en verklig hjärna, de är mer som en knotig hög med bläckfiskar, med hundratals lemmar sammanflätade. Detta gör att förstå hur de beter sig och interagerar till en stor utmaning för neuroforskare.
Ett sätt att reda ut vårt neurala virrvarr är genom mikroskopisk avbildning. Genom att ta fotografier av mycket tunna lager av en hjärna och rekonstruera dem i tredimensionell form, det är möjligt att avgöra var strukturerna finns och hur de förhåller sig.
Men detta medför sina egna utmaningar. Få högupplösta bilder, och fånga dem snabbt för att täcka en rimlig del av hjärnan, är en stor uppgift.
En del av problemet ligger i de avvägningar och kompromisser som alla fotografer är bekanta med. Öppna bländaren tillräckligt länge för att släppa in mycket ljus och alla rörelser kommer att orsaka en oskärpa; ta en snabb bild för att undvika oskärpa och motivet kan bli mörkt.
Men andra problem är specifika för de metoder som används vid hjärnrekonstruktion. För en, Högupplöst hjärnavbildning tar oerhört lång tid. För en annan, i den allmänt använda tekniken som kallas seriell blockansiktselektronmikroskopi, en bit vävnad skärs till ett block, ytan avbildas, en tunn sektion skärs bort och blocket avbildas sedan igen; processen upprepas tills den är klar. Dock, elektronstrålen som skapar de mikroskopiska bilderna kan faktiskt få provet att smälta, förvränger motivet som den försöker fånga.
Uri Manor, chef för Waitt Advanced Biophotonics Core Facility vid Salk Institute for Biological Studies i San Diego, är ansvarig för att köra ett flertal kraftfulla mikroskop som används av forskare över hela landet. Han har också i uppdrag att identifiera och distribuera nya mikroskop och utveckla lösningar som kan ta itu med problem som dagens teknologier brottas med.
"Om någon kommer med ett problem och våra instrument inte kan göra det, eller vi kan inte hitta en som kan, det är mitt jobb att utveckla den förmågan, sa Manor.
Medvetna om de avbildningsproblem som neuroforskare står inför, han beslutade att ett nytt tillvägagångssätt var nödvändigt. Om han hade nått de fysiska gränserna för mikroskopi, Manor resonerade, kanske bättre mjukvara och algoritmer kan ge en lösning.
"Det finns sofistikerade matematiska och beräkningsmetoder som har studerats i decennier för att ta bort brus utan att ta bort signal, sa Manor. Det var där jag började.
Arbetar med Linjing Fang, en bildanalysspecialist på Salk, de utarbetade en strategi för att använda GPU:er (grafiska bearbetningsenheter) för att påskynda mikroskopisk bildbehandling.
De började med ett bildbehandlingsknep som heter deconvolution som delvis hade utvecklats av John Sedat, en av Manors vetenskapliga hjältar och en mentor på Salk. Tillvägagångssättet användes av astronomer som ville lösa bilder av stjärnor och planeter med högre upplösning än de kunde uppnå direkt från teleskop.
"Om du känner till de optiska egenskaperna hos ditt system, då kan du sudda ut dina bilder och få dubbelt så hög upplösning som originalet, " han förklarade.
De trodde att djupinlärning – en form av maskininlärning som använder flera lager av analys för att successivt extrahera funktioner på högre nivå från rådata – kunde vara mycket användbar för att öka upplösningen i mikroskopbilder, en process som kallas superupplösning.
MRI, satellitbilder, och fotografier hade alla fungerat som testfall för att utveckla djupinlärningsbaserad, superupplösningsmetoder, men anmärkningsvärt lite hade gjorts i mikroskopi. Kanske, Manor tänkte, samma sak kan göras med mikroskopi.
Det första steget i att träna ett system för djupinlärning innebär att hitta en stor mängd data. För detta, Manor slog sig ihop med Kristen Harris, en neurovetenskapsprofessor vid University of Texas i Austin och en av de ledande experterna inom hjärnmikroskopi.
"Hennes protokoll används över hela världen. Hon gjorde öppen vetenskap innan det var coolt, "Sade Manor. "Hon får otroligt detaljerade bilder och har samarbetat med Salk i ett antal år."
Harris erbjöd Manor så mycket data som han behövde för träning. Sedan, med Maverick-superdatorn vid Texas Advanced Computing Center (TACC) och flera dagar av kontinuerlig beräkning, han skapade lågupplösta analoger av högupplösta mikroskopbilder och tränade ett djupinlärningsnätverk på dessa bildpar.
"TACC har varit otroligt hjälpsam, Manor sa. "De försåg oss med hårdvara för att träna innan vårt hår föll av och försåg oss med beräkningsexpertis och hjälpte till och med att köra beräkningsexperiment för att finjustera vår process."
Tyvärr, Manors första försök att skapa superupplösta versioner av lågupplösta bilder misslyckades. "När vi försökte testa systemet på verkliga lågupplösta data som var mycket bullrigare än våra lågupplösta träningsdata, nätverket gick inte så bra."
Manor hade ännu ett lyckokast när Jeremy Howard, grundare av fast.ai, och Fred Monroe, från Wicklow AI Medical Research Initiative (WAMRI.ai), kom till Salk och letade efter forskningsproblem som kunde dra nytta av djupinlärning.
"De var entusiastiska över vad vi gjorde. Det var en perfekt applikation för deras djupinlärningsmetoder och deras önskan att hjälpa till att föra djupinlärning till nya domäner, " Minns Manor. "Vi började använda några av deras knep som de hade etablerat, inklusive skräp."
Vid tidpunkten för deras möte, Manor och Fang hade beräkningsmässigt minskat upplösningen på sina bilder för träningspar, men de var fortfarande inte tillräckligt dåliga. De använde också en typ av djupinlärningsarkitektur som kallas generativa motstridiga nätverk (GAN).
"De föreslog att man skulle lägga till mer brus beräkningsmässigt, "mindes han. "'Släng in lite oskärpa, och olika typer av ljud, för att göra bilder riktigt taskiga.' De hade byggt ett bibliotek med smutskastningar och vi snickrade våra bilder tills det såg mycket mer ut som, eller ännu värre än, hur det ser ut när du skaffar en lågupplöst bild i världen. De hjälpte oss också att byta bort från GAN till U-Net-arkitekturer, som är mycket lättare att träna och bättre på att ta bort oljud."
Manor tränade om sitt AI-system med hjälp av de nya bildparen och djupinlärningsarkitekturen och fann att det kunde skapa högupplösta bilder som var väldigt lika de som ursprungligen hade skapats med större förstoring. Dessutom, Utbildade experter kunde hitta hjärncellsdrag i decrapparerade versionerna av lågupplösta prover som inte kunde detekteras i originalen.
Till sist, de testade sitt system på riktigt:att tillämpa metoden på bilder skapade i andra labb med olika mikroskop och preparat.
"Vanligtvis i djupinlärning, du måste omskola och finjustera modellen för olika datamängder, "Men vi var glada över att vårt system fungerade så bra för ett brett utbud av prov- och bilduppsättningar."
Framgången innebar att prover kunde avbildas utan att riskera skada, och att de kunde erhållas minst 16 gånger så snabbt som traditionellt gjorts.
"Att avbilda hela hjärnan i full upplösning kan ta över hundra år, " Manor förklarade. "Med en 16 gånger ökning i hela, det kanske blir 10 år, vilket är mycket mer praktiskt."
Teamet publicerade sina resultat i Biorxiv, presenterade dem på F8 Facebook Developer Conference och den andra NSF NeuroNex 3DEM Workshopen, och gjorde koden tillgänglig via GitHub.
"Det här tillvägagångssättet fungerar inte bara. Men vår träningsmodell kan användas direkt, ", sa Manor. "Det är extremt snabbt och enkelt. Och alla som vill använda det här verktyget kommer snart att kunna logga in på 3DEM.org [en webbaserad forskningsplattform fokuserad på att utveckla och sprida ny teknik för tredimensionell elektronmikroskopi med förbättrad upplösning, stöds av National Science Foundation] och kör deras data genom den."
"Uri främjar verkligen denna idé om bildförbättring genom djupinlärning, " sa Harris. "I slutändan, vi hoppas att vi inte får några fula bilder. Men just nu, många av bilderna har detta problem, så det kommer att finnas platser där du vill fylla i hålen baserat på vad som finns i de intilliggande sektionerna."
Manor hoppas kunna utveckla mjukvara som kan göra rekonstruktion i farten, så att forskare kan se superupplösta bilder direkt, snarare än vid efterbearbetning. Han ser också potentialen för att förbättra prestandan hos de miljontals mikroskop som redan finns på labb runt om i världen och för att bygga ett helt nytt mikroskop från grunden som drar fördel av AI-kapacitet.
"Billigare, högre upplösning, snabbare – det finns många områden som vi kan förbättra."
Med ett proof of concept på plats, Manor och hans team har utvecklat ett verktyg som möjliggör framsteg inom neurovetenskapen. Men utan slumpmässiga samarbeten med Kristen Harris, Howard och Monroe och TACC, det kanske aldrig har blivit verklighet.
"Det är ett vackert exempel på hur man verkligen gör framsteg inom vetenskapen. Du måste ha experter som är öppna för att arbeta tillsammans med människor från var i världen de än befinner sig för att få något att hända, " Sa Manor. "Jag känner mig bara så himla lyckligt lottad över att ha varit i en position där jag kunde ha kontakt med alla dessa lagkamrater i världsklass."