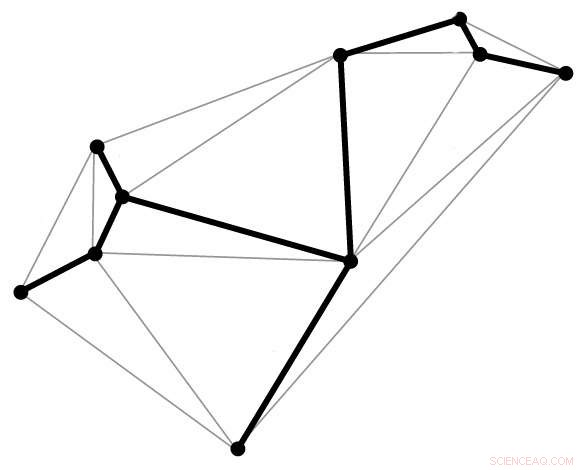
Den fetstilta linjen, känt som ett spannträd, följer den önskade geometriska formen, vidrör varje vertex bara en gång. En spännträdsalgoritm används i den nya DNA-origamimetoden för att kartlägga den rätta vägen för DNA-strängen. Kredit:Public Domain
Bland de värdefulla innehaven i Londons Wellcome Library finns en grov pennskiss gjord 1953 av Francis Crick. Ritningen är en av de första som visar dubbelspiralstrukturen hos DNA – naturens ritning för design av havssniglar, människor, och alla andra levande former på jorden.
Men få kunde ha förutsett att DNA:s enkla egenskaper för självmontering, och dess mångsidiga informationsbärande förmåga, skulle kunna användas på många sätt som Watson och Crick aldrig föreställt sig, (eller faktiskt, av naturen själv).
I ny forskning som dyker upp i tidskriftens förhandsupplaga på nätet Vetenskap , Mark Bathe, tillsammans med kollegorna Hao Yan från ASU och Wah Chiu från Baylor College of Medicine beskriver en ny metod för att designa geometriska former byggda från DNA. De presenterar en ny variant på en teknik som kallas DNA-origami, där DNA:s basparningsegenskaper utnyttjas för konstruktion av små strukturer i 2 och 3 dimensioner.
"En viktig utmaning inom området DNA-nanoteknik är att designa alla önskvärda strukturer på ett top-down sätt, utan mycket mänsklig input angående detaljer om DNA-strängens vikningsbanor, " säger Yan.
Hans medarbetare vid MIT, ledd av Mark Bathe, utvecklat en datoralgoritm för att designa DNA-nanostrukturer genom att endast mata in en målform. De konstruerade en mjukvaruplattform som kan beräkna och mata ut nödvändiga DNA-strängar för att bilda designerarkitekturer. Bildandet av dessa strukturer karakteriserades sedan systematiskt och bekräftades experimentellt vid de tre instituten. "Detta visar verkligen upp tvärvetenskaplig kollaborativ vetenskap över hela landet, " säger Yan
Yan leder Biodesign Center for Molecular Design and Biomimetics vid Arizona State University.
Andra världar
Teamet designar användbara strukturer i en häpnadsväckande liten skala. (En nanometer är en miljarddels meter eller ungefär lika stor som en sockermolekyl.) Specialiserade bildtekniker, inklusive atomkraft- och kryoelektronmikroskopi används för att visualisera de resulterande formerna.
Den förenklade tekniken som beskrivs lovar att avsevärt utöka användningen av DNA-origami utanför specialistgemenskapen och utöka utbudet av möjliga tillämpningar inom biomolekylär vetenskap och nanoteknik. Dessa inkluderar användningen av nanopartiklar för läkemedelsleverans och cellinriktning, konstruktion av robotar i nanoskala som kan utföra olika aktiviteter inom medicin och industri och design av skräddarsydda optiska enheter.
En av de mer spännande innovationerna vid horisonten involverar användningen av DNA som ett lagringsmedium - ett med retentionstider i miljontals år. (Ett enda gram DNA kan lagra cirka 700 terabyte information – en mängd motsvarande 14, 000 50-gigabyte Blu-ray-skivor. Ytterligare, ett sådant nukleinsyraminne skulle potentiellt kunna drivas med en bråkdel av den energi som krävs för andra alternativ för informationslagring.)
Formskiftande
Den nya designmetoden, som kan producera praktiskt taget vilken polyedrisk form som helst, förlitar sig på en top-down-strategi, som börjar med en kontur av den önskade formen och arbetar bakåt i steg för att definiera den erforderliga DNA-sekvensen som kommer att vikas ordentligt för att bilda den färdiga produkten.
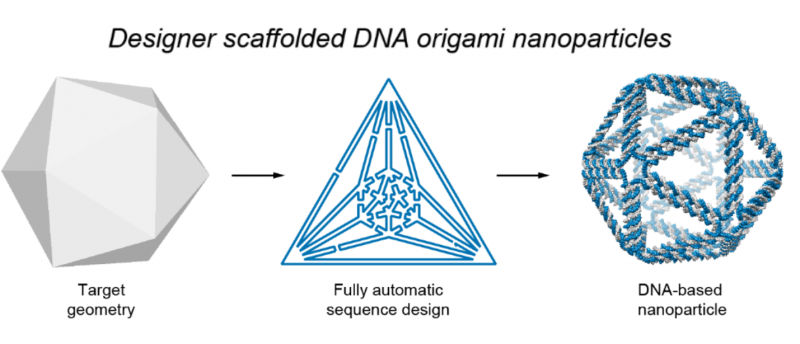
Illustrationen visar den grundläggande processen som används för att designa DNA-origami nanostrukturer. Först, en trådram av den avsedda måldesignen görs. Programvaran översätter sedan detta till en plan för routing av DNA-ställningar och stapelsträngar, som monteras för att bilda önskad form. Kredit:Biodesign Institute
Den autonoma processen utförs med hjälp av ett program som designats av författarna. Känd som DAEDALUS (för DNA Origami Sequence Design Algorithm for User-Defined Structures), Programmet utför invers design av godtyckliga DNA-origami nanoformer, baserat på ett inmatat trådramsnät, (en visuell representation av det stängda, 3-dimensionell geometrisk yta).
Programmet är inte bara användarvänligt, men mycket mångsidig, producera former som inte är begränsade till sfärisk topologi, (dvs. stängd, tvåsidiga strukturer utan gränser eller hål). När målformen har beskrivits som ett nätverk av noder och kanter, DNA-ställningssträngar av anpassad längd och sekvens genereras med hjälp av teknik som kallas asymmetrisk polymeraskedjereaktion.
Den nya studien beskriver tillverkningen av en mängd olika geometriska DNA-objekt, inklusive 35 polyedriska former (platoniska, Archimedean, Johnson och katalanska fasta ämnen) 6 asymmetriska strukturer, och fyra polyedrar med ickesfärisk topologi, använda omvända designprinciper. Metoden kan producera nanoformer med hög tillförlitlighet och stabilitet utan den normala mödosamma processen att manuellt utforma baspar för att bilda den avsedda målstrukturen.
Går in i vecket
DNA-origami för ned den urgamla japanska metoden för pappersvikning till molekylär skala. Grunderna är enkla:Ta en längd enkelsträngat DNA och styr det till en önskad form, fästa samman strukturen med kortare så kallade stapelsträngar, som binder på strategiska platser längs DNA:s längre längd. Metoden bygger på det faktum att DNA:s fyra nukleotidbokstäver - A, T, C, &G håller ihop på ett konsekvent sätt; Som alltid paras ihop med Ts och Cs med Gs.
DNA-molekylen i sin karakteristiska dubbelsträngade form är ganska styv, jämfört med enkelsträngat DNA, som är flexibel. Av denna anledning, enkelsträngat DNA ger ett idealiskt spetsliknande ställningsmaterial. Ytterligare, dess parningsegenskaper är förutsägbara och konsekventa, (till skillnad från RNA, som anses promiskuöst, på grund av baspar som kan vara oväntade).
Tekniken har visat sig mycket framgångsrik för att skapa otaliga former i 2- och 3-dimensioner, som bekvämt sätter sig själv när de designade DNA-sekvenserna blandas ihop. Den knepiga delen är att förbereda den rätta DNA-sekvensen och routingdesignen för byggnadsställningar och stapelsträngar för att uppnå den önskade målstrukturen. Vanligtvis, detta är mödosamt arbete som måste utföras manuellt.
Med den nya tekniken, målstrukturen beskrivs först i termer av ett trådnät som består av polyedrar. Från detta, en spännträdsalgoritm genereras. Detta är i grund och botten en karta som automatiskt kommer att styra ruttningen av DNA-ställningssträngen genom hela origamistrukturen, beröra varje vertex i geometrisk form, en gång. Kompletterande klammersträngar tilldelas sedan och den slutliga formen monteras själv.
För att testa metoden, enklare former kända som platoniska fasta ämnen tillverkades först, följt av allt mer komplexa strukturer. Dessa inkluderade objekt med ickesfärisk topologi och ovanliga interna detaljer, som aldrig tidigare hade förverkligats experimentellt.
De färdiga designerna visade uppifrån-och-ned-teknikens förmåga att automatiskt generera ställnings- och häftrutter för ett omfattande utbud av nanoformer, baserat enbart på ytgeometri. Cryo-EM användes för att bekräfta strukturell trohet och stabilitet hos de sammansatta origamistrukturerna.
Ytterligare experiment bekräftade att de framställda DNA-strukturerna var potentiellt lämpliga för biologiska tillämpningar eftersom de uppvisade långtidsstabilitet i serum- och lågsaltförhållanden.
Forskningen banar väg för utveckling av designade system i nanoskala som efterliknar egenskaperna hos virus, fotosyntetiska organismer och andra sofistikerade produkter av naturlig evolution.
Förutom sin anställning vid Biodesign Institute, Hao Yan är Milton D. Glick Distinguished Professor, College of Liberal Arts and Sciences, Skolan för molekylära vetenskaper vid ASU.