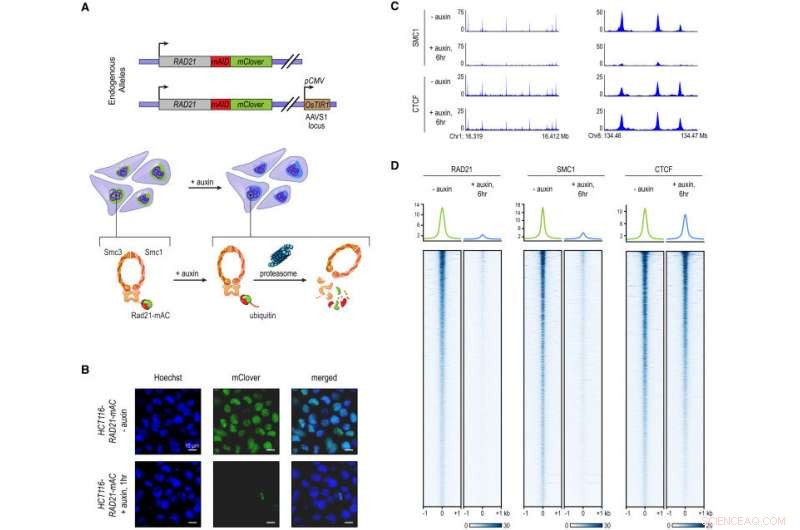
Märkning av endogen RAD21 med en auxininducerbar degron möjliggör snabb, Nära komplett Cohesin -förlust. (A) I HCT-116-RAD21-mAC-celler, båda RAD21-alleler är märkta med auxininducerbara degroner och en mClover-reporter, och OsTIR1 -genen är integrerad på AAVS1 -locuset. Auxinbehandling leder till proteasomal nedbrytning av RAD21. (B) Levande cellavbildning efter Hoechst 33342 -färgning för att märka kärnor. Kärn -mClover -fluorescens motsvarande märkt RAD21 förlorades efter 1 timme med auxinbehandling. (C) SMC1 och CTCF ChIP-seq-signal med och utan auxinbehandling. (D) RAD21, SMC1, och CTCF ChIP-seq-signal (vänster, mitten, till höger) över alla toppar som kallas för vart och ett av proteinerna i obehandlade RAD21-mAC-celler. Överst:Genomsnittliga berikningar för varje protein. Efter nedbrytning av RAD21, kohesinkomplexet binder inte längre till kromatin. CTCF -bindning påverkas inte. Kreditera: Cell . DOI:10.1016/j.cell.2017.09.026
Ett multinstitutionellt team som spänner över Baylor College of Medicine, Rice University, Stanford University och Broad Institute of MIT och Harvard har skapat den första högupplösta 4-D-kartan över genomviktning, spåra ett helt mänskligt genom när det viks över tiden. Rapporten, vilket kan leda till nya sätt att förstå genetiska sjukdomar, visas på omslaget till Cell .
Anslutningar
I årtionden, forskare har misstänkt att när en mänsklig cell svarar på en stimulans, DNA -element som ligger långt ifrån varandra i genomet hittar snabbt varandra, bildar öglor längs kromosomen. Genom att omorganisera dessa DNA -element i rymden, cellen kan ändra vilka gener som är aktiva.
Under 2014, samma team av forskare visade att det var möjligt att kartlägga dessa slingor. Men de första kartorna var statiska, utan förmågan att se slingorna förändras. Det var oklart om, i kärnans trånga utrymme, DNA -element kan hitta varandra tillräckligt snabbt för att kontrollera cellulära svar.
"Innan, vi kunde göra kartor över hur genomet veckade sig när det var i ett visst tillstånd, men problemet med en statisk bild är att om ingenting någonsin förändras, det är svårt att komma på hur saker fungerar, "sa Suhas Rao, första författare till den nya studien. "Vår nuvarande strategi är mer som att göra en film; vi kan se veck när de försvinner och dyker upp igen."
En ring att härska över dem alla
För att spåra vikningsprocessen över tid, forskargruppen började med att störa cohesin, ett ringformat proteinkomplex som var beläget vid gränserna för nästan alla kända slingor. År 2015, laget föreslog att kohesin skapar DNA -slingor i cellkärnan genom en extruderingsprocess.
"Extrudering fungerar som remlängdsjusteraren på en ryggsäck, "förklarade Dr Erez Lieberman Aiden, chef för Center for Genome Architecture vid Baylor College of Medicine och seniorförfattare på den nya studien. "När du matar remmen genom vardera sidan, det bildar en slinga. DNA verkar göra samma sak - förutom att kohesinringar verkar spela rollen som justeraren. "
Aiden sa att en avgörande förutsägelse för 2015 -modellen är att alla slingor ska försvinna i avsaknad av kohesin. I den nya forskningen, Aiden, Rao och kollegor testade det antagandet.
"Vi fann att när vi störde sammanhållningen, tusentals slingor försvann, "sa Rao, en medicinsk student vid Stanford University, Hertz Fellow och medlem i Aiden lab. "Sedan, när vi sätter tillbaka cohesin, alla dessa slingor kom tillbaka - ofta på några minuter. Detta är exakt vad du skulle förutsäga från extruderingsmodellen, och det tyder på att den hastighet med vilken kohesin rör sig längs DNA är bland de snabbaste för något känt humant protein. "
Slingor kontra grupper
Men allt hände inte som forskarna förväntade sig. I vissa fall, loopar gjorde precis motsatsen till vad forskarna förväntade sig.
"När vi såg tusentals loopar över genomet bli svagare, vi märkte ett roligt mönster, "sa Aiden, också en McNair Scholar, Hertz Fellow och en senior utredare vid Rice Universitys centrum för teoretisk biologisk fysik. "Det var några udda slingor som faktiskt blev starkare. Sedan, när vi sätter tillbaka cohesin, de flesta slingor återhämtade sig helt - men dessa udda slingor gjorde igen motsatsen - de försvann! "
Genom att granska hur kartorna förändrades över tiden, laget insåg att extrudering inte var den enda mekanismen som för samman DNA -element. En andra mekanism, kallas fackalisering, involverade inte kohesin.
"Den andra mekanismen vi observerade skiljer sig helt från extrudering, "förklarade Rao." Extrudering tenderar att föra ihop DNA -element två åt gången, och bara om de ligger på samma kromosom. Denna andra mekanism kan ansluta stora grupper av element till varandra, även om de ligger på olika kromosomer. Och det verkar vara lika snabbt som extrudering. "
Broad Institute Director Eric Lander, en medförfattare till studien, sa, "Vi börjar förstå reglerna för hur DNA -element samlas i kärnan. Nu när vi kan spåra elementen när de rör sig över tiden, de bakomliggande mekanismerna börjar bli tydligare. "