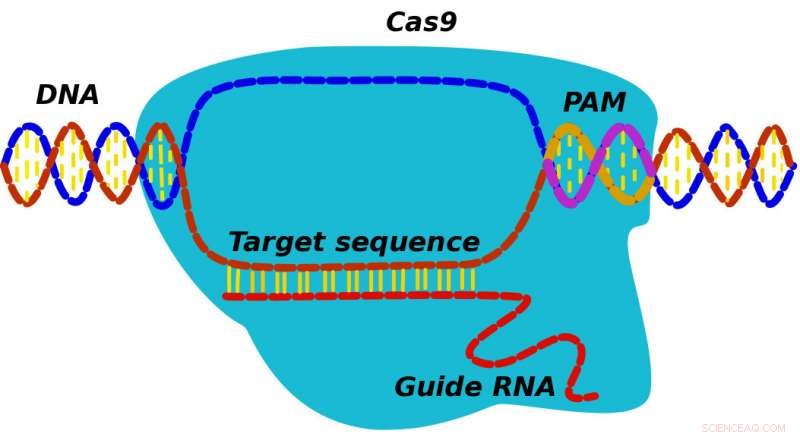
När ett CRISPR-Cas9-protein hittar sitt mål, den hittar först en spacer-sekvens känd som PAM och söker sedan efter intilliggande DNA för att se om det matchar Cas9s guide-RNA. En ny modell utvecklad av forskare vid Rice University kan hjälpa till att avslöja detaljer om mekanismen genom vilken CRISPR-Cas9 kan ersätta mutationer med korrekt DNA. Kredit:Alexey Shvets/Rice University
Rice University-forskare har utvecklat en beräkningsmodell för att kvantifiera mekanismen genom vilken CRISPR-Cas9-proteiner hittar sina genomredigerande mål.
Anatoly Kolomeisky, en risprofessor i kemi och kemisk och biomolekylär teknik, och alumnen Alexey Shvets anpassade ett system som de utvecklade tidigare för att visa hur proteiner i allmänhet hittar sina biologiska mål. De hoppas att den reviderade modellen kommer att hjälpa till att låsa upp de återstående mysterierna med CRISPR.
I sitt naturliga tillstånd, CRISPR, som står för "klustrade, regelbundet mellanrum korta palindromiska upprepningar, " är den biologiska mekanismen genom vilken bakterier skyddar sig mot virusinfektioner. Bakterierna inkorporerar en kopia av det främmande DNA:t och bygger upp ett register över alla de som invaderar. De hänvisar till den posten när nya inkräktare upptäcks och använder den för att förstöra dem.
På senare år har forskare har börjat anpassa mekanismen för användning i genomredigering, som har potential att bota sjukdomar och förbättra organismer, inklusive människor. Men en stötesten har varit risken att CRISPR-Cas9-proteiner, ett av systemen som använder CRISPR-metoden, kommer att klippa och ersätta fel målsekvenser, införa mutationer.
Rismodellen som beskrivs i Biofysisk tidskrift fann det troligt att CRISPR-Cas9 lokaliserar bra mål mer effektivt när dessa redigeringar utanför målet tillåts ske, eftersom proteinerna inte slösar tid på att ta avstånd från off-targets för att fortsätta söka.
Det kanske är bra eller inte, men det är verkligen värt att studera, sa Kolomeisky.
"Felfrekvensen (utanför målet) är ibland 10-20 procent, " sa han. "Vi har två idéer om detta:En är att virus muterar mycket snabbt och kanske bakterier försöker skära mål som bara är något muterade som ett sätt att vara mer flexibel. Den andra är att det finns proteiner som kan rätta till misstag, så om det inte är många felsnitt, systemet kan tolerera dem.
Kolomeisky sa att hans modell är ett enkelt steg mot att ta reda på dynamiken i CRISPR-redigering. "CRISPR-Cas9 är den mest populära varianten eftersom den bara har ett protein och är enklare, biologiskt, att arbeta med, " han sa.
Rice-labbet utvecklade sin ursprungliga modell för att lära sig hur proteiner glider längs DNA för att hitta mål och utlösa processer som gentranskription. Kolomeisky noterade att CRISPR-pionjären Jennifer Doudna upptäckte att CRISPR-Cas9 inte söker på samma sätt. "Hon fann att det inte glider någonstans på DNA:t, " han sa.
Istället, enligt Doudna och hennes team, proteinet känner initialt igen tre-nukleotid PAM (för protospacer angränsande motiv) sekvenser som markerar platsen för potentiella mål. "CRISPR hittar och binder till PAM och sedan utforskar dess associerade RNA det intilliggande DNA för att se om detta är målet, " sa Kolomeisky. "Om det är det, proteinet börjar skära sig. Om inte, det löser sig och letar sig någon annanstans."
I Doudnas efterföljande experiment med PAM-sekvenser borttagna, CRISPR-Cas9-proteiner kunde inte hitta sina mål alls. Så PAMs har en viktig roll och är inte bara en generisk spacer, han sa. "Så fort jag läste detta, Jag förstod att vi kunde använda vår modell här också."
Den teoretiska modellen tittar på processer i första passagen - de som händer när ett system passerar en fysisk eller kemisk tröskel, som att hitta en relevant PAM - att spåra CRISPR-Cas9-proteiner som infogats i en cell när de först undersöker PAM-sekvenser och sedan, medan den är bunden till PAMs, sök efter DNA-målet som matchar Cas9:s RNA.
De fann att CRISPRs som undviker skärningar utanför målet genom att skilja sig från "fel" DNA tar längre tid att sätta sig än en som helt enkelt skär bort från målen. "Att gå till fel PAM tar tid, ", sa Kolomeisky. "Vår beräkning visar att CRISPR kan hitta riktiga mål snabbare när det ibland skär på fel ställen. Bråkdelen som går till rätt mål kan vara mindre, men du kommer att klippa dem så småningom.
"Det är en enkel modell och exakt lösbar, " sa Kolomeisky. "Om någon vill testa, modellen kan ge specifika förutsägelser och i vissa fall erbjuda trender för vad som bör observeras." Vad som fortfarande saknas i modellen är förmågan att se om RNA-nyckeln känner igen sitt mål samtidigt - binder till DNA på en gång - eller sekventiellt, nukleotid för nukleotid.
"Det mest imponerande med CRISPR är inte upptäckten av ett immunsystem i bakterier utan det faktum att detta har skapat en revolution inom bioteknik, eftersom det betyder att vi i vilken cell som helst kan skära vilket DNA som helst på en specifik plats, mycket exakt, " sade Kolomeisky. "Jag hoppas att vårt arbete kommer att stimulera mer grundläggande studier, eftersom jag gillar CRISPR-metoden väldigt mycket. Men jag är inte glad när folk tillämpar det utan att förstå hur det fungerar på molekylär nivå."
Shvets är nu postdoktor vid Massachusetts Institute of Technology. Kolomeisky är professor i kemi och i kemi- och biomolekylär teknik.