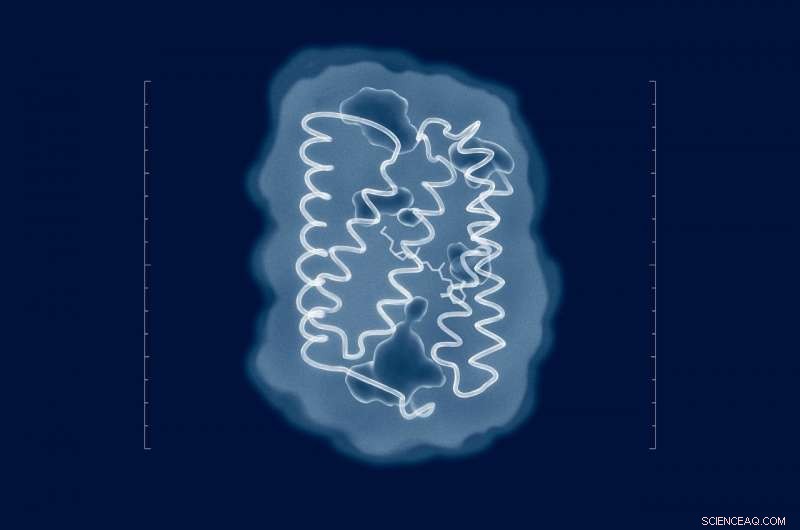
Försöken att lösa strukturen för ChR2 går ända tillbaka till tiden för dess upptäckt 2003. Men trots ansträngningarna från många forskargrupper från hela världen, proteinets struktur i dess naturliga tillstånd har förblivit okänd. Nu när forskare har strukturen, meningsfulla mutationer kan införas i proteinet för att anpassa dess egenskaper till kraven i ett specifikt experiment. Kredit:MIPT
Ett internationellt team av forskare har bestämt 3D-strukturen för kanalrodopsin 2, ett membranprotein som används i stor utsträckning inom optogenetik för att kontrollera nervceller med ljus. Optogenetik är en relativt ny teknik som innebär användning av ljus för att manipulera nerv- och muskelceller i en levande organism. Liknande tillvägagångssätt används för att delvis vända förlusten av hörsel och syn och kontrollera muskelsammandragningar.
Dessutom, optogenetikens metoder används för att studera egenskaperna hos naturliga neuronnätverk, som är ansvariga för känslor, beslutsfattande, och andra komplexa processer i levande organismer. Optogenetik var Natur s "Årets metod 2010, " samt att vara namngiven bland Vetenskap s "Beakthroughs of 2010 and Insights of the Decade."
Channelrhodopsin 2, eller ChR2, är ett viktigt optogenetiskt verktyg. Det är ett ljuskänsligt protein som ursprungligen extraherades 2003 från en grön alg som heter Chlamydomonas reinhardtii. Forskare kan infoga ChR2 i membranet av en levande cell för att kontrollera den. När den är upplyst, detta protein tillåter positivt laddade joner att passera in i cellen genom cellmembranet. I en nervcell, detta depolariserar membranet, efterliknar effekten av en nervimpuls och får just denna neuron att avfyras.
Eftersom ChR2 fungerar snabbt och är relativt ofarligt för celler, det är den aktuella lösningen för nervcellsaktivering. En rad artificiellt inducerade mutationer finns tillgängliga för att förändra proteinets egenskaper. Till exempel, det är möjligt att öka strömmen den genererar eller ändra våglängden på ljuset den reagerar på. Sådana modifieringar gör det möjligt för experimenterande att arbeta med proteiner som är skräddarsydda för deras behov. Forskare kan till och med kombinera flera proteinvarianter för ett distinkt svar vid olika våglängder av ljus.
De flesta av mutationerna som används för att modifiera egenskaperna hos ChR2 har hittills introducerats mer eller mindre slumpmässigt - antingen via riktad evolution eller baserat på data om kända proteinstrukturer. Det närmaste forskarna någonsin kommit en realistisk ChR2-struktur är en udda kombination som kallas C1C2, 70 procent av dessa är baserade ChR1, ett relaterat protein, med resten baserat på den faktiska ChR2. Denna blandade struktur kan inte förklara alla egenskaper hos proteinet. Som ett resultat, mutationerna som förutsägs av denna modell är inte riktigt realistiska och är därför av begränsat intresse för optogenetik.
För att avslöja strukturen för ChR2, författarna till studien använde en analytisk teknik som kallas röntgendiffraktion, som bara fungerar med prover i form av en kristall. Dessa erhölls av forskarna via in meso kristallisation. Det vill säga, proteinkristallerna odlades i den så kallade kubiska lipidmesofasen – ett medium som tillåter proteiner att röra sig fritt utan att lämna membranet. För att bestämma proteinstrukturer, deras kristaller bestrålades med röntgenstrålar vid en våglängd av cirka 1 ångström, vilket är något mindre än längden på bindningarna mellan atomerna i proteinet. I röntgenkristallografi, strukturer härleds genom att analysera hur strålning sprids av ett prov.
"Försöken att lösa strukturen för ChR2 går ända tillbaka till tiden för dess upptäckt 2003. Men trots ansträngningarna från många forskargrupper från hela världen, proteinets struktur i dess naturliga tillstånd har förblivit okänd, säger Valentin Borshchevskiy, en av författarna till uppsatsen och biträdande chef för Laboratory for Advanced Studies of Membrane Proteins vid MIPT. "Nu när vi har strukturen, meningsfulla mutationer kan införas i proteinet för att anpassa dess egenskaper till kraven i ett specifikt experiment. Att inte känna till strukturen, vi var tvungna att tråkigt räkna ut de användbara mutationerna genom att trial and error eller nöja oss med data om relaterade proteiner."