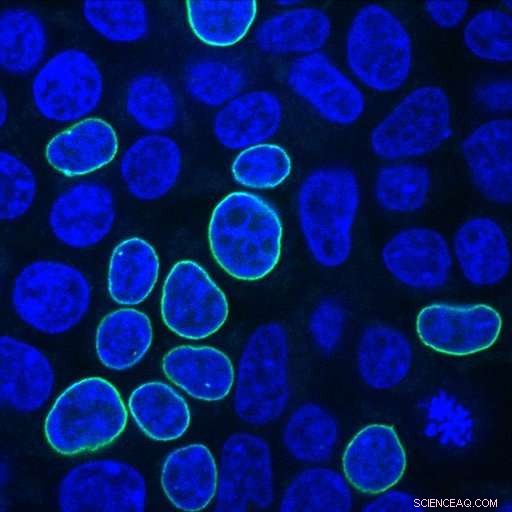
Mänskliga embryonala njurceller lyser grönt efter reparation av ett CRISPR-inducerat DNA-brott med ett PCR-fragment som kodar för ett fluorescerande protein och homologiarmar med 33 nukleotider. Kredit:Alexandre Paix
Johns Hopkins-forskare har utvecklat en strömlinjeformad metod och åtföljande effektivitets-"regler" för att introducera nya DNA-sekvenser i celler efter att ha använt genskärningsverktyget CRISPR. Forskarna säger att metoden, som de baserade på tester med musembryon och tusentals mänskliga celler, kan förbättra konsekvensen och effektiviteten av genomredigering.
Den nya metoden och dess utveckling beskrivs online i den 28 november i Proceedings of the National Academy of Sciences .
"CRISPR är ett verktyg för att hjälpa forskare att modifiera genomet, förutsäga resultatet av vissa egenskaper och studera dem, men själva verktyget skapar bara avbrott i arvsmassan. Det styr inte hur en ny DNA-sekvens infogas i genomet, säger Geraldine Seydoux, Ph.D., Huntington Sheldon professor i medicinsk upptäckt vid avdelningen för molekylärbiologi och genetik och vice dekan för grundforskning vid Johns Hopkins University School of Medicine, och en utredare vid Howard Hughes Medical Institute.
"Vi bestämde oss för att studera hur celler reparerar avbrott inducerade av CRISPR med målet att använda cellens naturliga DNA-reparationsprocess för att introducera nya sekvenser i genomet. Vi blev förvånade över att finna att celler lätt kommer att kopiera sekvenser från främmande DNA för att reparera DNA-avbrott. , så länge de främmande DNA:na är linjära, " tillägger Seydoux. "Genom att studera hur främmande DNA-fragment kopieras under reparationsprocessen, vi kom på några enkla regler för att göra genomredigering så effektiv som möjligt, optimera verktyget, och gör det med tillförsikt."
CRISPR, som står för klustrade, regelbundet mellanrum kort palindromisk upprepning, har vunnit popularitet bland forskare under de senaste fem åren som ett verktyg för att effektivt skära DNA. Det var anpassat för användning i däggdjursceller från en naturlig viral försvarsprocess i bakterieceller som involverar att skapa dödliga skärsår i viralt DNA. Väsentligen, verktyget är en strömlinjeformad uppsättning molekylära "saxar".
Den rådande tron, bland forskare, är att celler reparerar DNA-avbrott genom att infoga en slumpmässig uppsättning nukleotider, DNA:s kemiska byggstenar. Detta förstör vanligtvis alla gener som finns på platsen där DNA:t är brutet.
Det är också välkänt för forskare att ibland, celler använder en annan källa – en sekvens från en annan del av DNA, eller "donator"-DNA – för att försegla brottet i DNA. Dock, den nya "donator"-sekvensen kan inte infogas av sig själv i ett tomt utrymme i genomet.
Istället, det nya donator-DNA:t behöver en sorts tejp i varje ände för att hjälpa det att fästa i gapet som snittet gör. Forskare hänvisar till detta band som "homologi"-armarna hos donatorns DNA.
Homologiarmarna består av nukleotider som överlappar de intakta delarna av DNA:t med matchande genetisk kod. Detta hjälper donatorns DNA att "fastna" till det intakta DNA:t.
Än, forskare ansåg att använda donator-DNA som ett ineffektivt sätt att reparera genomet, antar att det krävde långa homologiarmar, speciellt när man infogar en lång DNA-sekvens, och enkelsträngat eller cirkulärt DNA, som är svåra att förbereda i långa storlekar.
Allt eftersom forskare fick mer erfarenhet av CRISPR, Seydoux säger, "Frågor uppstod om de optimala designreglerna för donator-DNA och längden på homologiarmarna."
Söker svar på dessa frågor, Johns Hopkins-forskarna infogade olika kombinationer av donator-DNA i mänskliga embryonala njurceller, kända för sin förmåga att växa bra och för att de ofta används i cancerforskning. Forskarna använde donator-DNA med en gen som kodar för ett fluorescerande protein, som lyser grönt i cellens kärnmembran när geninsättningen lyckas.
Johns Hopkins forskarassistent Alexandre Paix fann att linjära DNA-fragment fungerar mycket bra som donatorer, och är två till fem gånger effektivare än cirkulära DNA (kända som plasmider) i mänskliga celler. "Linjärt DNA är mycket lätt att förbereda i laboratoriet, använder PCR, säger Paix, hänvisar till verktyg för polymeraskedjereaktion, som används för att amplifiera DNA.
Paix testade också olika längder av homologiarmar. Han fann att sweet spot för homologiarmar är cirka 35 nukleotider långa, mycket kortare än vad forskare vanligtvis använder.
Specifikt, det visade sig att homologiarmar med 33 till 38 nukleotider långa var lika framgångsrika som de med 518 nukleotider, ger mellan 10 och 20 procent framgångsrika redigeringar under optimala förhållanden. I kontrast, när forskarna testade homologiarmar med 15 och 16 nukleotider långa, andelen framgångsrika insättningar sjönk med hälften. De upprepade dessa resultat på tre olika platser i det mänskliga genomet.
De fann också att den nyligen infogade sekvensen, utan att räkna homologiarmarna, kan vara upp till 1, 000 nukleotider långa.
Teamet uppnådde framgångsfrekvenser mellan 10 och 50 procent med inlägg som sträckte sig från 57 till 993 nukleotider långa. Kortare sekvenser infogades mer framgångsrikt än längre. Till exempel, nya sekvenser som var 57, 714 och 993 nukleotider långa infogades framgångsrikt 45,4, 23,5 och 17,9 procent av tiden, respektive. Bortom 1, 000 nukleotider, nya skär med 1, 122 och 2, 229 nukleotider hade liten framgång - cirka 0,5 procent av tiden. "I den storleken, det blir mycket svårt att introducera den mängd donator-DNA som behövs för redigering. Celler tenderar att "kvävas" av så mycket DNA, säger Seydoux.
Till sist, teamet fann också att framgångsfrekvensen för redigering toppar när den nya sekvensen är placerad inom 30 nukleotider från CRISPR-klippplatsen. "Utöver 30 nukleotider, insättningen fungerar inte, säger Seydoux.
"Dessa parametrar borde rymma de flesta gener som forskare försöker redigera. Faktum är att de flesta experiment involverar redigering av endast två till tre nukleotider nära CRISPR-snittplatsen, ", tillägger Seydoux.
Forskargruppen testade också om samma tillvägagångssätt kunde fungera i musembryon. Genom att använda ett PCR-fragment med 36-nukleotiders homologiarmar, teamet har framgångsrikt infogat en 739 nukleotider lång sekvens som kodar för ett fluorescerande protein i 27 av 87 (31 procent) musembryon.
Seydouxs forskargrupp använder redan reparationsreglerna för att studera DNA i Caenorhabditis elegans, en art av mask, och forskarna studerar om reparationsreglerna gäller andra typer av mänskliga celler.
Innan riktlinjerna antas allmänt, Seydoux säger att de borde testas i fler mänskliga celltyper och andra organismer.