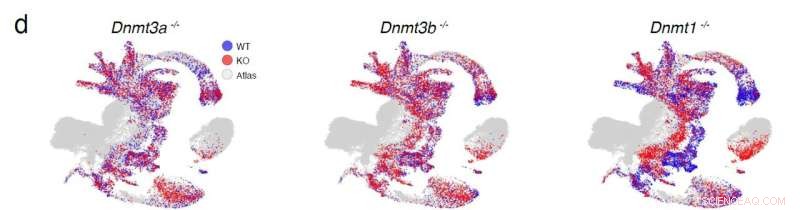
Kartläggning av celler från DNMT-knock-out-mösslinjerna till referensatlasen (grå) med vildtypsceller indikerade i blått och knock-out-celler indikerade i rött. Kredit:Babraham Institute
Forskning som tillämpar encellsanalystekniker och jämförelser med en cellatlasresurs gjorde det möjligt för forskare vid Babraham Institute att koppla observerade utvecklingsdefekter orsakade av störda DNA-metyleringsprocesser med en förståelse för de celltyper som påverkas. Detta arbete, publicerat i Genome Biology , bygger på tidigare arbete som utförts med medarbetare för att upprätta en detaljerad cellatlas som kartlägger cellöden genom tidig utveckling. Detta arbete bidrar med viktig ny kunskap för att förstå rollen av DNA-metylering under embryogenes, vilket hjälper till att dechiffrera reglerna som styr hur olika celltyper uppstår. En förståelse för dessa regler kommer att vara avgörande för att forskare ska kunna exakt och säkert styra cellödet för att producera kliniskt relevanta celltyper för regenerativ medicin.
Forskning från Reik labat, Babraham Institute, har ökat vår förståelse av rollen av DNA-metylering under de tidigare utvecklingsstadierna. Teknologiska framsteg inom detta område, som ger möjligheten att samla in parallella datatyper från en enda cell och förekomsten av cellatlasreferenser och omfattande datamängder, revolutionerar vad vi vet om processerna som avgör cellernas öde. Att klargöra reglerna för hur olika celltyper bildas har tillämpningar inom regenerativ medicin såväl som för att förstå utvecklingsstörningar och sjukdomar.
Radering och återinförande av DNA-metylering är känt för att vara avgörande för att etablera cellidentitet när embryots vävnader och organ bildas. Borttagning av viktiga metyleringsenzymer hos möss orsakar allvarliga utvecklingsdefekter och embryodödlighet i vissa fall. Trots vikten av DNA-metylering i utvecklingen är de underliggande mekanismerna för hur detta uppnås dåligt förstådda. Detta beror på begränsningar av den information som forskare tidigare kunde samla in för att förstå effekterna av förändringar i de vanliga processerna för DNA-metylering under utveckling, vilket var begränsat till analys av utvecklingsdefekter, provavbildning och begränsad genomomfattande analys med hjälp av bulkprover. Dessa metoder var inte tillräckliga för att lösa effekter på nivån för olika celltyper.
Med hjälp av möss där viktiga metyleringsenzymer raderades utförde forskare från Reik-labbet i institutets Epigenetics-program encellsgenexpressionsanalys i början av organutveckling, vilket inträffar dag 8,5 efter befruktning. Genom att använda kraften i encellsstrategier kunde forskarna följa vilka celltyper som påverkades, i termer av att de inte kunde bildas i musembryot, vilket tyder på mekanismerna bakom effekterna som ses i helorganismskala.
Forskningen använde genetiskt modifierade möss där två nyckelgrupper av metyleringsenzymer raderades:knock-out-mösslinjer där DNA-metyltransferaser (DNMT 1, 3a och 3b) som introducerar och upprätthåller DNA-metylering raderades individuellt, och ett system för att undersöka effekterna av en kombinerad deletion av alla tre TET-enzymer (tio-elva translokations (TET) metylcytosindioxygenaser) 1/2/3), som orsakar demetylering.
Dr. Stephen Clark, en senior forskare i Reik-labbet när denna forskning genomfördes, sa:"Användningen av encelliga metoder ger verkligen den upplösning vi behöver för att studera mekaniken för DNA-metylering under utvecklingen. Den bild vi kunde att bygga upp bekräftar den repressiva rollen av DNA-metylering vid denna utvecklingstidpunkt, för det första att upprätthållande av korrekt DNA-metylering krävs för att undertrycka tidigare och alternativa celltypsidentiteter, och för det andra att DNA-metylering måste avlägsnas från delar av genomet för att tillåta vissa celler typer att bilda."
En teknik som kallas encellig RNA-sekvensering användes för att mäta genuttryck över genomet i varje muslinje. Att jämföra dessa uttrycksprofiler med en referensdatauppsättning gjorde att alla celltyper av embryot kunde identifieras. Efter det steget kunde effekten av metyleringsstörningar på cellödet bedömas genom att jämföra sammansättningen av knock-out-embryona (där metyleringsenzymer raderades) med vildtypsembryon i samma utvecklingsstadium för att belysa skillnader i celltyp proportioner.
Forskarna kunde korrelera effekter på celltypsbildning på dag 8.5 av utvecklingen som matchade observerade fenotyper och analyserade celltypsspecifika förändringar i genuttryck som kunde kopplas till defekter i cellödets engagemang.
Dr. Ricard Argelaguet, en före detta postdoktor i Reik-labbet vid institutet och medförsta författare på uppsatsen, sa:"Förmågan att både ha hela organismens perspektiv och granulariteten i att observera förändringar i celltyper och genuttryck har gett oss möjligheten att reta isär rollen av DNA-metylering och demetylering i det utvecklande embryot vid denna speciella tidpunkt för att skapa nya insikter. Det kommer att vara lika intressant att tillämpa detta tillvägagångssätt på senare tidpunkter för att förstå mer om rollen av DNA-metylering allt eftersom utvecklingen fortskrider."
Forskningen har skapat en interaktiv dataplattform som ger genuttrycksavläsningar på encellsnivå från Dnmt- och Tet-mutantmöss-embryon.
Professor Wolf Reik, chef för Altos Cambridge Institute of Science, som ledde forskningen medan han var gruppledare i epigenetikprogrammet vid Babraham Institute, sa:"Denna forskning ger en rik resurs för att undersöka sambandet mellan DNA-metylering och etablering av Denna forskning drog nytta av publicerade datamängder och referensatlaser och vi hoppas att vårt arbete i sin tur kommer att vara till nytta för andra forskare inom både utvecklings- och epigenetikområdet." + Utforska vidare