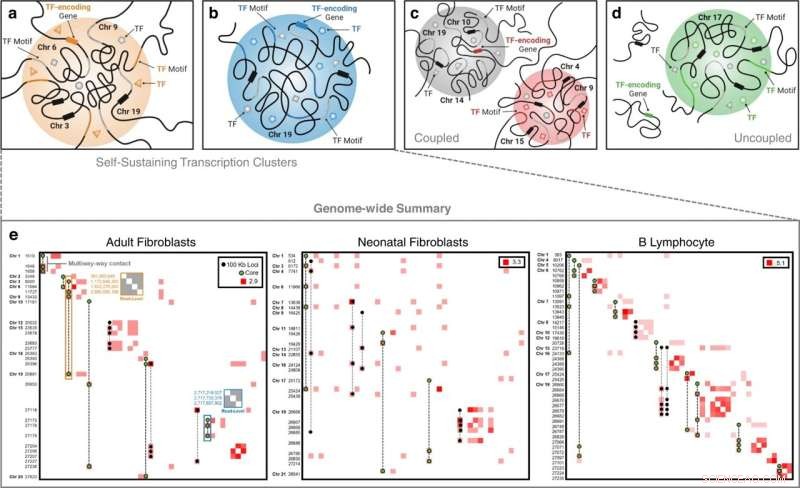
Klasser av transkriptionskluster. I ett självförsörjande transkriptionskluster finns både en TF och genen som kodar för den TF närvarande. De inter- och intrakromosomala exemplen i (a) respektive (b) illustrerar detta fenomen där vi i a ser TF av intresse (orange triangel) cirkulera vid klustret, dess bindningsmotiv finns på kromatinet (orange del). ) och dess motsvarande gen uttryckt (orange rektangel på kromosom 6). De grå formerna representerar ytterligare TF:er med bindningsmotiv (grå del av kromatin) vid klustret. Svarta rektanglar på kromosomerna 3, 9 och 19 representerar ytterligare gener som finns i klustret. c En analog-oberoende klass av transkriptionskluster där vi observerar en TF (röd kvadrat) binder till ett transkriptionskluster (rött kluster) och dess motsvarande gen uttryckt i ett separat transkriptionskluster (gråt kluster), men ändå inte i samma kluster. d En analogoberoende klass av transkriptionskluster där vi observerar en TF (grön cirkel) binder till ett transkriptionskluster (grönt kluster) och dess motsvarande gen uttryckt men inte inom ett transkriptionskluster. e Genomomfattande celltypspecifika självuppehållande transkriptionskluster extraherade från flervägskontaktdata och sönderdelade i Hi-C-kontaktmatriser vid 100 kb upplösning. Kontaktfrekvenser log-transformeras för bättre visualisering. Frekvenser längs diagonalen indikerar interaktion mellan två eller flera unika flervägsloki som faller inom samma 100 kb stora bin. Axeletiketter är icke sammanhängande 100 kb bin-koordinater i kromosomordning. Flervägskontakter som utgör de självförsörjande transkriptionsklustren är överlagrade. Flervägskontakter med grönfärgade loci representerar "kärn"-transkriptionskluster - transkriptionskluster som innehåller en masterregulator och dess genanalog. Ett exempel på läsnivåkontaktkarta för det interkromosomala FOXO3 självuppehållande transkriptionsklustret betecknas med den orange markerade rutan i vuxenfibroblastkontaktmatrisen och en läsnivåkontaktkarta för det intrakromosomala ZNF320 självuppehållande transkriptionsklustret är betecknas med den blå markerade rutan. Värden längs den vänstra axeln för dessa läsnivåkontaktmatriser är basparpositioner för kontaktställena i genomet. Kredit:Nature Communications (2022). DOI:10.1038/s41467-022-32980-z
Science fiction-författaren Arthur C. Clarkes tredje lag säger att "all tillräckligt avancerad teknik går inte att skilja från magi."
Indika Rajapakse, Ph.D., är en troende. Ingenjören och matematikern är nu biolog. Och han tror att skönheten i att blanda dessa tre discipliner är avgörande för att reda ut hur celler fungerar.
Hans senaste utveckling är en ny matematisk teknik för att börja förstå hur en cells kärna är organiserad. Tekniken, som Rajapakse och medarbetare testade på flera typer av celler, avslöjade vad forskarna kallade självförsörjande transkriptionskluster, en undergrupp av proteiner som spelar en nyckelroll för att upprätthålla cellidentitet.
De hoppas att denna förståelse kommer att avslöja sårbarheter som kan riktas mot att omprogrammera en cell för att stoppa cancer eller andra sjukdomar.
"Fler och fler cancerbiologer tror att genomorganisation spelar en stor roll för att förstå okontrollerbar celldelning och huruvida vi kan programmera om en cancercell. Det betyder att vi måste förstå mer detaljer om vad som händer i kärnan", säger Rajapakse, docent i beräkningsteknik. medicin och bioinformatik, matematik och biomedicinsk teknik vid University of Michigan. Han är också medlem i U-M Rogel Cancer Center.
Rajapakse är senior författare på tidningen, publicerad i Nature Communications . Projektet leddes av en trio doktorander med ett tvärvetenskapligt team av forskare.
Teamet förbättrade en äldre teknologi för att undersöka kromatin, kallad Hi-C, som kartlägger vilka delar av genomet som ligger nära varandra. Det kan identifiera kromosomtranslokationer, som de som förekommer i vissa cancerformer. Dess begränsning är dock att den bara ser dessa intilliggande genomiska regioner.
Den nya tekniken, kallad Pore-C, använder mycket mer data för att visualisera hur alla delar i en cells kärna interagerar. Forskarna använde en matematisk teknik som kallas hypergrafer. Tänk:tredimensionellt Venn-diagram. Det tillåter forskare att se inte bara par av genomiska regioner som interagerar utan helheten av de komplexa och överlappande genomomfattande relationerna inom cellerna.
"Det här multidimensionella förhållandet kan vi förstå entydigt. Det ger oss ett mer detaljerat sätt att förstå organisatoriska principer inuti kärnan. Om du förstår det kan du också förstå var dessa organisatoriska principer avviker, som i cancer," sa Rajapakse. "Det här är som att sätta samman tre världar - teknik, matematik och biologi - för att studera mer detaljer i kärnan."
Forskarna testade sin metod på neonatala fibroblaster, biopsierade vuxna fibroblaster och B-lymfocyter. De identifierade organisationer av transkriptionskluster specifika för varje celltyp. De hittade också vad de kallade självuppehållande transkriptionskluster, som fungerar som nyckeltranskriptionssignaturer för en celltyp.
Rajapakse beskriver detta som det första steget i en större bild.
"Mitt mål är att konstruera den här typen av bild över cellcykeln för att förstå hur en cell går igenom olika stadier. Cancer är okontrollerbar celldelning," sa Rajapakse. "Om vi förstår hur en normal cell förändras över tid kan vi börja undersöka kontrollerade och okontrollerade system och hitta sätt att omprogrammera det systemet." + Utforska vidare