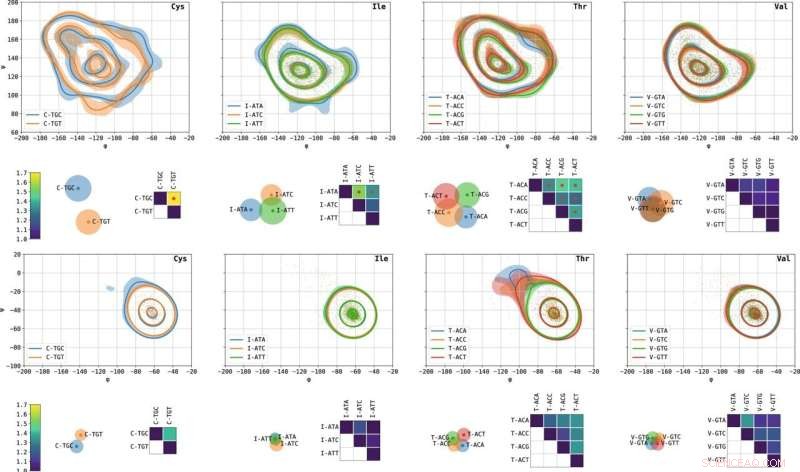
Kodonspecifika Ramachandran-plottar av utvalda aminosyror och avstånd mellan dem. Från vänster till höger visas cystein, isoleucin, treonin och valin. Konturplottar visar nivålinjerna som innehåller 10, 50 och 90 % av sannolikhetsmassan. Skuggade områden representerar 10 %–90 % konfidensintervall beräknade på 1000 slumpmässiga bootstraps. β- (överst) och α- (botten) lägena är avbildade. Matriserna visar L1 avstånd mellan par av kodonspecifika Ramachandran-diagram, normaliserade så att självavståndet är 1. Röda prickar indikerar par med signifikant olika dihedriska vinkelfördelningar baserat på deras p-värde. Spridningsdiagrammen som visualiserar avståndsmatriserna erhölls med en variant av multidimensionell skalning (MDS). Varje punkt representerar ett kodon; parvis euklidiska avstånd mellan punkterna approximerar L1 avståndet mellan motsvarande kodon. Cirklar approximerar osäkerhetsradierna. Ju fler två cirklar överlappar, desto mindre särskiljbara är motsvarande kodonspecifika Ramachandran-diagram. Kredit:Nature Communications (2022). DOI:10.1038/s41467-022-30390-9
En studie som integrerar biologiska idéer och nya datavetenskapliga verktyg har avslöjat nya samband mellan genetisk kodning och proteinstruktur, vilket potentiellt kan förändra vårt sätt att tänka på proteinproduktion i ribosomen - cellens "proteinbindningslinje". Forskningen, ledd av professor Alex Bronstein, Dr Ailie Marx och Ph.D. student Aviv Rosenberg, publicerades i Nature Communications .
Proteiner, de komplexa molekyler som spelar avgörande roller i praktiskt taget alla biologiska mekanismer, produceras av ribosomer i en process som kallas translation. Ribosomen avkodar inkommande "genetiska instruktioner" för att syntetisera kedjor av aminosyror - proteiners byggstenar. När aminosyror sekventiellt binds samman till en lång kedja, vikas de till en unik tredimensionell struktur som ger proteinet dess biologiska egenskaper och funktionalitet. Översättningsfel kan leda till felveckning och följaktligen fysiologiska störningar, både lindriga och allvarliga.
Proteinproduktionsinstruktioner levereras till ribosomen som kodoner, sekvenser av tre "bokstäver" från den genetiska nukleotidkoden, som specificerar identiteten och ordningen för aminosyror som ska läggas till av ribosomen till proteinkedjan. Till exempel signalerar kodonet UUU för tillsats av aminosyran fenylalanin, medan kodonet UAC instruerar för tillsats av tyrosin. På detta sätt kodar kodonsekvensen för den unika sekvensen av aminosyror som är karakteristisk för varje protein. Denna kartläggning av genetiska kodon till aminosyror som används vid översättning är gemensam för alla levande varelser på planeten och anses vara en urtidsmekanism.
Som om allt detta inte var tillräckligt komplicerat är det viktigt att påpeka att det finns 61 kodon som är avkodade till bara 20 aminosyror. Med andra ord, alla utom två aminosyror kodas av flera kodon.
Det är här den nuvarande forskningen kommer in i bilden. Baserat på experiment utförda på 1960- och 1970-talen, säger den accepterade dogmen att proteiner inte bär något "minne" av det specifika kodonet från vilket varje aminosyra översatts så länge som aminosyraidentiteten förblir oförändrad. Dessa tidiga experiment med proteinveckning använde kemiska denatureringsmedel för att veckla ut fullt bildade proteiner och visade sedan att efter avlägsnande av dessa kemikalier kunde proteinkedjan återveckas spontant för att återfå sin ursprungliga struktur och funktion. Dessa experiment antydde att endast aminosyrasekvensen, och inte den specifika kodonsekvensen, bestämmer ett proteins struktur. Med tanke på denna dogm kallas mutationer som ändrar den genetiska kodningen utan att ändra aminosyran allmänt som "tysta" och anses vara oviktiga för proteinstruktur och funktion.
Forskargruppen från Technion har upptäckt ett samband mellan kodonets identitet och den lokala strukturen hos det översatta proteinet, vilket tyder på att detta kanske inte är det allmänna fallet och att proteiner verkligen kan "komma ihåg" de specifika instruktionerna från vilka de syntetiserades. Forskargruppen analyserade tusentals tredimensionella proteinstrukturer med hjälp av dedikerade verktyg de utvecklade, som integrerar avancerade datavetenskapliga metoder, maskininlärning och statistik. På detta sätt jämförde de exakt fördelningarna av vinklar som bildas i dessa strukturer under olika synonyma genetiska koder. Deras resultat visar att det för vissa kodon finns ett signifikant statistiskt beroende mellan kodonets identitet och proteinets lokala struktur, vid positionen för aminosyran som kodonet kodar för.
Forskarna framhåller att fynden fortfarande inte kan belysa riktningen för orsakssambandet, vilket innebär att det ännu inte är möjligt att säga om en förändring i genetisk kodning kan orsaka en förändring i den lokala proteinstrukturen eller om strukturella förändringar kan orsaka olika kodning, till exempel genom evolutionära processer. Denna fråga ligger till grund för en efterföljande forskningsstudie som nu genomförs av gruppen. According to Dr. Marx, a biologist by training and education, "If we find in subsequent research that the codon indeed has a causal effect on protein folding, this is likely to have a huge impact on our understanding of protein folding, as well as on future applications, such as engineering new proteins."
Dr. Marx emphasizes that the discovery presented in the article would not have been possible without Prof. Bronstein's computer and analysis skills. "This research is truly interdisciplinary, because biology alone cannot cope with such vast quantities of data without the help of data science, and computer scientists cannot themselves perform research of this kind, since they lack familiarity with the complex biological processes being probed. Therefore, our research highlights the huge advantage of interdisciplinary research that integrates skills from different fields to create a whole that is greater than the sum of its parts."