På grund av den höga transparensen hos celler är det mycket svårt att observera organellerna i dem. Biologer kan märka specifika organeller för observation genom fluorescensfärgning. Detta är lite analogt med att vara i en miljö utan ljus där alla är helt klädda i svart, vilket gör det svårt att hitta dina vänner. Genom att låta våra vänner hålla i en fluorescerande sticka kan vi enkelt hitta dem.
En intressant fråga är:Om vinkeln på den fluorescerande stickan som hålls av min vän representerar en slags signal, hur kan vi upptäcka sådan vinkelinformation?
Precis som det här pusslet, på grund av cellernas mycket transparenta natur, är det mycket svårt att observera organellerna i dem. Med fluorescerande färgning kan biologer märka specifika organeller för observation. De flesta fluorescerande molekyler uppträder som riktade dipoler under absorption eller emission.
Orienteringen av fluoroforer kan avslöja viktig information om strukturen och dynamiken hos deras associerade organeller. Fluorescenspolarisationsmikroskopi har också utvecklats som ett oumbärligt verktyg för att studera biomolekylers orienteringsegenskaper.
För att övervinna utmaningen med konventionell fluorescenspolarisationsmikroskopi begränsad av optisk diffraktion, har förbättrade superupplösningsfluorescenspolarisationsmikroskopitekniker föreslagits, såsom enkelmolekylorientering-lokaliseringsmikroskopi (SMOLM) och polarisationsmodulering (t.ex. SDOM, SPoD, etc. ).
Men ur bioteknologisk synvinkel, trots den betydande rollen av biologiska filament (t.ex. aktinfilament och mikrotubuli) i cellulära funktioner, finns det en brist på tillvägagångssätt med 3D-orienteringsupplösning och hög temporal-spatial upplösning för att studera dem in vivo.
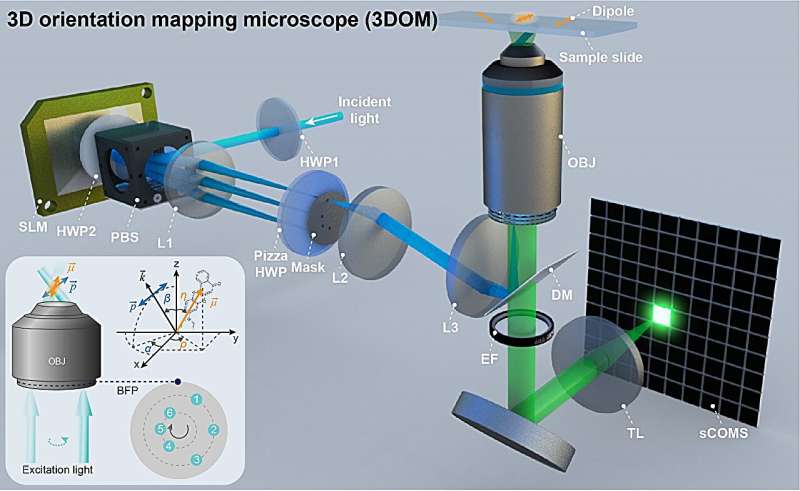
För att ta itu med problemet med dipolorienteringsupplösning har professor Xi Pengs forskargrupp från Peking University utvecklat en 2D-dipolorienteringskartläggningsmetod, SDOM, och optisk inlåsningsdetektering med superupplösningsdipolorientering, OLID-SDOM. I PhotoniX , forskargruppen rapporterar ett superupplöst 3D-orienteringsmappningsmikroskop som kallas 3DOM.
3DOM-metoden bygger på den polariserade strukturerade belysningsmikroskopin som utvecklats av forskargruppen. Genom att vända principen för Youngs dubbelslitsinterferens och kombinera den med principen om reversibla ljusbanor, används olika vinklar på ränderna för att producera positiva och negativa första ordningens strålar i olika riktningar.
Dessutom kan en enda riktning av lutad belysning produceras genom att helt enkelt blockera motsvarande negativa första ordningens ljus. Genom att projicera denna lutning till olika vinklar på z-axeln och rekonstruera bilden med hjälp av FISTA-algoritmen kan högprecisionsupplösning av dipolorienteringen uppnås genom att kombinera polarisationsmodulationskoefficienterna och rekonstruktionen resulterar i reciprokt utrymme.
Sammantaget övervinner den föreslagna 3DOM-metoden effektivt begränsningarna av fluorescenspolarisationsmikroskopi i rumslig upplösning och kartläggning av 3D-orientering med bredfältsavbildning.
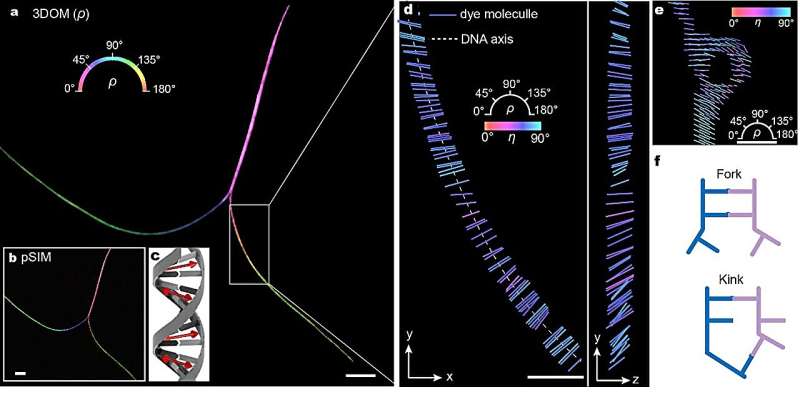
3DOM ger en mer omfattande förståelse av 3D-rumsstrukturen hos fluoroformolekyler. Detta gör det möjligt för oss att inte bara särskilja olika cytoskelettorganisationer (aktinfilament och mikrotubuli) utan också att få värdefulla insikter om filamentbindningskompakthet och ordningen för subcellulära strukturer.
Dessutom har 3DOM betydande potential i DNA-böjning och orientering av membranösa organeller. En av de viktigaste fördelarna med 3DOM är dess lätthet att uppgradera till befintliga widefield-system. Den enkla implementeringen, exakt 3D-dipolorienteringsinformation och överlägsen spatiotemporal upplösning av 3DOM gör den lämplig för ett brett spektrum av applikationer, vilket förbättrar dess tillgänglighet och användbarhet i olika forskningsmiljöer.
Detta kraftfulla verktyg ger forskare möjlighet att reda ut de intrikata komplexiteten i subcellulär struktur, biomekanik och biodynamik, vilket revolutionerar vår förståelse av cellulära processer. Forskarna förutser att 3DOM främjar förståelsen över en mängd biologiska strukturer och interaktioner verksamma på nanoskala.
Mer information: Suyi Zhong et al, Tredimensionell dipolorienteringskartläggning med hög temporal-spatial upplösning med polarisationsmodulering, PhotoniX (2024). DOI:10.1186/s43074-024-00127-6
Tillhandahålls av Peking University