Molecular dynamics (MD)-simuleringar har blivit ett kraftfullt verktyg inom de ständigt växande områdena molekylärbiologi och läkemedelsutveckling. Även om det finns många MD-simuleringstekniker, är parallellkaskadselektion MD (PaCS-MD) särskilt användbar när man studerar veckningen (eller "konformationen") av proteiner eller interaktionerna mellan proteiner och ligander.
Kärnan i denna metod består av att köra flera MD-simuleringar parallellt, och därigenom utforska olika möjliga konformationer samtidigt. Med hjälp av noggrant utformade urvalskriterier kan lovande konformationer detekteras automatiskt i "temporala ögonblicksbilder" och undersökas vidare. Denna strategi påskyndar avsevärt upptäckten av viktiga molekylära interaktioner och dynamiska processer som hjälper forskare att förstå proteinfunktionella rörelser.
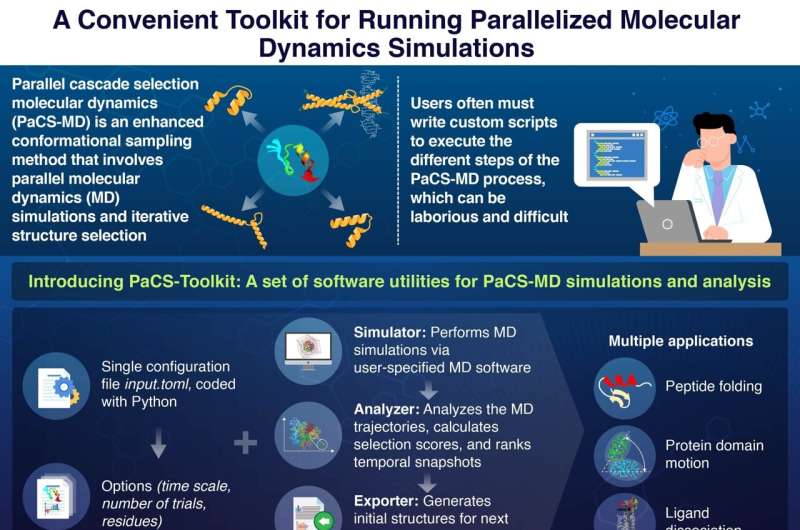
Ett av hindren med PaCS-MD är dock att användare måste skriva anpassade skript för att utföra sina önskade MD-simuleringar. I dessa skript måste de specificera de initiala villkoren och målfunktionerna, välja MD-programvara att använda, implementera en ögonblicksbildsrankningsprocedur och förbereda de initiala strukturerna för nästa simuleringscykel. Denna process, som kan vara ganska komplex och felbenägen, skapar en betydande inträdesbarriär för forskare som är intresserade av att använda PaCS-MD.
Lyckligtvis har ett team av forskare från School of Life Science and Technology vid Tokyo Institute of Technology, Japan, nyligen tagit sig an denna fråga. I deras senaste studie, som publicerades i The Journal of Physical Chemistry B och ledd av professor Akio Kitao utvecklade teamet ett mjukvarupaket som heter PaCS-Toolkit för att göra PaCS-MD mer tillgängligt och bekvämt att använda.
En anmärkningsvärd fördel med PaCS-Toolkit är att hela simuleringsprocessen ställs in via en enda konfigurationsfil. I den här filen specificerar användarna viktiga parametrar för simuleringarna, inklusive typen av PaCS-MD, antalet MD-simuleringar som ska köras parallellt och proteinrester eller atomer att hålla reda på som urvalskriterier för parallella grenar.
PaCS-Toolkit tar den här konfigurationsfilen, tillsammans med vanliga MD-indatafiler, och kör PaCS-MD-simuleringar enligt den specificerade MD-mjukvaran. Värt att notera, eftersom paketet är öppen programvara skriven i Python – ett populärt programmeringsspråk – kan användare hjälpa till att förbättra PaCS-Toolkit och utöka dess funktionalitet.
"Vår verktygslåda bibehåller flexibiliteten så att nya funktioner, bibliotek och MD-mjukvara kan läggas till genom att introducera ansvarsfulla klasser i Python. Användare som kan programmera på detta språk bör kunna modifiera koden för PaCS-Toolkit och implementera nya metoder efter behov, säger Prof. Kitao.
En annan avgörande fördel med PaCS-Toolkit ligger i dess optimering och kompatibilitet med olika datormiljöer, vare sig det är en uppsättning superdatorer med ett meddelandeöverföringsgränssnitt (MPI), servrar utrustade med flera grafikkort (GPU) eller persondatorer som bärbara datorer.
"PaCS-Toolkit innehåller i hög grad parallellisering med MPI, GPU och Pythons multiprocessing-paket, vilket möjliggör optimering av beräkningstiden beroende på tillgängliga beräkningsresurser", förklarar Prof. Kitao.
För att visa upp potentialen i deras verktygslåda genomförde forskarna PaCS-MD-simuleringar skräddarsydda för tre olika applikationer. Dessa inkluderade veckningen av miniproteinet chignolin, rörelsen av proteindomäner i ett SARS-CoV-2-enzym och liganddissociation från en viktig adenosinreceptor.
"Sammantaget indikerar våra resultat att PaCS-Toolkit lätt kan användas för att simulera en mängd olika dynamik för olika typer av molekylära system", avslutar Prof. Kitao.
Denna verktygslåda kan hjälpa till att låsa upp den verkliga potentialen hos PaCS-MD-simuleringar inom olika områden, vilket gör det möjligt för intresserade forskare att belysa komplexa molekylära processer och påskynda läkemedelsupptäckten.
Mer information: Shinji Ikizawa et al, PaCS-Toolkit:Optimized Software Utilities for Parallel Cascade Selection Molecular Dynamics (PaCS-MD) Simulations and Subsequent Analyses, The Journal of Physical Chemistry B (2024). DOI:10.1021/acs.jpcb.4c01271
Journalinformation: Journal of Physical Chemistry B
Tillhandahålls av Tokyo Institute of Technology