Att identifiera nya sätt att rikta in sig på proteiner involverade i mänskliga sjukdomar är en prioritet för många forskare runt om i världen. Det kan dock vara svårt att upptäcka hur man ändrar funktionen hos dessa proteiner, särskilt i levande celler. Nu har forskare från Scripps Research utvecklat en ny metod för att undersöka hur proteiner interagerar med läkemedelsliknande små molekyler i mänskliga celler – vilket avslöjar viktig information om hur man potentiellt kan rikta in dem terapeutiskt.
Strategin, publicerad i Nature Chemical Biology den 2 januari 2024, använder en kombination av kemi och analytiska tekniker för att avslöja de specifika platser där proteiner och små molekyler binder samman. I slutändan kan denna metod leda till utvecklingen av mer riktade och effektiva terapier.
"Vår nya teknik skulle kunna användas för att hitta nya läkemedelsställen på proteiner för alla mänskliga sjukdomar, från cancer till Alzheimers sjukdom", säger Docent Christopher Parker, Ph.D., senior författare till studien vid Institutionen för kemi. "Vi är obegränsade i hur detta skulle kunna användas. Vårt arbete har potential att inleda ett helt nytt sätt att upptäcka läkemedel."
Parker-labbet syftar till att upptäcka hur proteiner fungerar i varje mänsklig celltyp för att utveckla effektiva terapier för ett brett spektrum av mänskliga sjukdomar. I den här studien byggde Parker och hans team på sitt inledande arbete i Scripps Research-professor Benjamin Cravatts labb för att skapa en ny metod för att undersöka hur proteiner interagerar med små molekyler i levande celler.
De utvecklade en analytisk strategi för att bättre förstå hur dessa proteiner samverkar med små molekyler med mycket högre upplösning än någonsin tidigare. För att göra detta använde de kemiska sonder som kallas fotoaffinitetsprober, som är molekyler som kan aktiveras av ljus för att låta sonderna fånga ett bundet protein.
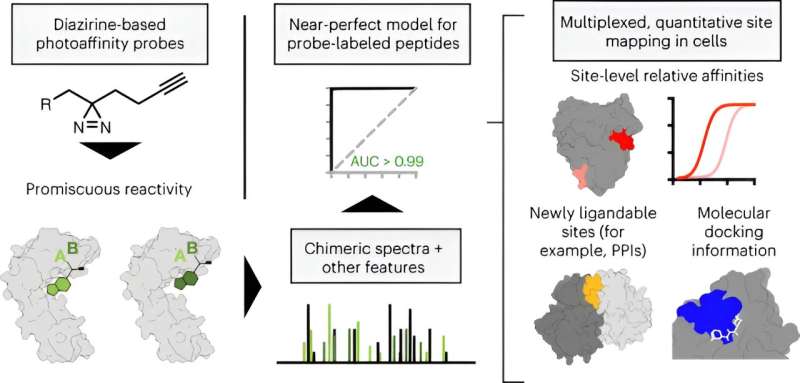
Genom att samla in data från interaktioner mellan proteiner och fotoaffinitetsprober, identifierade Parker-teamet platser på proteiner där små molekyler kunde ansluta och binda. I huvudsak hittade teamet över tusen nya lås (bindningsställen på proteinerna) och motsvarande nycklar (små molekyler), varav den stora majoriteten var nya platser för bindning av små molekyler som inte hade rapporterats tidigare. Dessutom hittade de nya egenskaper hos bindningsställena – som nya former.
"Att identifiera dessa specifika bindningsställen kommer att hjälpa forskare att designa nya molekyler som passar dessa fickor ännu bättre, vilket potentiellt kan leda till mer effektiva terapier", säger Jacob M. Wozniak, medförfattare och tidigare postdoktor i Parker-labbet. Den andra medförsta författaren till artikeln var Weichao Li, Ph.D., en forskarassistent också i Parker-labbet.
Med hjälp av mängden data i denna studie och i samarbete med medförfattaren Stefano Forli, Ph.D., docent vid institutionen för integrativ strukturell och beräkningsbiologi, modellerade författarna sedan hur vissa molekyler kan binda till dessa proteiner. Detta informationsbibliotek skulle kunna användas för att designa terapier som interagerar med proteiner på ett mer riktat sätt.
"Vår nya process avslöjar ytterligare möjligheter för terapeutisk intervention och upptäckt i mänskliga celler", säger Parker. "Närnäst planerar vi att använda denna teknik för att rikta in oss på proteiner som är relevanta för autoimmuna sjukdomar och cancer."
Mer information: Jacob M. Wozniak et al, Enhanced mapping of small-molecule binding sites in cells, Nature Chemical Biology (2024). DOI:10.1038/s41589-023-01514-z
Journalinformation: Kemisk naturbiologi
Tillhandahålls av The Scripps Research Institute