Tabell som visar partiella resultat från montörer som ansökt till den första CAMI-utmaningen, Datauppsättning 1. Klicka här för att se hela tabellen.
De är överallt, men osynlig för blotta ögat. Mikrober är det osynliga, inflytelserika krafter bakom regleringen av viktiga miljöprocesser som kolcykeln, men de flesta av dem är fortfarande okända. I mer än ett decennium, US Department of Energy Joint Genome Institute (DOE JGI), en användaranläggning för DOE Office of Science, har gjort det möjligt för forskare att studera oodlade mikrober som inte kan växa i labbet, använda state-of-the-art tillvägagångssätt som genomisk sekvensering med hög genomströmning av miljögemenskaper ("metagenomics") och utveckling av beräkningsverktyg för att avslöja och karakterisera mikrobiella samhällen från miljön. För att tackla sammansättningen av metagenomer till en uppsättning överlappande DNA-segment som tillsammans representerar en konsensusregion av DNA eller kontiger, sedan binda dessa sammansättningar till genomfack, och slutligen genomföra taxonomisk profilering av genom bins, analytiker runt om i världen har utvecklat en rad olika beräkningsverktyg, men hittills har det saknats konsensus om hur man ska utvärdera deras prestationer.
Publicerad 2 oktober, 2017 in Naturmetoder , ett team inklusive DOE JGI-forskare beskrev resultaten av Critical Assessment of Metagenome Interpretation (CAMI) Challenge, den första någonsin, gemenskapsorganiserad benchmarkingbedömning av beräkningsverktyg för metagenomer. CAMI Challenge leddes av Alexander Sczyrba, chef för Computational Metagenomics-gruppen vid Bielefeld University och tidigare en DOE JGI-postdoktor, och Alice McHardy, chef för Computational Biology of Infection Research Lab vid Helmholtz centrum för infektionsforskning.
"Det är mycket svårt för forskare att ta reda på vilket program som ska användas för en viss datamängd och analys baserat på resultaten från metoduppsatser, ", sade McHardy. "Datauppsättningarna och utvärderingsmåtten som används i utvärderingar varierar kraftigt. En annan fråga är att utvecklare vanligtvis lägger ner mycket tid på att benchmarka det senaste när de bedömer prestandan hos ny programvara på det sättet. CAMI vill förändra dessa saker och involverar samhället i att definiera standarder och bästa praxis för utvärdering och att tillämpa dessa principer i benchmarking utmaningar."
CAMI Challenge ägde rum under tre månader 2015. För att bedöma beräkningsverktygen, arrangörerna utvecklade 3 simulerade metagenomdatauppsättningar med hjälp av mer än 300 utkast av genom av bakteriella och arkeala isolat sekvenserade och sammansatta av DOE JGI, som ingick i projektet Genomic Encyclopedia of Bacteria and Archaeal som nyligen publicerades i Naturens bioteknik . Dessa genom delades med CAMI Challenges-konsortiet innan de släpptes till allmänheten för att underlätta objektiv benchmarking av olika verktyg. Datauppsättningarna inkluderade också ungefär samma antal genom från Max Planck Institute i Köln, Tyskland, tillsammans med cirkulära element och virus. De simulerade datamängderna var en enstaka provdatauppsättning på 15 miljarder baser (Gb), en 40 Gb datamängd med 40 genom och 20 cirkulära element, och en 75 Gb tidsseriedatauppsättning som består av fem prover och inkluderar hundratals genom och cirkulära element.
"JGI har ett starkt intresse av benchmarking av verktyg och teknologier som skulle främja analysen av metagenomer och förbättra kvaliteten på data som vi tillhandahåller användarna. Efter att ha publicerat den allra första studien om användningen av simulerade datauppsättningar för benchmarking av metagenomiska verktyg från JGI, det är fantastiskt att se hur denna metodik har expanderat under åren och nu genom denna studie, utvecklas till en modell för standardiserade samhällsinsatser på området, sa Nikos Kyrpides, DOE JGI Prokaryote Super Programledare.
"JGI är mycket inriktad på inte bara benchmarking av labbprotokoll, men också beräkningsarbetsflöden, " tillade DOE JGI Microbial Genomics chef Tanja Woyke. "Detta gör vårt deltagande i viktiga samhällsinsatser som CAMI så viktigt."
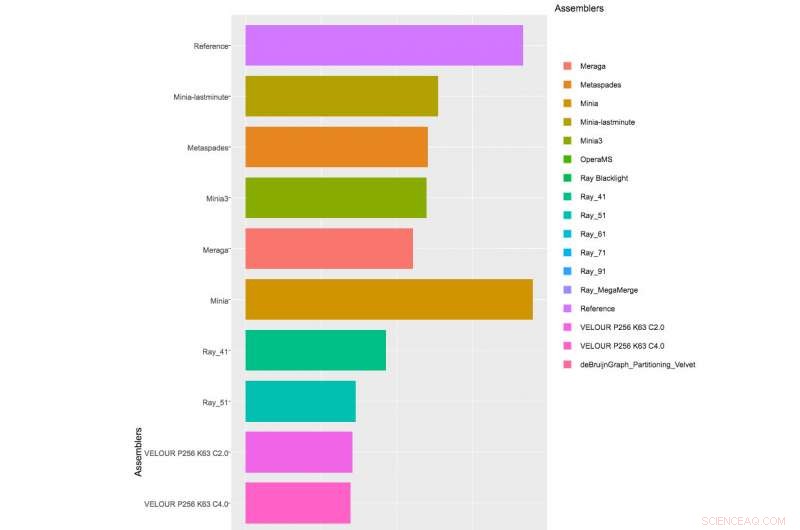
Med mer än 40 lag anmälda till utmaningen, och CAMI-arrangörerna fick 215 bidrag från 25 program runt om i världen, även om endast 17 team var villiga att få sina programvaruimplementeringar publicerade. CAMI-arrangörerna utvärderade beräkningsverktyg i 3 kategorier. Ett halvdussin assemblers och monteringspipelines utvärderades för att sätta samman genomsekvenser genererade från kortlästa sekvenseringsteknologier. I binning-utmaningen, fem genom-binner och 4 taxonomiska binners utvärderades på kriterier inklusive verktygens effektivitet för att återvinna individuella genom. Till sist, 10 taxonomic profilers with various parameter settings were evaluated on how well they could predict the identities and relative abundances of the microbes and circular elements. The benchmarking results are available on https://data.cami-challenge.org/results.
The CAMI organizers are already planning future benchmarking challenges, for example to evaluate and aid method development for long read sequencing technologies. "CAMI is an ongoing initiative, " noted Sczyrba. "We are currently further automating the benchmarking and comparative result visualizations. And we invite everyone interested to join and work with CAMI on providing comprehensive performance overviews of the computational metagenomics toolkit, to inform developers about current challenges in computational metagenomics and applied scientists of the most suitable software for their research questions."