Grafiskt abstrakt. Kredit:Molecular Cell (2022). DOI:10.1016/j.molcel.2022.06.023
Med hjälp av en innovativ ny teknik har forskare vid Duke-NUS Medical School och deras medarbetare identifierat tusentals tidigare okända DNA-sekvenser i det mänskliga genomet som kodar för mikroproteiner och peptider som är potentiellt kritiska för människors hälsa och sjukdomar.
"Mycket av det vi förstår om de kända 2% av genomet som kodar för proteiner kommer från att leta efter långa strängar av proteinkodande nukleotidsekvenser, eller långa öppna läsramar", förklarade beräkningsbiologen Dr. Sonia Chothani, en forskare med Duke-NUS program för kardiovaskulära och metabola störningar (CVMD) och första författare till studien. "Nyligen har dock forskare upptäckt små öppna läsramar (smORF) som också kan översättas från RNA till små peptider, som har roller i DNA-reparation, muskelbildning och genetisk reglering."
Forskare har försökt identifiera smORFs och de små peptider de kodar för, eftersom störningar i dessa smORFs kan orsaka sjukdomar. Men de för närvarande tillgängliga tillvägagångssätten är mycket begränsade.
"Mycket av de nuvarande datamängderna ger inte information som är tillräckligt detaljerad för att identifiera smORFs i RNA," tillade Dr. Chothani. "Majoriteten kommer också från analyser av odödliga mänskliga celler som förökas - ibland i årtionden - för att studera cellfysiologi, funktion och sjukdomar. Men dessa cellinjer är inte alltid korrekta representationer av mänsklig fysiologi."
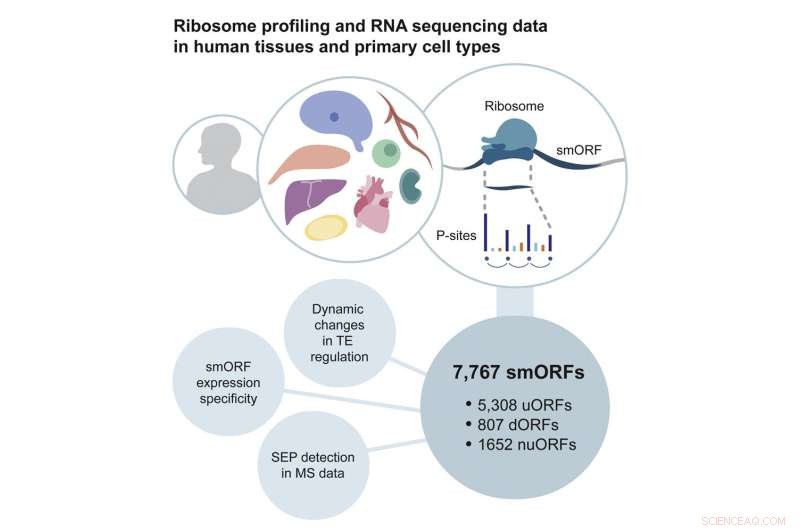
Publicering i Molecular Cell , beskriver Chothani och hennes kollegor i Singapore, Tyskland, Storbritannien och Australien en metod som de utvecklat för att ta itu med dessa problem. De screenade för närvarande tillgängliga ribosomprofileringsdatauppsättningar för korta RNA-strängar med periodiska trebassektioner, som täcker mer än 60% av RNA:s längd. De genomförde sedan sin egen RNA-sekvensering och ribosomprofilering för att generera en kombinerad dataresurs av sex typer av celler och fem typer av vävnader, såsom från hjärtat och hjärnan, från hundratals patienter.
Analyser av dessa data identifierade nästan 8 000 smORFs. Intressant nog var de mycket specifika för de vävnader som de hittades i, vilket innebär att dessa smORFs kan utföra en funktion som är specifik för deras miljö. Teamet identifierade också 603 mikroproteiner kodade av några av dessa smORFs.
"Genomet är full av smORFs", säger biträdande professor Owen Rackham, senior författare till studien från CVMD-programmet. "Vår heltäckande och rumsligt lösta karta över mänskliga smORFs lyfter fram förbisedda funktionella komponenter i genomet, pekar ut nya aktörer inom hälsa och sjukdomar och tillhandahåller en resurs för det vetenskapliga samfundet som en plattform för att påskynda upptäckter."
Professor Patrick Casey, senior vice-dekan för forskning vid Duke-NUS, sa:"Med sjukvårdssystemet som utvecklas för att inte bara behandla sjukdomar utan också förebygga dem, kan identifiering av potentiella nya mål för sjukdomsforskning och läkemedelsutveckling öppna vägar till nya lösningar. Denna forskning av Dr. Chothani och hennes team, publicerad som en resurs för det vetenskapliga samfundet, ger viktiga insikter till området." + Utforska vidare