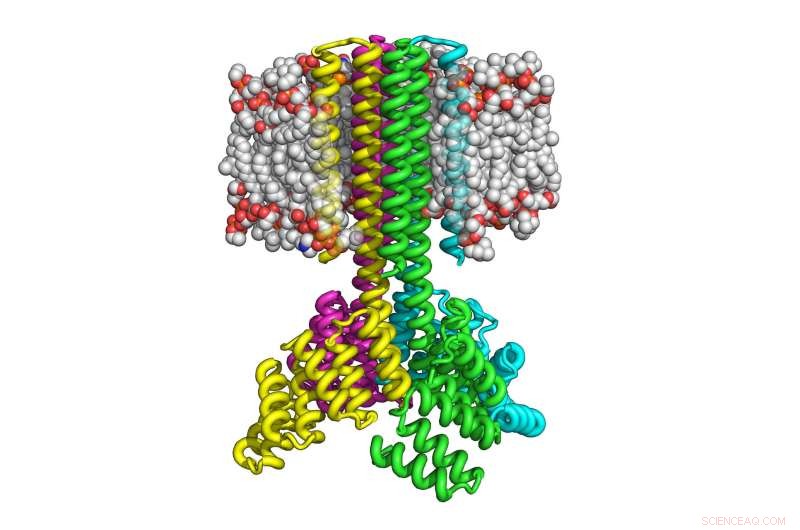
Denna illustration visar hur fyra kopior av datordesignat transmembranprotein som samlats för att bilda en raketformad tetramer med en bred cytoplasmatisk bas som trasar in i åtta transmembranhelixer och som orienteras korrekt i membranet. Kredit:University of Washington Institute for Protein Design
Det är nu möjligt att skapa komplexa, skräddarsydda transmembranproteiner från grunden, forskare rapporterar den här veckan. Förskottet, ledd av molekylära ingenjörer vid University of Washington Institute for Protein Design, kommer att göra det möjligt för forskare att skapa transmembranproteiner som inte finns i naturen för att utföra specifika uppgifter.
I den levande världen, transmembranproteiner finns inbäddade i membranet i alla celler och cellulära organeller. De är viktiga för att de ska fungera normalt. Till exempel, många naturligt förekommande transmembranproteiner fungerar som portar för förflyttning av specifika ämnen över ett biologiskt membran. Vissa transmembranproteiner tar emot eller överför cellsignaler. På grund av sådana roller, många läkemedel är utformade för att rikta transmembranproteiner och ändra deras funktion.
"Våra resultat banar väg för design av multispan-membranproteiner som kan efterlikna proteiner som finns i naturen eller har helt ny struktur, funktion och användningsområden, sa David Baker, en University of Washington School of Medicine professor i biokemi och chef för UW Institute of Protein Design som ledde projektet. Forskningen rapporteras i tidningen 1 mars Vetenskap . Peilong Lu, en senior i Baker -labbet, är tidningens huvudförfattare.
Men att förstå hur transmembranproteiner sätts ihop och hur de fungerar har visat sig vara utmanande. Eftersom de verkar medan de är inbäddade i cellmembranet, transmembranproteiner har visat sig vara svårare att studera än proteiner som verkar i den vattniga lösningen som utgör cellens cytoplasma eller i den extracellulära vätskan.
I den nya studien, Lu och hans kollegor använde ett datorprogram, utvecklat i Baker -labbet och kallat Rosetta, som kan förutsäga strukturen ett protein kommer att vikas till efter att det har syntetiserats. Arkitekturen för ett protein är avgörande eftersom ett proteins struktur avgör dess funktion.
Ett proteins form bildas från komplexa interaktioner mellan aminosyrorna som utgör proteinkedjan och mellan aminosyrorna och den omgivande miljön. I sista hand, proteinet antar den form som bäst balanserar ut alla dessa faktorer så att proteinet uppnår lägsta möjliga energitillstånd.
Rosetta -programmet som används av Lu och hans kollegor kan förutsäga strukturen hos ett protein genom att ta hänsyn till dessa interaktioner och beräkna det lägsta totala energitillståndet. Det är inte ovanligt att programmet skapar tiotusentals modellstrukturer för en aminosyrasekvens och sedan identifierar dem med lägsta energitillstånd. De resulterande modellerna har visat sig exakt representera strukturen som sekvensen sannolikt kommer att anta i naturen.
Det är svårt att bestämma strukturen hos transmembranproteiner eftersom delar av transmembranproteiner måste passera genom membranets inre, som är gjord av oljiga fetter som kallas lipider.
I vattenhaltiga vätskor, aminosyrarester som har polära sidokedjor - komponenter som kan ha en laddning under vissa fysiologiska förhållanden eller som deltar i vätebindning - tenderar att vara lokaliserade på ytan av proteinet där de kan interagera med vatten, som har negativa och positiva sidladdningar till sin molekyl. Som ett resultat, polära rester på proteiner kallas hydrofila, eller "vattenälskande".
Icke-polära rester, å andra sidan, tenderar att hittas packade i proteinkärnan bort från den polära vattenhaltiga vätskan. Sådana rester kallas hydrofoba eller "vattenrädda". Som ett resultat, interaktionen mellan de vattenälskande och vattenrädda resterna av proteinet och de omgivande vattniga vätskorna hjälper till att driva proteinveckningen och stabilisera proteinets slutliga struktur.
I membran, dock, proteinvikning är mer komplicerad eftersom lipidinsidan av membranet är opolärt, det är, det har ingen separation av elektriska laddningar. Detta innebär att för att vara stabilt måste proteinet placera opolärt, vattenfarliga rester på dess yta, och packa dess polare, vattenälskande rester inuti. Då måste den hitta ett sätt att stabilisera sin struktur genom att skapa bindningar mellan de hydrofila resterna i kärnan.
Nyckeln till att lösa problemet, säger Lu, skulle tillämpa en metod som utvecklats av Baker lab för att designa proteiner så att polära, hydrofila rester passar på ett sådant sätt att tillräckligt mycket skulle bilda polära-polära interaktioner som kan binda samman proteinet inifrån.
"Att sätta ihop dessa "begravda vätebindningsnätverk" var som att lägga ett pussel, " sa Baker.
Med detta tillvägagångssätt, Lu och hans kollegor kunde tillverka de designade transmembranproteinerna inuti bakterier och däggdjursceller genom att använda så många som 215 aminosyror. De resulterande proteinerna visade sig vara mycket termiskt stabila och kunna orientera sig korrekt på membranet. Liksom naturligt förekommande transmembranproteiner, proteinerna är multipass, vilket betyder att de passerar membranet flera gånger, och sätts samman till stabila multiproteinkomplex, såsom dimerer, trimerer och tetramerer.
"Vi har visat att det nu är möjligt att exakt utforma komplexa, multipass transmembranproteiner som kan uttryckas i celler. Detta kommer att göra det möjligt för forskare att designa transmembranproteiner med helt nya strukturer och funktioner, sa Lu.