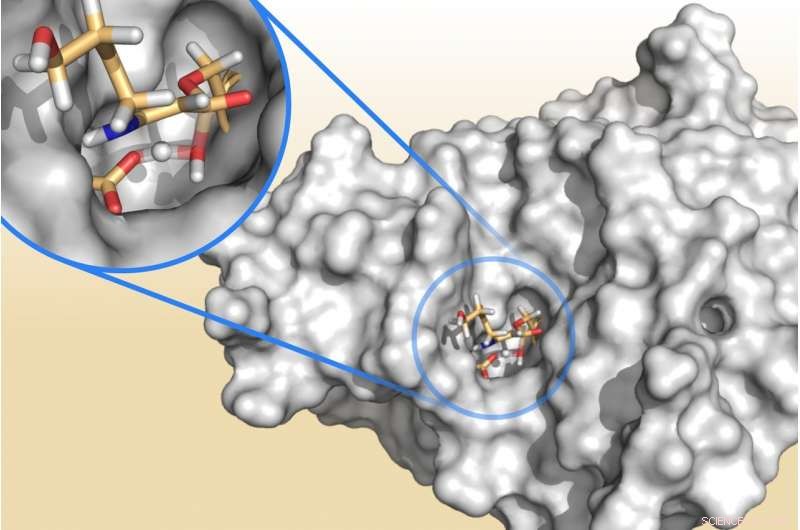
Ett bakteriellt enzym som frisätter fragment av klavulansyra, en "resistensblockerare" utformad för att övervinna antibiotikaresistenta infektioner. Att förstöra klavulansyra gör det möjligt för enzymet att skydda bakterier från effekterna av antibiotika. Kredit:Marc van der Kamp
Genom datorsimuleringar, forskare kan förutsäga om bakterier kan stoppas med populära antibakteriella terapier eller inte – ett genombrott som kommer att hjälpa till att välja ut och utveckla effektiva behandlingar för bakterieinfektioner.
Ökad resistens mot antibiotika är ett av de allvarligaste globala hälsohoten vi står inför. Det finns ett akut behov av att utveckla nya antibiotika som är kostnadseffektiva eftersom det beräknas att år 2050, 10 miljoner liv per år riskerar att drabbas av antibiotikaresistenta infektioner. I ett försök att hjälpa till att hantera denna utmaning, forskare vid University of Bristol har utvecklat datorsimuleringar som kan vara nyckeln till att komma vidare i den pågående "kapprustningen" med bakterier.
Forskare fokuserade på enzymer i bakterier som kan dela strukturen hos antibiotika av penicillintyp, leder till motstånd. För att återställa effektiviteten av dessa antibiotika, "resistensblockerande" molekyler har utvecklats för att blockera aktiviteten hos dessa enzymer. Genom att behandla patienter med rätt kombinationer av antibiotika och resistensblockerare, läkarna kan få övertaget i striden. Tyvärr, bakterier kan göra att många olika enzymer kan förstöra penicilliner, och tillgängliga motståndsblockerare fungerar endast mot några av dessa.
Nya fynd, publiceras i Biokemi , visa att det nu är möjligt att använda datorsimuleringar för att förutsäga om dessa motståndsblockerare kommer att vara effektiva eller inte. Förhoppningen är att denna information kommer att hjälpa forskare att utveckla förbättrade resistensblockerare, som kan återställa verkan av populära antibiotika mot ett bredare spektrum av resistenta bakterier.
Med hjälp av en datorsimuleringsteknik kallad QM/MM (kvantmekanik/molekylär mekanik-simuleringar) kunde Bristols forskargrupp få en insikt på molekylär nivå i hur resistensenzymer reagerar med resistensblockerare.
Forskare fokuserade specifikt på klavulansyra, ett läkemedel som förhindrar förstörelse av vanliga penicillinliknande antibiotika. Klavulansyra används vanligtvis i kombination med antibiotikumet amoxicillin för att behandla öron, bihålor och urinvägsinfektioner (co-amoxiclav). Några sådana bakteriella infektioner, dock, blir mycket svårare att behandla eftersom klavulansyra inte fungerar effektivt mot de enzymer som de producerar.
QM/MM-simuleringarna undersökte hur klavulansyra interagerar med dessa bakteriella enzymer och avslöjade det viktigaste steget för att avgöra om enzymet är effektivt blockerat. Ett enzym som inte kan stoppas frisätter en nedbruten molekyl av klavulansyra och fortsätter att bryta ner antibiotikan som den administreras med, resulterar i antibiotikaresistens. Om nedbrytningen av klavulansyra tar lång tid, då blir enzymet "täppt" och kan inte bryta ner antibiotikan, som sedan kan döda bakterien och rensa infektionen.
Dr Marc van der Kamp, från Bristol University's School of Biochemistry, sa:
"Vi är spännande att se hur våra datorsimuleringar kan användas i framtiden för att testa enzymer från bakterier och förutsäga när en resistensblockerande hämmare kommer att vara effektiv.
Vi hoppas att detta kommer att identifiera hur vi bättre kan blockera sådana bakteriella enzymer, så att antibiotika kan användas effektivt för behandling av läkemedelsresistenta infektioner.
Våra simuleringar kan också vara ett värdefullt verktyg för att hjälpa till att välja vilka kombinationer av läkemedel som är bäst för att behandla ett visst infektionsutbrott, så att vi kan bli bättre rustade i detta "vapenlopp" med bakterier. "