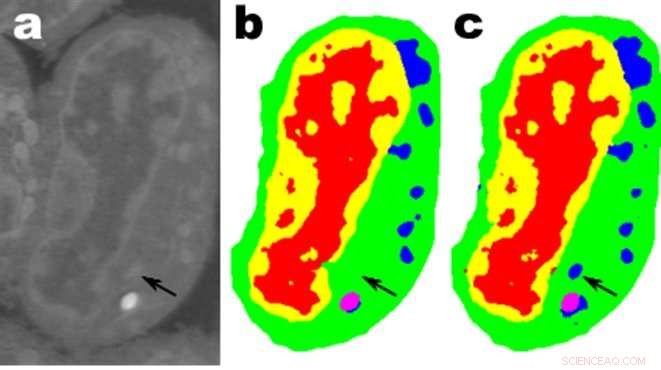
Bilder av en skiva lymfblastoidceller från mus; a. är rådata, b är motsvarande manuella segmentering och c är utdata från ett MS-D-nätverk med 100 lager. Kredit:Data från A. Ekman och C. Larabell, Nationellt centrum för röntgentomografi.
Matematiker vid Department of Energy Lawrence Berkeley National Laboratory (Berkeley Lab) har utvecklat en ny metod för maskininlärning som syftar till experimentell bilddata. Istället för att förlita sig på de tiotals eller hundratusentals bilder som används av typiska maskininlärningsmetoder, detta nya tillvägagångssätt "lär sig" mycket snabbare och kräver mycket färre bilder.
Daniël Pelt och James Sethian från Berkeley Labs Center for Advanced Mathematics for Energy Research Applications (CAMERA) vände det vanliga maskininlärningsperspektivet på huvudet genom att utveckla vad de kallar ett "Mixed-Scale Dense Convolution Neural Network (MS-D)" som kräver mycket färre parametrar än traditionella metoder, konvergerar snabbt, och har förmågan att "lära sig" av ett anmärkningsvärt litet träningsset. Deras tillvägagångssätt används redan för att extrahera biologisk struktur från cellbilder, och är redo att tillhandahålla ett stort nytt beräkningsverktyg för att analysera data inom ett brett spektrum av forskningsområden.
Eftersom experimentanläggningar genererar bilder med högre upplösning vid högre hastigheter, forskare kan kämpa för att hantera och analysera de resulterande data, vilket ofta görs mödosamt för hand. Under 2014, Sethian etablerade CAMERA på Berkeley Lab som en integrerad, tvärvetenskapligt centrum för att utveckla och leverera grundläggande ny matematik som krävs för att dra nytta av experimentella undersökningar vid DOE Office of Sciences användaranläggningar. CAMERA är en del av labbets Computational Research Division.
"I många vetenskapliga tillämpningar, Det krävs enormt manuellt arbete för att kommentera och tagga bilder – det kan ta veckor att producera en handfull noggrant avgränsade bilder, sa Sethian, som också är professor i matematik vid University of California, Berkeley. "Vårt mål var att utveckla en teknik som lär sig av en mycket liten datamängd."
Detaljer om algoritmen publicerades 26 december, 2017 i en tidning i Proceedings of the National Academy of Sciences .
"Genombrottet var resultatet av att man insåg att den vanliga nedskalningen och uppskalningen som fångar funktioner i olika bildskalor kunde ersättas av matematiska faltningar som hanterar flera skalor inom ett enda lager, sa Pelt, som också är medlem i Computational Imaging Group vid Centrum Wiskunde &Informatica, det nationella forskningsinstitutet för matematik och datavetenskap i Nederländerna.
För att göra algoritmen tillgänglig för en bred uppsättning forskare, ett Berkeley-team ledd av Olivia Jain och Simon Mo byggde en webbportal "Segmenting Labeled Image Data Engine (SlideCAM)" som en del av CAMERA-sviten med verktyg för DOE-experimentanläggningar.

Tomografiska bilder av en fiberförstärkt minikomposit, rekonstruerade med 1024 projektioner (a) och 120 projektioner (b). I (c), utgången från ett MS-D-nätverk med bild (b) som ingång visas. Ett litet område indikerat med en röd fyrkant visas förstorat i det nedre högra hörnet av varje bild. Kredit:Daniël Pelt och James Sethian, Berkeley Lab
En lovande tillämpning är att förstå biologiska cellers interna struktur och ett projekt där Pelts och Sethians MS-D-metod endast behövde data från sju celler för att bestämma cellstrukturen.
"I vårt laboratorium, vi arbetar med att förstå hur cellstruktur och morfologi påverkar eller styr cellbeteende. Vi spenderar otaliga timmar på att handsegmentera celler för att extrahera struktur, och identifiera, till exempel, skillnader mellan friska och sjuka celler, sa Carolyn Larabell, Direktör för National Center for X-ray Tomography och professor vid University of California San Francisco School of Medicine. "Det här nya tillvägagångssättet har potential att radikalt förändra vår förmåga att förstå sjukdomar, och är ett nyckelverktyg i vårt nya Chan-Zuckerberg-sponsrade projekt för att upprätta en mänsklig cellatlas, ett globalt samarbete för att kartlägga och karakterisera alla celler i en frisk människokropp."
Få mer vetenskap från mindre data
Bilder finns överallt. Smarta telefoner och sensorer har producerat en skattkammare av bilder, många taggade med relevant information som identifierar innehåll. Genom att använda denna stora databas med korsreferensbilder, konvolutionella neurala nätverk och andra maskininlärningsmetoder har revolutionerat vår förmåga att snabbt identifiera naturliga bilder som ser ut som de som tidigare setts och katalogiserats.
Dessa metoder "lär sig" genom att ställa in en fantastiskt stor uppsättning dolda interna parametrar, guidad av miljontals taggade bilder, och kräver stora mängder superdatortid. Men vad händer om du inte har så många taggade bilder? På många områden, en sådan databas är en ouppnåelig lyx. Biologer spelar in cellbilder och skisserar noggrant gränserna och strukturen för hand:det är inte ovanligt att en person ägnar veckor åt att komma på en enda helt tredimensionell bild. Materialforskare använder tomografisk rekonstruktion för att titta in i stenar och material, och sedan kavla upp ärmarna för att märka olika regioner, identifiera sprickor, frakturer, och tomrum för hand. Kontraster mellan olika men ändå viktiga strukturer är ofta mycket små och "brus" i data kan maskera funktioner och förvirra det bästa av algoritmer (och människor).
Dessa värdefulla handkurerade bilder är långt ifrån tillräckligt för traditionella maskininlärningsmetoder. För att möta denna utmaning, matematiker vid CAMERA attackerade problemet med maskininlärning från mycket begränsade mängder data. Försöker göra "mer med mindre, "Deras mål var att ta reda på hur man bygger en effektiv uppsättning matematiska "operatorer" som avsevärt skulle kunna minska antalet parametrar. Dessa matematiska operatorer kan naturligtvis inkorporera viktiga begränsningar för att hjälpa till med identifiering, till exempel genom att inkludera krav på vetenskapligt rimliga former och mönster.
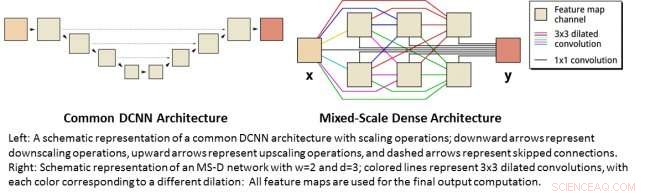
Till vänster:En schematisk representation av en vanlig DCNN-arkitektur med skalningsoperationer; nedåtpilar representerar nedskalningsoperationer, Uppåtpilar representerar uppskalningsoperationer och streckade pilar representerar överhoppade anslutningar. Höger:Schematisk representation av ett MS-D-nätverk med w=2 och d=3; färgade linjer representerar 3x3 vidgade veck, med varje färg som motsvarar en annan utvidgning:Alla funktionskartor används för den slutliga utdataberäkningen. Kredit:Daniël Pelt och James Sethian, Berkeley Lab
Mixed-Scale Dense Convolution Neural Networks
Många tillämpningar av maskininlärning på bildproblem använder deep convolutional neural networks (DCNN), där ingångsbilden och mellanbilderna är hopfällda i ett stort antal på varandra följande lager, så att nätverket kan lära sig mycket olinjära funktioner. För att uppnå exakta resultat för svåra bildbehandlingsproblem, DCNN:er förlitar sig vanligtvis på kombinationer av ytterligare operationer och anslutningar, inklusive, till exempel, nedskalning och uppskalning för att fånga funktioner i olika bildskalor. För att träna djupare och kraftfullare nätverk, Ytterligare lagertyper och anslutningar krävs ofta. Till sist, DCNN:er använder vanligtvis ett stort antal mellanliggande bilder och träningsbara parametrar, ofta mer än 100 miljoner, att uppnå resultat för svåra problem.
Istället, den nya "Mixed-Scale Dense" nätverksarkitekturen undviker många av dessa komplikationer och beräknar dilaterade faltningar som ett substitut till skalningsoperationer för att fånga funktioner i olika rumsliga intervall, använder flera skalor inom ett enda lager, och tätt sammanbinda alla mellanliggande bilder. Den nya algoritmen uppnår exakta resultat med få mellanliggande bilder och parametrar, eliminerar både behovet av att justera hyperparametrar och ytterligare lager eller anslutningar för att möjliggöra träning.
Få högupplöst vetenskap från lågupplösta data
En annan utmaning är att producera högupplösta bilder från lågupplöst input. Som alla som har försökt förstora ett litet foto och upptäckt att det bara blir värre när det blir större, det här låter nästan omöjligt. Men en liten uppsättning träningsbilder som bearbetats med ett Mixed-Scale Dense-nätverk kan ge verklig framgång. Som ett exempel, tänk dig att försöka förkasta tomografiska rekonstruktioner av ett fiberförstärkt minikompositmaterial. I ett experiment som beskrivs i tidningen, bilder rekonstruerades med 1, 024 förvärvade röntgenprojektioner för att få bilder med relativt låga mängder brus. Brusiga bilder av samma objekt erhölls sedan genom att rekonstruera med 128 projektioner. Träningsinmatningar var brusiga bilder, med motsvarande brusfria bilder som används som målutgång under träning. Det tränade nätverket kunde sedan effektivt ta bullriga indata och rekonstruera bilder med högre upplösning.
Nya applikationer
Pelt och Sethian tar sitt förhållningssätt till en mängd nya områden, som snabb realtidsanalys av bilder som kommer ur synkrotronljuskällor och rekonstruktionsproblem vid biologisk rekonstruktion som för celler och hjärnkartering.
"De här nya tillvägagångssätten är verkligen spännande, eftersom de kommer att möjliggöra tillämpningen av maskininlärning på en mycket större mängd bildproblem än vad som är möjligt för närvarande, ", sa Pelt. "Genom att minska mängden nödvändiga träningsbilder och öka storleken på bilder som kan bearbetas, den nya arkitekturen kan användas för att svara på viktiga frågor inom många forskningsområden."